
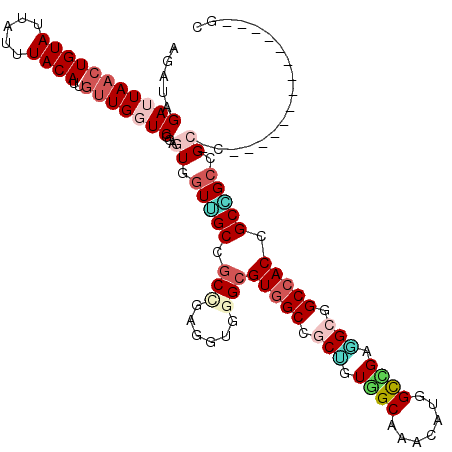
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 27,757,769 – 27,757,911 |
| Length | 142 |
| Max. P | 0.990968 |

| Location | 27,757,769 – 27,757,871 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.73 |
| Mean single sequence MFE | -49.80 |
| Consensus MFE | -29.38 |
| Energy contribution | -30.60 |
| Covariance contribution | 1.22 |
| Combinations/Pair | 1.30 |
| Mean z-score | -3.08 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.24 |
| SVM RNA-class probability | 0.990968 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27757769 102 + 27905053 UGAUAGAUUAACUGUAUUAUUUACAUUAUUGGUCGGAGUGGUGGCCGCCGAAGUGGGCGUGGCCGCGGUAGCGAACAUGGCCGACGAGGCCACCGCCGCCG------------------C .....((((((.((((.....))))...))))))((.(((((((((..((...(((.((((..(((....)))..)))).))).)).)))))))))..)).------------------. ( -44.70) >DroVir_CAF1 85257 114 + 1 ACAUAGAUUAACUGUAUUAUUUACAUUGAUGUUCGGAGUGCUUGCCGCUGAGGUGGGCGUGGCAGCUGUUGCAAACAUAGUGGCAGCGGCCACAGCGGCGGCUCCACUGCAG------GC ...........(((((.((((......))))...((((((((..((.....))..)))(((((.(((((..(.......)..))))).))))).......)))))..)))))------.. ( -45.70) >DroPse_CAF1 81255 108 + 1 UGAUAGAUUAACUGUAUUAUUUACAUUGUUGGUCGGUGUCGUUGCUGCGGACGUGGGCGUGGCGGCCGUGGCAAACAUCGCCGAGGCGGCCACUGCCGCCGCCC------------CAGC .....(((((((((((.....))))..))))))).((.((((....)))))).((((.((((((((.(((((......(((....)))))))).))))))))))------------)).. ( -48.80) >DroGri_CAF1 86149 114 + 1 ACAUAGAUUAACUGUAUUAUUUACAUUGUUGUUCGGAGUGGUCGCCGCCGAGGUGGGCGUGGCGGCUGUCGCAAACAUGGCGGCAGCGGCAACAGCGGCCGCUCCAUUGCAG------GC ...........(((((......(((....)))..(((((((((((.(((......)))((.((.((((((((.......)))))))).)).)).)))))))))))..)))))------.. ( -53.70) >DroWil_CAF1 85844 120 + 1 AGAUAGAUUAACUGUAUUAUUUACAUUGUUGGUCGGUGUUGUUGCUGCAGAUGUGGGCGUGGCUGCUGUGGCAAACAUGGCCGAGGCGGCCACCGCAGCGGCACCACUGCAGGCGGCAGC .....(((((((((((.....))))..)))))))(((((((((((.((........))(((((((((.((((.......)))).))))))))).))))))))))).((((.....)))). ( -55.80) >DroAna_CAF1 75289 102 + 1 AGAUAGAUUAACUGUAUUAUUUACAUUGUUGGUCGGUGUAGUGGCCGCCGAGGUGGGGGUGGCCGCGGUGGCGAACAUGGCCGAGGAGGCCACCGCCGCCG------------------C .....(((((((((((.....))))..)))))))......(((((((((........)))))))))(((((((....(((((.....))))).))))))).------------------. ( -50.10) >consensus AGAUAGAUUAACUGUAUUAUUUACAUUGUUGGUCGGAGUGGUUGCCGCCGAGGUGGGCGUGGCCGCUGUGGCAAACAUGGCCGAGGCGGCCACCGCCGCCGC_C______________GC .....(((((((((((.....))))..)))))))...((.(((((.(((......)))(((((.(((.((((.......)))).))).))))).))))).)).................. (-29.38 = -30.60 + 1.22)


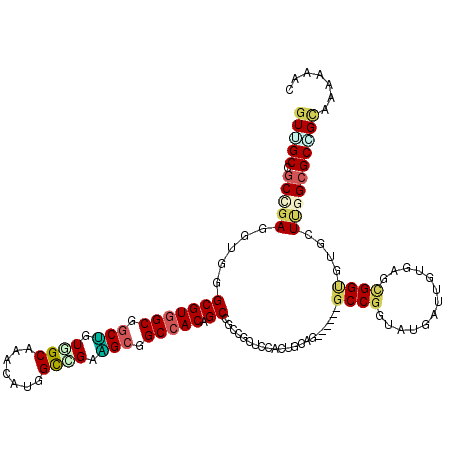
| Location | 27,757,809 – 27,757,911 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 73.51 |
| Mean single sequence MFE | -53.58 |
| Consensus MFE | -28.51 |
| Energy contribution | -28.85 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.53 |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.919256 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27757809 102 + 27905053 GUGGCCGCCGAAGUGGGCGUGGCCGCGGUAGCGAACAUGGCCGACGAGGCCACCGCCGCCG------------------CCGGAAUGAUGGUCAAUGGAGUGCUCGGCGCCGUGAAGAAC ..(((.(((((.(((..(.(((((((((..(((....(((((.....))))).)))..)))------------------).........)))))...)..)))))))))))......... ( -42.80) >DroVir_CAF1 85297 114 + 1 CUUGCCGCUGAGGUGGGCGUGGCAGCUGUUGCAAACAUAGUGGCAGCGGCCACAGCGGCGGCUCCACUGCAG------GCCGGUAUGAUUGUGAGGGGUGUUGUUGGCGCGGCAAAAUAC .(((((((((.(((((..(((((.(((((..(.......)..))))).)))))(((....)))))))).)).------(((..((..(((......)))..))..))))))))))..... ( -50.10) >DroPse_CAF1 81295 108 + 1 GUUGCUGCGGACGUGGGCGUGGCGGCCGUGGCAAACAUCGCCGAGGCGGCCACUGCCGCCGCCC------------CAGCCGGUAUGAUCGUGAGCGGCGUGCUCGGCGCCGCAAAGAAC .....(((((.(.((((.((((((((.(((((......(((....)))))))).))))))))))------------))(((((((((.(((....)))))))).))))))))))...... ( -62.30) >DroGri_CAF1 86189 114 + 1 GUCGCCGCCGAGGUGGGCGUGGCGGCUGUCGCAAACAUGGCGGCAGCGGCAACAGCGGCCGCUCCAUUGCAG------GCCGGUAUGAUUGUGAGCGGUGUGGUUGGCGCUGCGAAAUAC .(((((((((..((((.(((.((.((((((((.......)))))))).))....))).))))..)....((.------((((.(((....)))..)))).))...))))..))))..... ( -53.90) >DroWil_CAF1 85884 120 + 1 GUUGCUGCAGAUGUGGGCGUGGCUGCUGUGGCAAACAUGGCCGAGGCGGCCACCGCAGCGGCACCACUGCAGGCGGCAGCCGGUAUGAUUGUCAGCGGUGUACUGGGAGCAGCAAAGAAU .(((((((...(((.(.((((((((((.((((.......)))).))))))))).((((.(....).)))).).).))).(((((((.((((....)))))))))))..)))))))..... ( -58.00) >DroMoj_CAF1 90009 111 + 1 GUCGCCGCUGAGGUGGGCGUGGCGGCUGUGGCAAACAUGGCGGCAGCAGCCACAGC---GGCUCCACUACAU------GCCGGUAUUAUCGUGAGCGGUGUUGUUGGCGCGGCAAAAUAC ...(((((((.((((((((((((.(((((.((.......)).))))).)))))...---.)).))))).)).------(((..((.(((((....))))).))..))))))))....... ( -54.40) >consensus GUUGCCGCCGAGGUGGGCGUGGCGGCUGUGGCAAACAUGGCCGAAGCGGCCACAGCCGCCGCUCCACUGCAG______GCCGGUAUGAUUGUGAGCGGUGUGCUUGGCGCCGCAAAAAAC (((((.(((((.....(((((((.(((.((((.......)))).))).))))).))......................((((.............))))....))))))))))....... (-28.51 = -28.85 + 0.34)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:54:13 2006