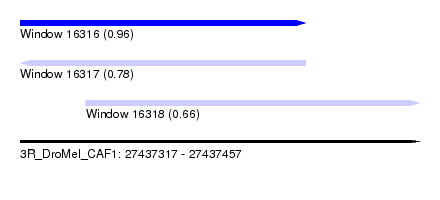
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 27,437,317 – 27,437,457 |
| Length | 140 |
| Max. P | 0.958039 |

| Location | 27,437,317 – 27,437,417 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.38 |
| Mean single sequence MFE | -47.43 |
| Consensus MFE | -34.46 |
| Energy contribution | -36.49 |
| Covariance contribution | 2.03 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.48 |
| SVM RNA-class probability | 0.958039 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
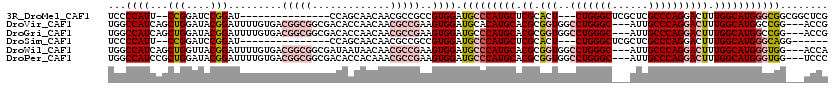
>3R_DroMel_CAF1 27437317 100 + 27905053 UCCCCAUU--CCGGAUCCGGAU---------------CCAGCAACAACGCCGCCGUGGAUGCCCAUGCUCGCACU---CUGGGCUCGCUCGCCCAGGACUUUGGCAUGGGCGGCGGCUCG .......(--(((....)))).---------------...........((((((......(((((((((.....(---((((((......))))))).....)))))))))))))))... ( -47.20) >DroVir_CAF1 18597 114 + 1 UGGCCAUCAGCUGGAUACGGAUUUUGUGACGGCGGCGACACCAACAACGCCGAAGUGGAUGCACAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGCCGG---ACCG .((((....((((..((((.....)))).))))(((.(..((((...(((((..(((.((....)).))).))))).(((((((---...)))))))...))))..).)))))---.)). ( -46.10) >DroGri_CAF1 14704 114 + 1 UGGCCAUCAGCUGGAUACGGAUUUUGUGACGGCGGCGACACCAACAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGCCGG---ACCG .((((....((((..((((.....)))).))))(((.(..((((...(((((..((((....)))).....))))).(((((((---...)))))))...))))..).)))))---.)). ( -46.30) >DroSim_CAF1 18702 94 + 1 UCCCCAUU--CCGGAUCCGGAU---------------CCAGCAACAACGCCGCCGUGGAUGCCCAUGCUCGCACU---CUGGGCUCGCUCGCCCAGGACUUUGGCAUGGGCAGG------ ...(((((--(((....)))).---------------...((......))....)))).((((((((((.....(---((((((......))))))).....))))))))))..------ ( -41.00) >DroWil_CAF1 11535 114 + 1 UGGCCAUCAGCUGGUUACGGAUUUUGUGACGGCGGCGAUAAUAACAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGGUGG---ACCA .(((((((.(((.((((((.....))))))..(((((..........))))).))).)))((((((((.((.(((..(((((((---...)))))))))).))))))))))))---.)). ( -50.20) >DroPer_CAF1 15501 114 + 1 UGGCCAUCCGCUGGAUACGGAUUUUGUGACGGCGGCGACACCACAAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGGUGG---UCCC .(((((((((((.....(((..(((((...((.(....).)))))))..))).)))))))((((((((.((.(((..(((((((---...)))))))))).))))))))))))---)).. ( -53.80) >consensus UGGCCAUCAGCUGGAUACGGAUUUUGUGACGGCGGCGACACCAACAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC___AUUGCCCAGGACUUUGGCAUGGGCGG___ACCG ...((((...(((....))).........(((((.............)))))..)))).(((((((((.((.(((..(((((((......)))))))))).)))))))))))........ (-34.46 = -36.49 + 2.03)
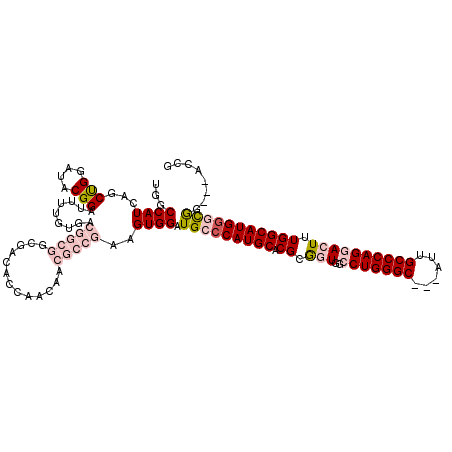

| Location | 27,437,317 – 27,437,417 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.38 |
| Mean single sequence MFE | -45.58 |
| Consensus MFE | -25.35 |
| Energy contribution | -27.97 |
| Covariance contribution | 2.61 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.779872 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27437317 100 - 27905053 CGAGCCGCCGCCCAUGCCAAAGUCCUGGGCGAGCGAGCCCAG---AGUGCGAGCAUGGGCAUCCACGGCGGCGUUGUUGCUGG---------------AUCCGGAUCCGG--AAUGGGGA ...((((((((((((((....(..((((((......))))))---..)....))))))))......)))))).(..((.((((---------------((....))))))--))..)... ( -50.10) >DroVir_CAF1 18597 114 - 1 CGGU---CCGGCCAUGCCAAAGUCCUGGGCAAU---GCCCAGGCCACCGCGUGCAUGUGCAUCCACUUCGGCGUUGUUGGUGUCGCCGCCGUCACAAAAUCCGUAUCCAGCUGAUGGCCA .(((---(((((.(((((.(((((((((((...---))))))).....(.((((....)))).))))).))))).)))))....)))(((((((.................))))))).. ( -43.73) >DroGri_CAF1 14704 114 - 1 CGGU---CCGGCCAUGCCAAAGUCCUGGGCAAU---GCCCAGGCCACCGCGUGCAUGGGCAUCCACUUCGGCGUUGUUGGUGUCGCCGCCGUCACAAAAUCCGUAUCCAGCUGAUGGCCA .(((---(((((.(((((.(((((((((((...---))))))).....(.((((....)))).))))).))))).)))))....)))(((((((.................))))))).. ( -43.33) >DroSim_CAF1 18702 94 - 1 ------CCUGCCCAUGCCAAAGUCCUGGGCGAGCGAGCCCAG---AGUGCGAGCAUGGGCAUCCACGGCGGCGUUGUUGCUGG---------------AUCCGGAUCCGG--AAUGGGGA ------(((((((((((....(..((((((......))))))---..)....))))))))).((((((((((...))))))).---------------.((((....)))--).))))). ( -47.60) >DroWil_CAF1 11535 114 - 1 UGGU---CCACCCAUGCCAAAGUCCUGGGCAAU---GCCCAGGCCACCGCGUGCAUGGGCAUCCACUUCGGCGUUGUUAUUAUCGCCGCCGUCACAAAAUCCGUAACCAGCUGAUGGCCA ..((---(((..(((((....(.(((((((...---))))))))....)))))..))))).........((((.((....)).))))(((((((.................))))))).. ( -41.73) >DroPer_CAF1 15501 114 - 1 GGGA---CCACCCAUGCCAAAGUCCUGGGCAAU---GCCCAGGCCACCGCGUGCAUGGGCAUCCACUUCGGCGUUUGUGGUGUCGCCGCCGUCACAAAAUCCGUAUCCAGCGGAUGGCCA .(((---...((((((((...(((((((((...---)))))))..))...).)))))))..))).....(((.((((((..(.......)..))))))((((((.....)))))).))). ( -47.00) >consensus CGGU___CCGCCCAUGCCAAAGUCCUGGGCAAU___GCCCAGGCCACCGCGUGCAUGGGCAUCCACUUCGGCGUUGUUGGUGUCGCCGCCGUCACAAAAUCCGUAUCCAGCUGAUGGCCA .........((((((((....(((((((((......))))))).....))..))))))))........(((((..........)))))..................(((.....)))... (-25.35 = -27.97 + 2.61)

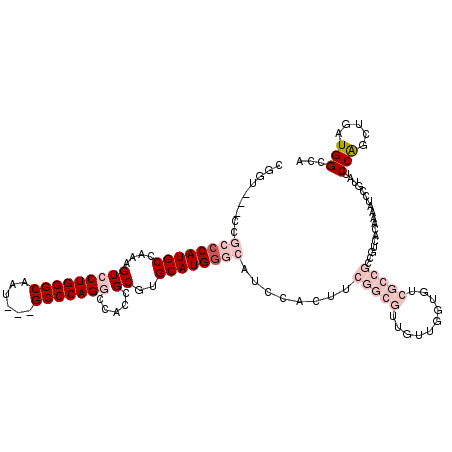
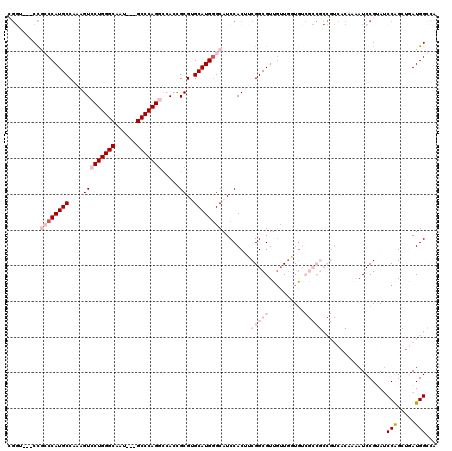
| Location | 27,437,340 – 27,437,457 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.30 |
| Mean single sequence MFE | -53.49 |
| Consensus MFE | -39.13 |
| Energy contribution | -39.97 |
| Covariance contribution | 0.83 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.26 |
| SVM RNA-class probability | 0.657318 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27437340 117 + 27905053 GCAACAACGCCGCCGUGGAUGCCCAUGCUCGCACU---CUGGGCUCGCUCGCCCAGGACUUUGGCAUGGGCGGCGGCUCGGGGGCGUGUCCGCCCGCCCACAUGUACGGCGCCGUAGCCC ((.((..(((((((((...((((((((((.....(---((((((......))))))).....)))))))))))))))..(((((((....))))).)).........))))..)).)).. ( -60.90) >DroVir_CAF1 18637 114 + 1 CCAACAACGCCGAAGUGGAUGCACAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGCCGG---ACCGGCCGCUGGUCCGCCGGCACAUAUGUACGGCGCCAUAGCGC ....((((((((..(((.((....)).))).))))).(((((((---...)))))))...)))(((((((.((---(((((...)))))))((((..((....)).))))))))).)).. ( -54.10) >DroPse_CAF1 16109 114 + 1 CCACAAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGGUGG---UCCCGGUGCCGGUCCGCCCGCCCACAUGUACGGCGCCGUAGCCC ((((..........))))..((((((((.((.(((..(((((((---...)))))))))).))))))))))((---(..((((((((...................))))))))..))). ( -54.01) >DroGri_CAF1 14744 114 + 1 CCAACAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGCCGG---ACCGGCCACUGGUCCCCCAGCCCAUAUGUACGGCGCCAUAGCGC ....((((((((..((((....)))).....))))).(((((((---...)))))))...)))(((((((.((---(((((...)))))))....(((.........)))))))).)).. ( -48.40) >DroWil_CAF1 11575 105 + 1 AUAACAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGGUGG---ACCAGGGGCAGGUCCGCCAUCUCAUAUGUAUGGG---------C .......(((....)))...(((((((((.((.(.(((((((((---...))))))).)).).))...(((((---(((.......))))))))........))))))))---------) ( -49.50) >DroPer_CAF1 15541 114 + 1 CCACAAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC---AUUGCCCAGGACUUUGGCAUGGGUGG---UCCCGGUGCCGGUCCGCCCGCCCACAUGUACGGCGCCGUAGCCC ((((..........))))..((((((((.((.(((..(((((((---...)))))))))).))))))))))((---(..((((((((...................))))))))..))). ( -54.01) >consensus CCAACAACGCCGAAGUGGAUGCCCAUGCACGCGGUGGCCUGGGC___AUUGCCCAGGACUUUGGCAUGGGCGG___ACCGGGCGCCGGUCCGCCCGCCCACAUGUACGGCGCCGUAGCCC .......(((((..((((((((((((((.((.(((..(((((((......)))))))))).))))))))))((....))........))))))..((......)).)))))......... (-39.13 = -39.97 + 0.83)

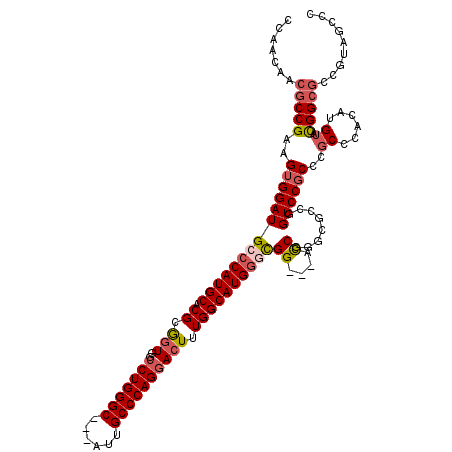
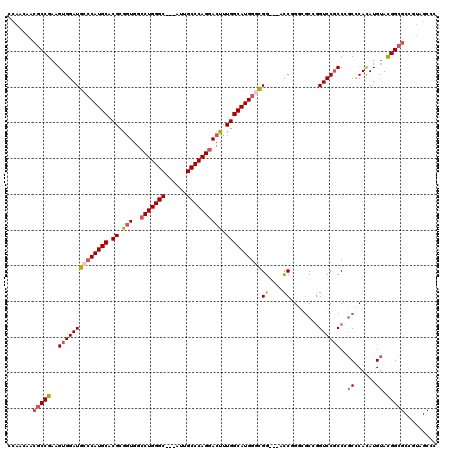
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:52:22 2006