
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 27,426,139 – 27,426,299 |
| Length | 160 |
| Max. P | 0.999717 |

| Location | 27,426,139 – 27,426,259 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.89 |
| Mean single sequence MFE | -51.62 |
| Consensus MFE | -48.69 |
| Energy contribution | -48.30 |
| Covariance contribution | -0.39 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.33 |
| SVM RNA-class probability | 0.999018 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27426139 120 + 27905053 GGGGAUGCCUUCCUCAAUGGUGGGCAGGUCGGAGGAGAAGCGGUUGAUCGGGCGCAAGACGCGCAGCGUGCGACUGGACAGCUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUGUUCUG .(((((((((.((.....)).)))).((((((..(.....)..))))))((((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)))))).))))). ( -51.10) >DroVir_CAF1 1955 120 + 1 CGAUAUACCCUCCUCGAUGGUGGGCAAAUCGGAUGAGAAGCGAUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAUAAUUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUG ((((...(((.((.....)).)))...))))....((((.(((....)))(((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)))))...)))). ( -49.60) >DroGri_CAF1 34 120 + 1 CGAUAUGCCCUCCUCGAUGGUGGGCAGAUCGGAUGAGAAGCGAUUGAUCGGGCGCAGAACGCGCAGCGUGCGACUGGAUAACUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUG ((((.(((((.((.....)).))))).))))....((((.(((....)))(((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)))))...)))). ( -55.00) >DroWil_CAF1 34 120 + 1 CGAGAUGCCUUCCUCAAUGGUGGGCAGAUCCGAGGAGAAACGAUUGAUAGGGCGCAACACACGCAGCGUGCGACUGGAUAACUUCCAGACAAGCCGUUGCUUAUAGGGCGCCUUAUUCUG .((..(((((.((.....)).)))))..))...............((((((((((.......((((((.((..(((((.....)))))....)))))))).......))))))))))... ( -47.04) >DroMoj_CAF1 1663 120 + 1 CGAUAUGCCCUCCUCAAUGGUGGGCAAAUCGGAUGAGAAACGAUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAUAAUUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUG ((((.(((((.((.....)).))))).))))....((((.(((....)))(((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)))))...)))). ( -56.40) >DroAna_CAF1 916 120 + 1 GGAGAUGCCUUCCUCAAUGGUGGGCAGGUCGGAUGAGAAGCGGUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAGAGCUUCCAGACUAGCCGCUGCUUGUAGGGCGCCUUGUUCUG ((((.(((((.((.....)).)))))((((((.((.....)).))))))((((((...(((.((((((.((..((((((...))))))....)))))))).)))...))))))..)))). ( -50.60) >consensus CGAGAUGCCCUCCUCAAUGGUGGGCAGAUCGGAUGAGAAGCGAUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAUAACUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUG .((..(((((.((.....)).)))))..)).....((((.(((....)))(((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)))))...)))). (-48.69 = -48.30 + -0.39)

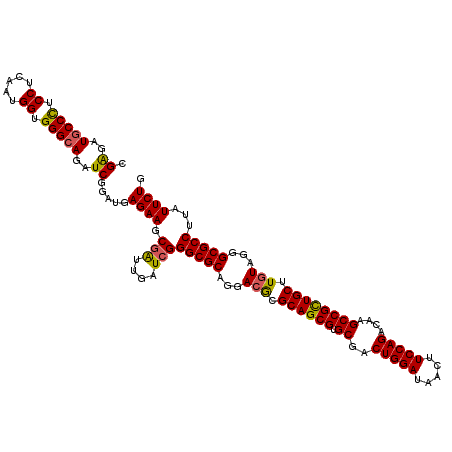

| Location | 27,426,139 – 27,426,259 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.89 |
| Mean single sequence MFE | -42.98 |
| Consensus MFE | -37.38 |
| Energy contribution | -37.43 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.880075 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27426139 120 - 27905053 CAGAACAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAGCUGUCCAGUCGCACGCUGCGCGUCUUGCGCCCGAUCAACCGCUUCUCCUCCGACCUGCCCACCAUUGAGGAAGGCAUCCCC ........(((((...((..((((((((..(.(((((.....))))).))).))))))..))...))))).........((((.(((((................)))))))))...... ( -40.39) >DroVir_CAF1 1955 120 - 1 CAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAAUUAUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAAUCGCUUCUCAUCCGAUUUGCCCACCAUCGAGGAGGGUAUAUCG .((((...(((((...((..((((((((..(.(((((.....))))).))).))))))..))...)))))(((....))).))))....((((.(((((.((.....)).))))).)))) ( -45.90) >DroGri_CAF1 34 120 - 1 CAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAGUUAUCCAGUCGCACGCUGCGCGUUCUGCGCCCGAUCAAUCGCUUCUCAUCCGAUCUGCCCACCAUCGAGGAGGGCAUAUCG .((((...(((((...((..((((((((..(.(((((.....))))).))).))))))..))...)))))(((....))).))))....((((.(((((.((.....)).))))).)))) ( -48.40) >DroWil_CAF1 34 120 - 1 CAGAAUAAGGCGCCCUAUAAGCAACGGCUUGUCUGGAAGUUAUCCAGUCGCACGCUGCGUGUGUUGCGCCCUAUCAAUCGUUUCUCCUCGGAUCUGCCCACCAUUGAGGAAGGCAUCUCG ..((.((.(((((..((((.(((.((((..(.(((((.....))))).))).)).))).))))..))))).))))...............((..((((..((.....))..))))..)). ( -35.60) >DroMoj_CAF1 1663 120 - 1 CAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAAUUAUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAAUCGUUUCUCAUCCGAUUUGCCCACCAUUGAGGAGGGCAUAUCG .((((...(((((...((..((((((((..(.(((((.....))))).))).))))))..))...)))))(((....))).))))....((((.(((((.((.....)).))))).)))) ( -48.20) >DroAna_CAF1 916 120 - 1 CAGAACAAGGCGCCCUACAAGCAGCGGCUAGUCUGGAAGCUCUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAACCGCUUCUCAUCCGACCUGCCCACCAUUGAGGAAGGCAUCUCC ........(((((...((..((((((((..(.(((((.....))))).))).))))))..))...)))))....................((..((((..((.....))..))))..)). ( -39.40) >consensus CAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAGUUAUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAAUCGCUUCUCAUCCGAUCUGCCCACCAUUGAGGAAGGCAUAUCG ........(((((...((..((((((((..(.(((((.....))))).))).))))))..))...)))))....................((..((((..((.....))..))))..)). (-37.38 = -37.43 + 0.06)



| Location | 27,426,179 – 27,426,299 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.67 |
| Mean single sequence MFE | -50.77 |
| Consensus MFE | -47.24 |
| Energy contribution | -46.30 |
| Covariance contribution | -0.94 |
| Combinations/Pair | 1.25 |
| Mean z-score | -3.14 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.94 |
| SVM RNA-class probability | 0.999717 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27426179 120 + 27905053 CGGUUGAUCGGGCGCAAGACGCGCAGCGUGCGACUGGACAGCUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUGUUCUGCCCGUUGUAAUGGGACAGAAUACCCGAAACGAUGCUGAAG ((((..(((((((((...(((.((((((.((..(((((.....)))))....)))))))).)))...))))).((((((((((((....))))).))))))).......))))))))... ( -54.80) >DroVir_CAF1 1995 120 + 1 CGAUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAUAAUUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUGUCCAUUGUAGUGGGACAGAAUGCCCGAGACAAUGCUAAAA ..((((.((((((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)).....(((((((((.........)))))))))))))))..))))....... ( -53.90) >DroGri_CAF1 74 120 + 1 CGAUUGAUCGGGCGCAGAACGCGCAGCGUGCGACUGGAUAACUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUGGCCAUUGUAAUGGGACAGAAUGCCCGACACAAUGCUAAAA ..((((.((((((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)).....((((((.((((....))))..))))))))))))..))))....... ( -49.20) >DroWil_CAF1 74 120 + 1 CGAUUGAUAGGGCGCAACACACGCAGCGUGCGACUGGAUAACUUCCAGACAAGCCGUUGCUUAUAGGGCGCCUUAUUCUGGCCAUUAUAAUGGGAUAGAAUGCCCGAGACAAUGCUGAAA .....((((((((((.......((((((.((..(((((.....)))))....)))))))).......))))))))))..(((.(((.(..((((........))))..).)))))).... ( -40.84) >DroMoj_CAF1 1703 120 + 1 CGAUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAUAAUUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUGUCCAUUGUAAUGAGACAGAAUGCCCGAGACAAUGCUAAAG ..((((.((((((((...(((.((((((.((..(((((.....)))))....)))))))).)))...)).....(((((((((((....))).))))))))))))))..))))....... ( -53.00) >DroAna_CAF1 956 120 + 1 CGGUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAGAGCUUCCAGACUAGCCGCUGCUUGUAGGGCGCCUUGUUCUGCCCGUUGUAGUGGGACAGAAUGCCCGACACGAUGCUGAAG ((((((.((((((((...(((.((((((.((..((((((...))))))....)))))))).)))...)).....((((((((((......)))).))))))))))))))....))))... ( -52.90) >consensus CGAUUGAUCGGGCGCAGGACGCGCAGCGUGCGACUGGAUAACUUCCAGACAAGCCGCUGCUUGUAGGGCGCCUUAUUCUGCCCAUUGUAAUGGGACAGAAUGCCCGAGACAAUGCUAAAA ..((((.((((((((...(((.((((((.((..(((((.....)))))....)))))))).)))...))))).((((((((((((....))))).)))))))..)))..))))....... (-47.24 = -46.30 + -0.94)

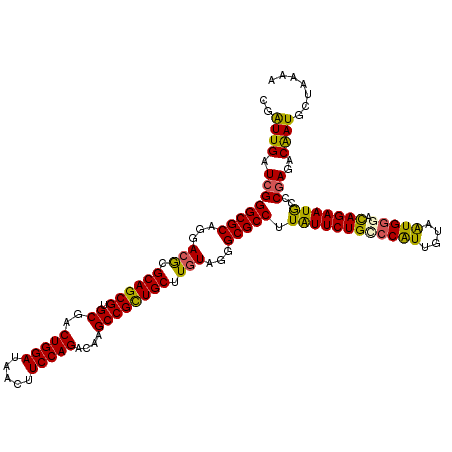

| Location | 27,426,179 – 27,426,299 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.67 |
| Mean single sequence MFE | -47.49 |
| Consensus MFE | -41.90 |
| Energy contribution | -42.59 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.17 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.93 |
| SVM RNA-class probability | 0.997772 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27426179 120 - 27905053 CUUCAGCAUCGUUUCGGGUAUUCUGUCCCAUUACAACGGGCAGAACAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAGCUGUCCAGUCGCACGCUGCGCGUCUUGCGCCCGAUCAACCG ..........(((((((((.(((((.(((........))))))))((((((((.......((((((((..(.(((((.....))))).))).)))))))))))))).))))))..))).. ( -50.11) >DroVir_CAF1 1995 120 - 1 UUUUAGCAUUGUCUCGGGCAUUCUGUCCCACUACAAUGGACAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAAUUAUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAAUCG .....(.((((..(((((((((((((((.........))))))))).((((((.......((((((((..(.(((((.....))))).))).))))))))).)))..)))))).))))). ( -51.21) >DroGri_CAF1 74 120 - 1 UUUUAGCAUUGUGUCGGGCAUUCUGUCCCAUUACAAUGGCCAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAGUUAUCCAGUCGCACGCUGCGCGUUCUGCGCCCGAUCAAUCG .....(.((((.(((((((((((((..((((....)))).)))))).....((...((..((((((((..(.(((((.....))))).))).))))))..))...)))))))))))))). ( -48.10) >DroWil_CAF1 74 120 - 1 UUUCAGCAUUGUCUCGGGCAUUCUAUCCCAUUAUAAUGGCCAGAAUAAGGCGCCCUAUAAGCAACGGCUUGUCUGGAAGUUAUCCAGUCGCACGCUGCGUGUGUUGCGCCCUAUCAAUCG .....(.(((((((.((.((((.((......)).)))).)))))(((.(((((..((((.(((.((((..(.(((((.....))))).))).)).))).))))..))))).)))))))). ( -35.20) >DroMoj_CAF1 1703 120 - 1 CUUUAGCAUUGUCUCGGGCAUUCUGUCUCAUUACAAUGGACAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAAUUAUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAAUCG .....(.((((..((((((((((((((.(((....))))))))))).((((((.......((((((((..(.(((((.....))))).))).))))))))).)))..)))))).))))). ( -50.91) >DroAna_CAF1 956 120 - 1 CUUCAGCAUCGUGUCGGGCAUUCUGUCCCACUACAACGGGCAGAACAAGGCGCCCUACAAGCAGCGGCUAGUCUGGAAGCUCUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAACCG ............(((((((.(((((.(((........))))))))((.(((((.......((((((((..(.(((((.....))))).))).))))))))))).)).)))))))...... ( -49.41) >consensus CUUCAGCAUUGUCUCGGGCAUUCUGUCCCAUUACAAUGGACAGAAUAAGGCGCCCUACAAGCAGCGGCUUGUCUGGAAGUUAUCCAGUCGCACGCUGCGCGUCCUGCGCCCGAUCAAUCG .............(((((((((((((((.........)))))))))..(((((.......((((((((..(.(((((.....))))).))).)))))))))))....))))))....... (-41.90 = -42.59 + 0.70)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:52:14 2006