
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 27,408,690 – 27,408,810 |
| Length | 120 |
| Max. P | 0.913136 |

| Location | 27,408,690 – 27,408,810 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.33 |
| Mean single sequence MFE | -40.91 |
| Consensus MFE | -23.76 |
| Energy contribution | -22.52 |
| Covariance contribution | -1.25 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.46 |
| SVM RNA-class probability | 0.743211 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
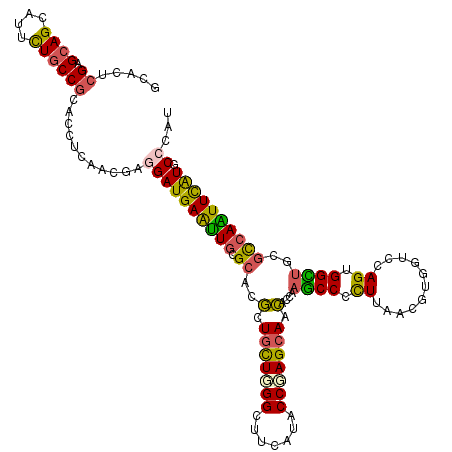
>3R_DroMel_CAF1 27408690 120 + 27905053 GCAUUCGAGCAGCAUUCUGCCGCUGCUAAACGAAGAUGAGUUGCGCACGCUUUUAGGGUUCAUUCCGAGCAACACCAAGCCCUUCAACGUUGUCCAGUGGUUGCGUCAGUUCAUUCCGAU ...((((((((((........))))))...))))(((((..((((((((((....(((.....)))(..((((...............))))..)))))..)))).))..)))))..... ( -32.06) >DroVir_CAF1 12025 120 + 1 UCACUCGAGCAGCAUUUUGCCGCAUCUCAGCGAGGAUGAAUUGCGCACACUGCUGGGUUUUAUACCCAGCAAUACGGAACCACUUAAUGUGGUACAGUGGUUGCGCCAAUUCGUGCCCAU ((.((.((((.((.....)).)).))..)).))(((((((((((((((((((((((((.....)))))))......(.(((((.....))))).)))))..)))).)))))))).))... ( -45.50) >DroPse_CAF1 11467 120 + 1 GCACUCGAGCAGCAUUCUGCCGCACCUCAACGAGGAUGAGCUGCGCACGCUGCUCGGCUUCAUACCGAGCAACACCAAGCCCUUUAACAUUGUCCAGUGGCUGCGCCAGUUCAUUCCGAU .....((.((((....))))))........((.((((((((((((((.(((((((((.......)))))))................((((....)))))))))).)))))))))))).. ( -42.70) >DroGri_CAF1 11721 120 + 1 UCACUCGAGCAGCAUUUUACCGCAACUCAACGACGAUGAAUUACGAACGCUGCUGGGCUUCAUACCCAGCAGCACUGUGCCGCUUAAUGUGGUGCAGUGGCUGCACCAAUUUGUGCCCAU ........(((((.......((.((.(((.......))).)).))...(((((((((.......)))))))))((((..((((.....))))..)))).)))))................ ( -44.80) >DroMoj_CAF1 11491 120 + 1 UCAUUCGAGCAGCGUUUUGCCACAUCUAAGCGAGGAUGAAUUGCGCACGCUGCUGGGCUUUAUACCAAGCACCACAGAGCCGCUUAAUGUGGUGCAGUGGCUCCGUCAAUUCGUGCCCAU .......((((((((..(((..(((((......)))))....))).))))))))((((......(((.((((((((...........))))))))..)))...((......)).)))).. ( -42.70) >DroPer_CAF1 10903 120 + 1 GCACUCGAGCAGCAUUCUGCCGCACCUCAACGAGGAUGAGCUGCGCACGCUGCUCGGCUUCAUACCGAGCAACACGAAGCCCUUUAACAUUGUCCAGUGGCUACGCCAGUUUAUACCGAU ....(((.((.((.....)).)).((((...))))((((((((.((..(.(((((((.......))))))).)..(.((((((............)).)))).)))))))))))..))). ( -37.70) >consensus GCACUCGAGCAGCAUUCUGCCGCACCUCAACGAGGAUGAAUUGCGCACGCUGCUGGGCUUCAUACCGAGCAACACCAAGCCCCUUAACGUGGUCCAGUGGCUGCGCCAAUUCAUGCCCAU .....((.((((....))))))...........((((((((((.((..(.(((((((.......))))))).)....((((.((...........)).))))..)))))))))).))... (-23.76 = -22.52 + -1.25)

| Location | 27,408,690 – 27,408,810 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.33 |
| Mean single sequence MFE | -47.00 |
| Consensus MFE | -27.57 |
| Energy contribution | -27.83 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.09 |
| SVM RNA-class probability | 0.913136 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27408690 120 - 27905053 AUCGGAAUGAACUGACGCAACCACUGGACAACGUUGAAGGGCUUGGUGUUGCUCGGAAUGAACCCUAAAAGCGUGCGCAACUCAUCUUCGUUUAGCAGCGGCAGAAUGCUGCUCGAAUGC .((((((((((((((.(((((.((..(....(......)...)..)))))))))))..............((....))........)))))))(((((((......)))))))))).... ( -31.00) >DroVir_CAF1 12025 120 - 1 AUGGGCACGAAUUGGCGCAACCACUGUACCACAUUAAGUGGUUCCGUAUUGCUGGGUAUAAAACCCAGCAGUGUGCGCAAUUCAUCCUCGCUGAGAUGCGGCAAAAUGCUGCUCGAGUGA ..(((...(((((((((((((((((...........)))))))....((((((((((.....)))))))))))))).)))))).)))(((((..((.(((((.....))))))).))))) ( -50.80) >DroPse_CAF1 11467 120 - 1 AUCGGAAUGAACUGGCGCAGCCACUGGACAAUGUUAAAGGGCUUGGUGUUGCUCGGUAUGAAGCCGAGCAGCGUGCGCAGCUCAUCCUCGUUGAGGUGCGGCAGAAUGCUGCUCGAGUGC .((((.((((.(((((((((((....(((...)))....))))....((((((((((.....)))))))))))))).))).))))((((...)))).(((((.....))))))))).... ( -49.20) >DroGri_CAF1 11721 120 - 1 AUGGGCACAAAUUGGUGCAGCCACUGCACCACAUUAAGCGGCACAGUGCUGCUGGGUAUGAAGCCCAGCAGCGUUCGUAAUUCAUCGUCGUUGAGUUGCGGUAAAAUGCUGCUCGAGUGA .....(((....((((((((...))))))))..(..(((((((....((((((((((.....))))))))))..((((((((((.......)))))))))).....)))))))..)))). ( -57.20) >DroMoj_CAF1 11491 120 - 1 AUGGGCACGAAUUGACGGAGCCACUGCACCACAUUAAGCGGCUCUGUGGUGCUUGGUAUAAAGCCCAGCAGCGUGCGCAAUUCAUCCUCGCUUAGAUGUGGCAAAACGCUGCUCGAAUGA .(((((.((......))..((((..((((((((...........)))))))).)))).....)))))(((((((..((....((((........))))..))...)))))))........ ( -45.60) >DroPer_CAF1 10903 120 - 1 AUCGGUAUAAACUGGCGUAGCCACUGGACAAUGUUAAAGGGCUUCGUGUUGCUCGGUAUGAAGCCGAGCAGCGUGCGCAGCUCAUCCUCGUUGAGGUGCGGCAGAAUGCUGCUCGAGUGC ..((((....))))(((.((((....(((...)))....)))).)))((((((((((.....))))))))))(..((((.((((.......)))).)))(((((....)))))...)..) ( -48.20) >consensus AUCGGCACGAACUGGCGCAGCCACUGGACAACAUUAAAGGGCUCCGUGUUGCUCGGUAUGAAGCCCAGCAGCGUGCGCAACUCAUCCUCGUUGAGAUGCGGCAAAAUGCUGCUCGAGUGA ........((.(((.(((((((.................)))....(((((((((((.....)))))))))))))))))).))...((((.......(((((.....))))).))))... (-27.57 = -27.83 + 0.26)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:51:56 2006