| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 27,217,638 – 27,217,743 |
| Length | 105 |
| Max. P | 0.781023 |

| Location | 27,217,638 – 27,217,743 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 77.28 |
| Mean single sequence MFE | -30.90 |
| Consensus MFE | -17.37 |
| Energy contribution | -17.54 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.40 |
| SVM RNA-class probability | 0.722179 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
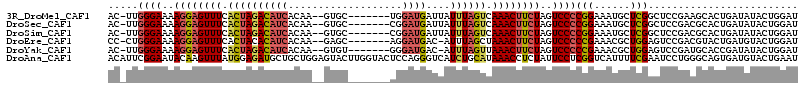
>3R_DroMel_CAF1 27217638 105 + 27905053 AC-UUGGGAAAAGGAGUUUCACUAGACAUCACAA--GUGC-------UGGAUGAUUAUUUAGUCAAACUUCUAGUCCCCGGAAAUGCUCGGCUCCGAAGCACUGAUAUACUGGAU .(-(.((((..((((((((.((((((((((.((.--....-------))))))....)))))).))))))))..)))).))...((((((....)).)))).............. ( -31.10) >DroSec_CAF1 31071 105 + 1 AC-UUGGGAAAAGGAGUUUCACUAGACAUCACAA--GUGC-------CGGAUGAUUAUUUAGUCAAACUUCUAGUCCCCGGAAAUGCUCGGCUCCGACGCACUGAUAUACUGGAU .(-(.((((..((((((((.((((((((((.(..--....-------.)))))....)))))).))))))))..)))).))...((((((....))).))).............. ( -29.70) >DroSim_CAF1 32025 105 + 1 AC-UUGGGAAAAGGAGUUUCACUAGACAUCACAA--GUGC-------CGGAUGAUUAUUUAGUCAAACUUCUAGUCCCCGGAAAUGCUCGGCUCCGACGCACUGAUAUACUGGAU .(-(.((((..((((((((.((((((((((.(..--....-------.)))))....)))))).))))))))..)))).))...((((((....))).))).............. ( -29.70) >DroEre_CAF1 34433 104 + 1 CC-CUGGGAAAAGGAGUUUCACUACACAUCACAA--GAGC-------AGGAUGAC-AUUUAGCUAAACUUCUAGUCCCCCGAAACGCUGGAGUCCGACGUACUGAUGUACUGGAU ..-..((((..((((((((..(((..((((.(..--....-------.)))))..-...)))..))))))))..))))(((......))).(((((..((((....))))))))) ( -25.70) >DroYak_CAF1 32232 104 + 1 AC-UUGGGAAAAGGAGUUUCACUAGACAUCACAA--GUGU-------GGGAUGAC-AUUUAGUUAAACUUCUAGUCCCCCGAAACGCUGGAGUCCGAUGCACCGAUAUACUGGAU ..-..((((..((((((((.((((((((((.((.--...)-------).))))..-.)))))).))))))))..))))(((......))).(((((.((........)).))))) ( -26.70) >DroAna_CAF1 34549 115 + 1 ACAUUCGGAAUACAAGUUUAUGGAGAUGCUGCUGGAGUACUUGGUACUCCAGGGUCAUCUGCAUAAACCUCUAUUCCUCGGUCAUUUUCGAAUCCUGGGCAGUGAUGUACUGAAU ..((((((((((...(((((((.(((((.(.(((((((((...))))))))).).))))).)))))))...))))))...((((((((((.....)))).)))))).....)))) ( -42.50) >consensus AC_UUGGGAAAAGGAGUUUCACUAGACAUCACAA__GUGC_______CGGAUGAUUAUUUAGUCAAACUUCUAGUCCCCGGAAAUGCUCGACUCCGACGCACUGAUAUACUGGAU .....((((..((((((((.((((((((((...................))))....)))))).))))))))..))))(((......)))......................... (-17.37 = -17.54 + 0.17)
| Location | 27,217,638 – 27,217,743 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 77.28 |
| Mean single sequence MFE | -29.67 |
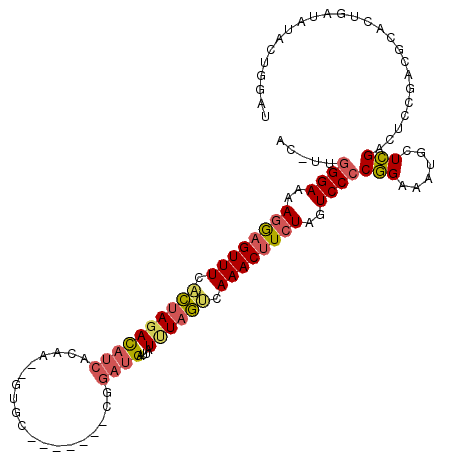
| Consensus MFE | -19.26 |
| Energy contribution | -18.35 |
| Covariance contribution | -0.91 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.781023 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27217638 105 - 27905053 AUCCAGUAUAUCAGUGCUUCGGAGCCGAGCAUUUCCGGGGACUAGAAGUUUGACUAAAUAAUCAUCCA-------GCAC--UUGUGAUGUCUAGUGAAACUCCUUUUCCCAA-GU ............((((((.((....))))))))....((((..((.(((((.((((....(((((...-------....--..)))))...)))).))))).))..))))..-.. ( -27.60) >DroSec_CAF1 31071 105 - 1 AUCCAGUAUAUCAGUGCGUCGGAGCCGAGCAUUUCCGGGGACUAGAAGUUUGACUAAAUAAUCAUCCG-------GCAC--UUGUGAUGUCUAGUGAAACUCCUUUUCCCAA-GU ............(((((.(((....))))))))....((((..((.(((((.((((....(((((...-------....--..)))))...)))).))))).))..))))..-.. ( -27.60) >DroSim_CAF1 32025 105 - 1 AUCCAGUAUAUCAGUGCGUCGGAGCCGAGCAUUUCCGGGGACUAGAAGUUUGACUAAAUAAUCAUCCG-------GCAC--UUGUGAUGUCUAGUGAAACUCCUUUUCCCAA-GU ............(((((.(((....))))))))....((((..((.(((((.((((....(((((...-------....--..)))))...)))).))))).))..))))..-.. ( -27.60) >DroEre_CAF1 34433 104 - 1 AUCCAGUACAUCAGUACGUCGGACUCCAGCGUUUCGGGGGACUAGAAGUUUAGCUAAAU-GUCAUCCU-------GCUC--UUGUGAUGUGUAGUGAAACUCCUUUUCCCAG-GG ..((.((((....)))).((((((......).)))))((((..((.(((((.((((.((-(((((...-------....--..))))))).)))).))))).))..)))).)-). ( -27.40) >DroYak_CAF1 32232 104 - 1 AUCCAGUAUAUCGGUGCAUCGGACUCCAGCGUUUCGGGGGACUAGAAGUUUAACUAAAU-GUCAUCCC-------ACAC--UUGUGAUGUCUAGUGAAACUCCUUUUCCCAA-GU .(((.((((....))))...)))..((........))((((..((.(((((.((((.((-(((((...-------....--..))))))).)))).))))).))..))))..-.. ( -24.20) >DroAna_CAF1 34549 115 - 1 AUUCAGUACAUCACUGCCCAGGAUUCGAAAAUGACCGAGGAAUAGAGGUUUAUGCAGAUGACCCUGGAGUACCAAGUACUCCAGCAGCAUCUCCAUAAACUUGUAUUCCGAAUGU ((((.....(((.((....)))))(((........)))((((((.(((((((((.(((((...(((((((((...)))))))))...))))).))))))))).)))))))))).. ( -43.60) >consensus AUCCAGUAUAUCAGUGCGUCGGAGCCGAGCAUUUCCGGGGACUAGAAGUUUAACUAAAUAAUCAUCCA_______GCAC__UUGUGAUGUCUAGUGAAACUCCUUUUCCCAA_GU .(((.((((....))))...)))..((........))((((..((.(((((.((((....(((((..................)))))...)))).))))).))..))))..... (-19.26 = -18.35 + -0.91)

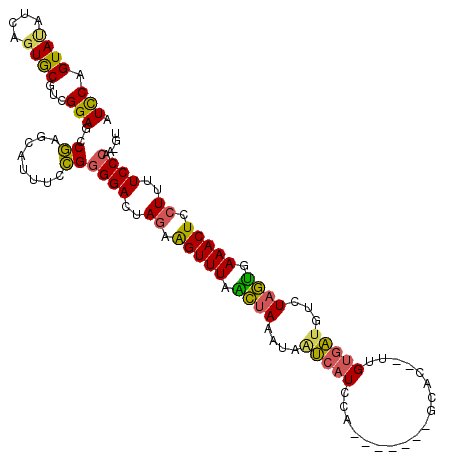
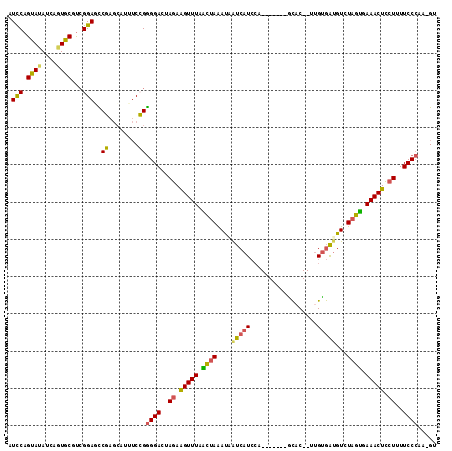
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:49:46 2006