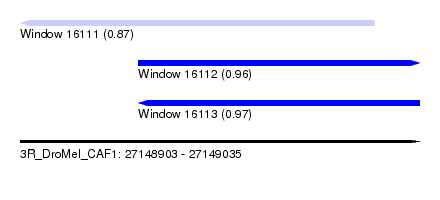
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 27,148,903 – 27,149,035 |
| Length | 132 |
| Max. P | 0.969805 |

| Location | 27,148,903 – 27,149,020 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 79.04 |
| Mean single sequence MFE | -30.34 |
| Consensus MFE | -18.47 |
| Energy contribution | -20.67 |
| Covariance contribution | 2.20 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.865915 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27148903 117 - 27905053 CCGUGGAAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCCGUUUCCGCAAUACCCAUUAUACCCGCCAUACCCCUCUUU ..((((((((((......(((((((.(((((.....(((((..(((...)))..))))))))))..)))))))...))))))))))............................... ( -36.10) >DroSec_CAF1 50514 117 - 1 CCGUGGCAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCCGUUUCCGCCGUACCCAUUAUACCCGCCAUACCCCUCUUU ..(((((.((((.(((..(((((((.(((((.....(((((..(((...)))..))))))))))..)))))))....)))..)))).(((.......)))..))))).......... ( -34.30) >DroSim_CAF1 53328 117 - 1 CCGUGGCAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCCGUUUCCGCCGUACCCAUUAUACCCGCCAUACCCCUCUUU ..(((((.((((.(((..(((((((.(((((.....(((((..(((...)))..))))))))))..)))))))....)))..)))).(((.......)))..))))).......... ( -34.30) >DroEre_CAF1 51283 117 - 1 CCGUGGAAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGCAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCCGUUUCCGCAAUACCCAUUAUGCCCGUCAUACCCCUCUUU ..((((((((((......(((((((.(((((.....(((((.............))))))))))..)))))))...))))))))))............................... ( -34.52) >DroAna_CAF1 55650 94 - 1 CCGUGGAAGCGGCACGCUCGUUGCAUGAUCAUAAAUCUGCAGGUAGACAAUAAAUGCAAUAAUCUC--CCACUCCUCCUGCUCCGGC-UAGCCAUCA-------------------- ....(((.(((((......)))))..............(((((..((...................--....))..))))))))((.-...))....-------------------- ( -18.60) >DroPer_CAF1 122843 103 - 1 CAAUGGAAGCGGCACGCUCGUUGCCUGAUCAUAAAUCUACAGGUAGACAAUAAAUGCAAUGAUCU--GCCGCACCAACGCCACCCCC------------CCAGCACUGCCUCUGCCU ...(((..((((((.(.(((((((...........((((....))))........))))))).))--))))).)))...........------------...(((.......))).. ( -24.21) >consensus CCGUGGAAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCCGUUUCCGCC_UACCCAUUAUACCCGCCAUACCCCUCUUU ..((((.(((((......((((((..((((((.....((((..((.....))..))))))))))...))))))...))))).))))............................... (-18.47 = -20.67 + 2.20)



| Location | 27,148,942 – 27,149,035 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 93 |
| Reading direction | forward |
| Mean pairwise identity | 89.39 |
| Mean single sequence MFE | -31.20 |
| Consensus MFE | -25.96 |
| Energy contribution | -26.83 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.90 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.955278 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27148942 93 + 27905053 GGGGGAGCUGCAGAGAUCACUGCAUUUAUUGUAUACCUGCAGAUUUAUGAUCAUGCAACCAGCGUGCCGCUUCCACGGCACCUACUCCCAAUC ..(((((.((((..((((((((((..(((...)))..))))).....))))).))))...((.((((((......)))))))).))))).... ( -33.70) >DroSec_CAF1 50553 93 + 1 GGGGGAGCUGCAGAGAUCACUGCAUUUAUUGUAUACCUGCAGAUUUAUGAUCAUGCAACCAGCGUGCCGCUGCCACGGCACCUACUCCCAAUC ..(((((.((((..((((((((((..(((...)))..))))).....))))).))))...((.((((((......)))))))).))))).... ( -33.70) >DroSim_CAF1 53367 93 + 1 GGGGGAGCUGCAGAGAUCACUGCAUUUAUUGUAUACCUGCAGAUUUAUGAUCAUGCAACCAGCGUGCCGCUGCCACGGCACCUACUCCCAAUC ..(((((.((((..((((((((((..(((...)))..))))).....))))).))))...((.((((((......)))))))).))))).... ( -33.70) >DroEre_CAF1 51322 93 + 1 GGGGGAGCUGCAGAGAUCACUGCAUUUAUUGUAUGCCUGCAGAUUUAUGAUCAUGCAACCAGCGUGCCGCUUCCACGGCACCUACUCCCAAUC ..(((((.((((..((((((((((..(((...)))..))))).....))))).))))...((.((((((......)))))))).))))).... ( -33.40) >DroYak_CAF1 53501 77 + 1 GGGGGAAUUGCAGAGAUCACUGCAUUUAUUGUGUACCUGCAGAUUUAUGAUCAUGCAACCAG----------------CACCUACUCCCAAUC (((((...(((((......)))))......((((...((((((((...)))).))))....)----------------)))...))))).... ( -22.60) >DroAna_CAF1 55668 91 + 1 GGAGGAGUGG--GAGAUUAUUGCAUUUAUUGUCUACCUGCAGAUUUAUGAUCAUGCAACGAGCGUGCCGCUUCCACGGCACCUACUCCCAAUC .(.(((((((--(.(.(..((((((((((.((((......))))..))))..))))))..).).(((((......)))))))))))))).... ( -30.10) >consensus GGGGGAGCUGCAGAGAUCACUGCAUUUAUUGUAUACCUGCAGAUUUAUGAUCAUGCAACCAGCGUGCCGCUUCCACGGCACCUACUCCCAAUC (((((...((((..((((((((((..((.....))..))))).....))))).))))...((.((((((......)))))))).))))).... (-25.96 = -26.83 + 0.88)



| Location | 27,148,942 – 27,149,035 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 93 |
| Reading direction | reverse |
| Mean pairwise identity | 89.39 |
| Mean single sequence MFE | -34.69 |
| Consensus MFE | -28.73 |
| Energy contribution | -30.07 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.16 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.65 |
| SVM RNA-class probability | 0.969805 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 27148942 93 - 27905053 GAUUGGGAGUAGGUGCCGUGGAAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCC ....((((.((((((((((....))))))).)))((((((.(((((.....(((((..(((...)))..))))))))))..)))))))))).. ( -38.30) >DroSec_CAF1 50553 93 - 1 GAUUGGGAGUAGGUGCCGUGGCAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCC ....((((.((((((((((....))))))).)))((((((.(((((.....(((((..(((...)))..))))))))))..)))))))))).. ( -38.30) >DroSim_CAF1 53367 93 - 1 GAUUGGGAGUAGGUGCCGUGGCAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCC ....((((.((((((((((....))))))).)))((((((.(((((.....(((((..(((...)))..))))))))))..)))))))))).. ( -38.30) >DroEre_CAF1 51322 93 - 1 GAUUGGGAGUAGGUGCCGUGGAAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGCAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCC ....((((((..(((((((....)))))))..((.(((((.(((.....)))))))).)).........(((((......))))))))))).. ( -37.00) >DroYak_CAF1 53501 77 - 1 GAUUGGGAGUAGGUG----------------CUGGUUGCAUGAUCAUAAAUCUGCAGGUACACAAUAAAUGCAGUGAUCUCUGCAAUUCCCCC ....((((((..(((----------------.((.(((((.(((.....)))))))).)))))......(((((......))))))))))).. ( -24.50) >DroAna_CAF1 55668 91 - 1 GAUUGGGAGUAGGUGCCGUGGAAGCGGCACGCUCGUUGCAUGAUCAUAAAUCUGCAGGUAGACAAUAAAUGCAAUAAUCUC--CCACUCCUCC ((.((((((...(((((((....)))))))....(((((((.........((((....))))......)))))))...)))--))).)).... ( -31.76) >consensus GAUUGGGAGUAGGUGCCGUGGAAGCGGCACGCUGGUUGCAUGAUCAUAAAUCUGCAGGUAUACAAUAAAUGCAGUGAUCUCUGCAGCUCCCCC ....((((.((((((((((....))))))).)))((((((.(((((.....(((((..((.....))..))))))))))..)))))))))).. (-28.73 = -30.07 + 1.34)

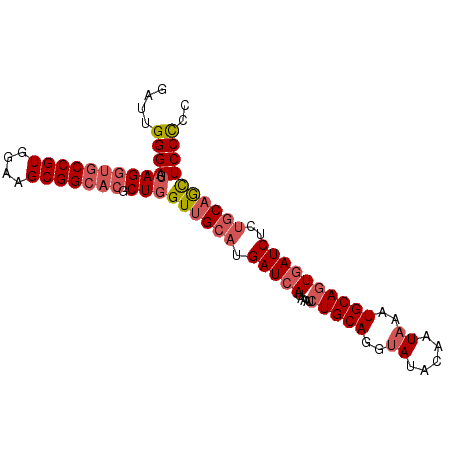

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:49:22 2006