
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 27,012,074 – 27,012,172 |
| Length | 98 |
| Max. P | 0.998071 |

| Location | 27,012,074 – 27,012,172 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 87.99 |
| Mean single sequence MFE | -38.60 |
| Consensus MFE | -33.52 |
| Energy contribution | -34.88 |
| Covariance contribution | 1.36 |
| Combinations/Pair | 1.04 |
| Mean z-score | -5.25 |
| Structure conservation index | 0.87 |
| SVM decision value | 3.00 |
| SVM RNA-class probability | 0.998071 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
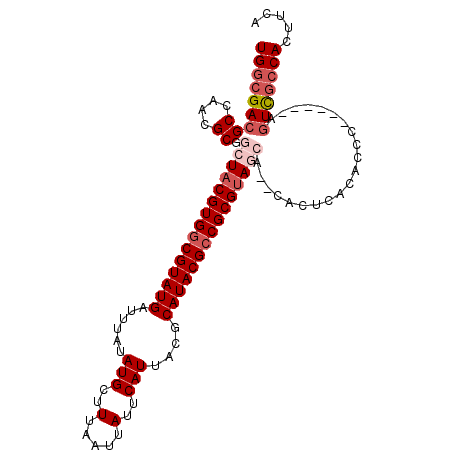
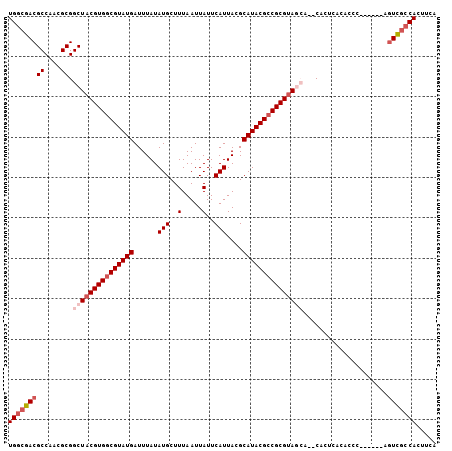
>3R_DroMel_CAF1 27012074 98 + 27905053 UGGCGACGCCAACGCGGCUACGUGGCGUAUGAUUUAUAUGCUUUAAUUAUUCAUUACGCAUACGCCGCGUA--ACACACUCACACCC------AGUCGCCACUUCA ((((((((((.....)))(((((((((((((......(((..(.....)..)))....)))))))))))))--..............------.)))))))..... ( -38.40) >DroSec_CAF1 59468 100 + 1 UGGCGACGCCAACGCGGCUACGUGGCGUAUGAUUUAUAUGCUUUAAUUAUUCAUUACGCAUACGCCGCGUAGCACACACUUACACCC------AGUCGCCACUUCA (((((((((....)).(((((((((((((((......(((..(.....)..)))....)))))))))))))))..............------.)))))))..... ( -41.80) >DroSim_CAF1 59961 100 + 1 UGGCGACGCCAACGCGGCUACGUGGCGUAUGAUUUAUAUGCUUUAAUUAUUCAUUACGCAUACGCCGCGUAGCACACACUCACACCC------AGUCGCCACUUCA (((((((((....)).(((((((((((((((......(((..(.....)..)))....)))))))))))))))..............------.)))))))..... ( -41.80) >DroEre_CAF1 62301 98 + 1 UGGCGACGCCAACGCGGCUACGUGGCGUAUGAUUUAUAUGCUUUAAUUAUUCAUUACGCAUACGACGCGUAGCA--CACUCACACUC------AGUCGCCACUUCA (((((((((....)).((((((((.((((((......(((..(.....)..)))....)))))).)))))))).--...........------.)))))))..... ( -34.70) >DroYak_CAF1 61852 104 + 1 UGGCGACGCCAACGCGGCUACGUGGCGUAUGAUUUAUAUGCUUUAAUUAUUCAUUACGCAUACGCCGCGUAGCA--CACUCAUACUCACACCCGGUUGCCACAUCA (((((((((....)).(((((((((((((((......(((..(.....)..)))....))))))))))))))).--..................)))))))..... ( -39.50) >DroAna_CAF1 63965 92 + 1 UGGCGACGCCAUAGCGGCUACGUGGCGUAUGAUUUAUAUGCUUUAAUUAUUCAUUACGCAUACGCCGCGAACGC--CG------CGA------GCUCCGCACUUCA ..(((..((....(((((..(((((((((((......(((..(.....)..)))....)))))))))))...))--))------)..------))..)))...... ( -35.40) >consensus UGGCGACGCCAACGCGGCUACGUGGCGUAUGAUUUAUAUGCUUUAAUUAUUCAUUACGCAUACGCCGCGUAGCA__CACUCACACCC______AGUCGCCACUUCA (((((((((....)).(((((((((((((((......(((..(.....)..)))....))))))))))))))).....................)))))))..... (-33.52 = -34.88 + 1.36)

| Location | 27,012,074 – 27,012,172 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 87.99 |
| Mean single sequence MFE | -37.65 |
| Consensus MFE | -27.92 |
| Energy contribution | -29.58 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.20 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.32 |
| SVM RNA-class probability | 0.689312 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
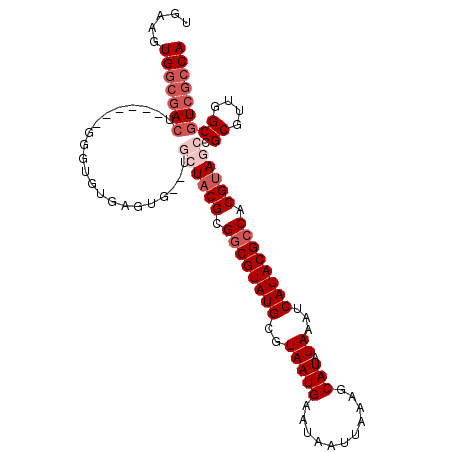
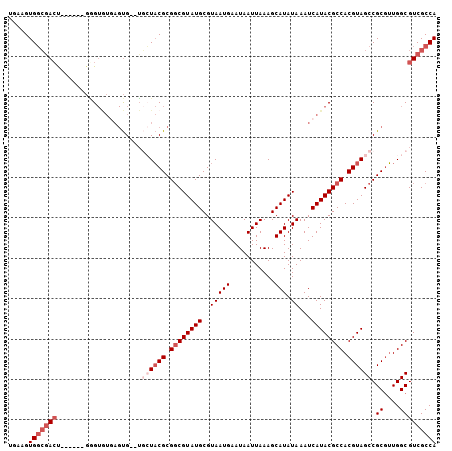
>3R_DroMel_CAF1 27012074 98 - 27905053 UGAAGUGGCGACU------GGGUGUGAGUGUGU--UACGCGGCGUAUGCGUAAUGAAUAAUUAAAGCAUAUAAAUCAUACGCCACGUAGCCGCGUUGGCGUCGCCA .....(((((((.------..(((((((((...--..)))...((((((.((((.....))))..))))))...))))))(((((((....))).))))))))))) ( -36.50) >DroSec_CAF1 59468 100 - 1 UGAAGUGGCGACU------GGGUGUAAGUGUGUGCUACGCGGCGUAUGCGUAAUGAAUAAUUAAAGCAUAUAAAUCAUACGCCACGUAGCCGCGUUGGCGUCGCCA .....(((((((.------....((...((((.((((((.((((((((..(((((...........))).))...)))))))).))))))))))...))))))))) ( -38.50) >DroSim_CAF1 59961 100 - 1 UGAAGUGGCGACU------GGGUGUGAGUGUGUGCUACGCGGCGUAUGCGUAAUGAAUAAUUAAAGCAUAUAAAUCAUACGCCACGUAGCCGCGUUGGCGUCGCCA .....(((((((.------....((.((((((.((((((.((((((((..(((((...........))).))...)))))))).)))))))))))).))))))))) ( -40.50) >DroEre_CAF1 62301 98 - 1 UGAAGUGGCGACU------GAGUGUGAGUG--UGCUACGCGUCGUAUGCGUAAUGAAUAAUUAAAGCAUAUAAAUCAUACGCCACGUAGCCGCGUUGGCGUCGCCA .....(((((((.------..(((((((((--((((((((((...))))))(((.....)))..)))))))...))))))(((((((....))).))))))))))) ( -38.50) >DroYak_CAF1 61852 104 - 1 UGAUGUGGCAACCGGGUGUGAGUAUGAGUG--UGCUACGCGGCGUAUGCGUAAUGAAUAAUUAAAGCAUAUAAAUCAUACGCCACGUAGCCGCGUUGGCGUCGCCA ...((.(....)))((((...((....(((--.((((((.((((((((..(((((...........))).))...)))))))).)))))))))....))..)))). ( -36.30) >DroAna_CAF1 63965 92 - 1 UGAAGUGCGGAGC------UCG------CG--GCGUUCGCGGCGUAUGCGUAAUGAAUAAUUAAAGCAUAUAAAUCAUACGCCACGUAGCCGCUAUGGCGUCGCCA ....(.(((..((------(.(------((--((...((.((((((((..(((((...........))).))...)))))))).))..)))))...)))..)))). ( -35.60) >consensus UGAAGUGGCGACU______GGGUGUGAGUG__UGCUACGCGGCGUAUGCGUAAUGAAUAAUUAAAGCAUAUAAAUCAUACGCCACGUAGCCGCGUUGGCGUCGCCA .....(((((((.....................((((((.((((((((..(((((...........))).))...)))))))).)))))).((....))))))))) (-27.92 = -29.58 + 1.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:48:31 2006