
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 21,398,290 – 21,398,410 |
| Length | 120 |
| Max. P | 0.995494 |

| Location | 21,398,290 – 21,398,410 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.89 |
| Mean single sequence MFE | -26.74 |
| Consensus MFE | -23.56 |
| Energy contribution | -23.92 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.58 |
| SVM RNA-class probability | 0.995494 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
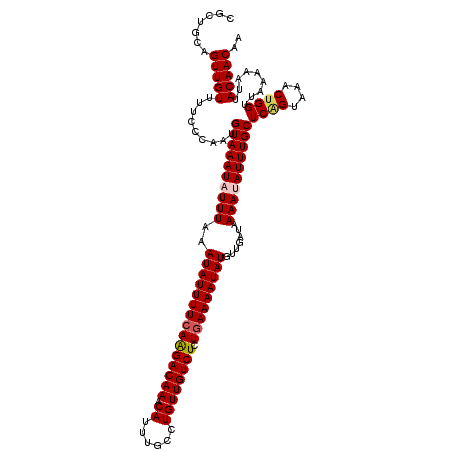
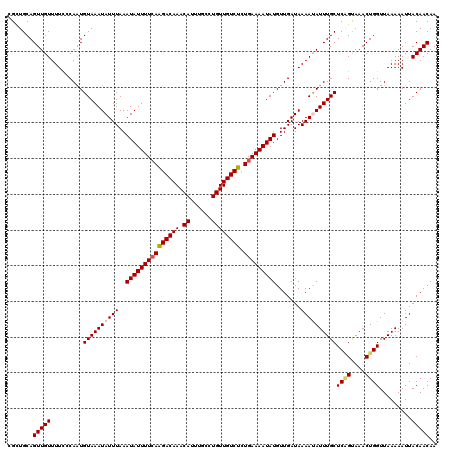
>3L_DroMel_CAF1 21398290 120 + 23771897 CGCUGCAGUUGUUUUCCCAAUGUAAAUAUUUAAAUAUUUUCAGGACAAACAUUUGCGUGUUGUCUCUGAAAAUAUGUUGAUAAAAUAUUUGCUCAGCAAACUGGUUAAAAAUUACAACAA .((((..((((......))))((((((((((..(((((((((((((((.(((....)))))))).)))))))))).......)))))))))).))))....................... ( -28.90) >DroPse_CAF1 17652 120 + 1 CGCUGCAGUUGUUUUUCCAAUGUAAAUAUUUAAAUAUUUUCAAGACAAACAUUUGCCUGUUGUCUCUGAAAAUAUGUUGAUAAAACAUUUGCUCGGUAAACUGGUUAAAAAUUACAACAA ....((((.((((((..((((............(((((((((((((((.((......)))))))).)))))))))))))..)))))).))))((((....))))................ ( -24.30) >DroGri_CAF1 17657 120 + 1 CGUUGCAGUUGUUUUCACAAUGUAAAUAUUUAAAUAUUUUGAAGACAAACAUUUGAUUGUUGUCUCUGAAAAUAUGUUGAUAAAAUAUUUGCUCAGUAAACUGGUUAAAAAUUACAACAA .((((.((((...........((((((((((..(((((((.(((((((.((......)))))))).).))))))).......))))))))))((((....)))).....)))).)))).. ( -23.30) >DroWil_CAF1 31927 120 + 1 CGCUGCAGUUGUUUUCACAAUGUAAAUAUUUAAAUAUUUUCAAGACAAACAUUGAGUUGUUGUCUCUGAAAAUAUGUUGAUAAAAUAUUUGCUCUGUAAACUGGUUAAAAAUUACAACAA .....(((((......(((..((((((((((..(((((((((((((((.((......)))))))).))))))))).......))))))))))..))).)))))................. ( -26.20) >DroAna_CAF1 20327 120 + 1 CGUUGCAGUUGUUUUCCCAGUGUAAAUAUUUAAAUAUUUUCAAGACAAACACCGGCGUGUUGUCUCUGAAAAUAUGUUGAUAAAAUAUUUGCUCAGCAAACUGGUUAAAAAUUACAACAA .((((.((((.(((..(((((((((((((((..(((((((((((((((.(((....))))))))).))))))))).......)))))))))).......)))))..))))))).)))).. ( -33.41) >DroPer_CAF1 17660 120 + 1 CGCUGCAGUUGUUUUUCCAAUGUAAAUAUUUAAAUAUUUUCAAGACAAACAUUUGCCUGUUGUCUCUGAAAAUAUGUUGAUAAAACAUUUGCUCGGUAAACUGGUUAAAAAUUACAACAA ....((((.((((((..((((............(((((((((((((((.((......)))))))).)))))))))))))..)))))).))))((((....))))................ ( -24.30) >consensus CGCUGCAGUUGUUUUCCCAAUGUAAAUAUUUAAAUAUUUUCAAGACAAACAUUUGCCUGUUGUCUCUGAAAAUAUGUUGAUAAAAUAUUUGCUCAGUAAACUGGUUAAAAAUUACAACAA .......(((((.........((((((((((..(((((((((((((((.((......)))))))).))))))))).......))))))))))((((....)))).........))))).. (-23.56 = -23.92 + 0.36)

| Location | 21,398,290 – 21,398,410 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.89 |
| Mean single sequence MFE | -25.46 |
| Consensus MFE | -21.73 |
| Energy contribution | -21.65 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.964172 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
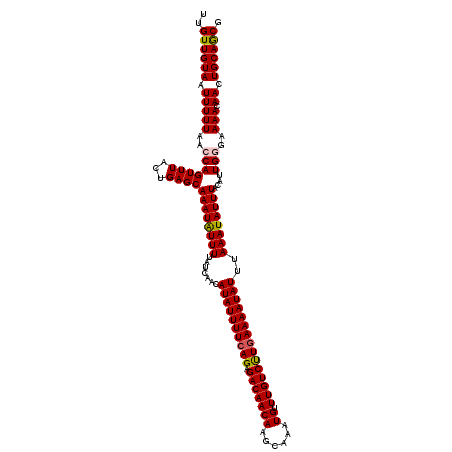
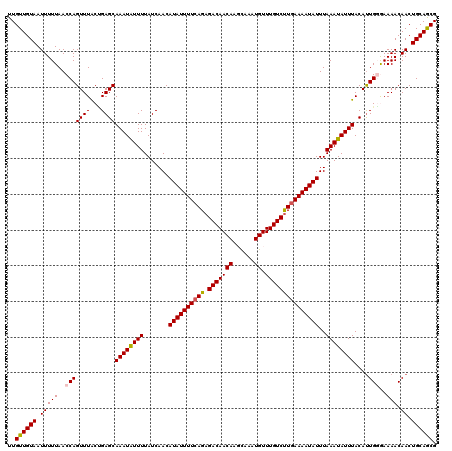
>3L_DroMel_CAF1 21398290 120 - 23771897 UUGUUGUAAUUUUUAACCAGUUUGCUGAGCAAAUAUUUUAUCAACAUAUUUUCAGAGACAACACGCAAAUGUUUGUCCUGAAAAUAUUUAAAUAUUUACAUUGGGAAAACAACUGCAGCG ..((((((.(((((..(((((((((...))))))...........((((((((((.(((((((......)).)))))))))))))))..............)))..))).)).)))))). ( -28.20) >DroPse_CAF1 17652 120 - 1 UUGUUGUAAUUUUUAACCAGUUUACCGAGCAAAUGUUUUAUCAACAUAUUUUCAGAGACAACAGGCAAAUGUUUGUCUUGAAAAUAUUUAAAUAUUUACAUUGGAAAAACAACUGCAGCG ..((((((.(((((..((.((((...)))).(((((..(((....(((((((((.((((((((......)).)))))))))))))))....)))...)))))))..))).)).)))))). ( -23.80) >DroGri_CAF1 17657 120 - 1 UUGUUGUAAUUUUUAACCAGUUUACUGAGCAAAUAUUUUAUCAACAUAUUUUCAGAGACAACAAUCAAAUGUUUGUCUUCAAAAUAUUUAAAUAUUUACAUUGUGAAAACAACUGCAACG ..((((((.........(((....))).(.((((((((.......(((((((..(((((((((......)).))))))).)))))))..)))))))).).((((....)))).)))))). ( -21.70) >DroWil_CAF1 31927 120 - 1 UUGUUGUAAUUUUUAACCAGUUUACAGAGCAAAUAUUUUAUCAACAUAUUUUCAGAGACAACAACUCAAUGUUUGUCUUGAAAAUAUUUAAAUAUUUACAUUGUGAAAACAACUGCAGCG ..((((((............(((((((.(.((((((((.......(((((((((.((((((((......)).)))))))))))))))..)))))))).).)))))))......)))))). ( -24.27) >DroAna_CAF1 20327 120 - 1 UUGUUGUAAUUUUUAACCAGUUUGCUGAGCAAAUAUUUUAUCAACAUAUUUUCAGAGACAACACGCCGGUGUUUGUCUUGAAAAUAUUUAAAUAUUUACACUGGGAAAACAACUGCAACG ..((((((.(((((..(((((..((...))((((((((.......(((((((((.(((((((((....))).)))))))))))))))..))))))))..)))))..))).)).)))))). ( -31.00) >DroPer_CAF1 17660 120 - 1 UUGUUGUAAUUUUUAACCAGUUUACCGAGCAAAUGUUUUAUCAACAUAUUUUCAGAGACAACAGGCAAAUGUUUGUCUUGAAAAUAUUUAAAUAUUUACAUUGGAAAAACAACUGCAGCG ..((((((.(((((..((.((((...)))).(((((..(((....(((((((((.((((((((......)).)))))))))))))))....)))...)))))))..))).)).)))))). ( -23.80) >consensus UUGUUGUAAUUUUUAACCAGUUUACUGAGCAAAUAUUUUAUCAACAUAUUUUCAGAGACAACAAGCAAAUGUUUGUCUUGAAAAUAUUUAAAUAUUUACAUUGGGAAAACAACUGCAGCG ..((((((.(((((..(((((((...))))((((((((.......((((((((((.(((((((......)).)))))))))))))))..))))))))....)))..))).)).)))))). (-21.73 = -21.65 + -0.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:06:54 2006