
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 21,121,217 – 21,121,410 |
| Length | 193 |
| Max. P | 0.971365 |

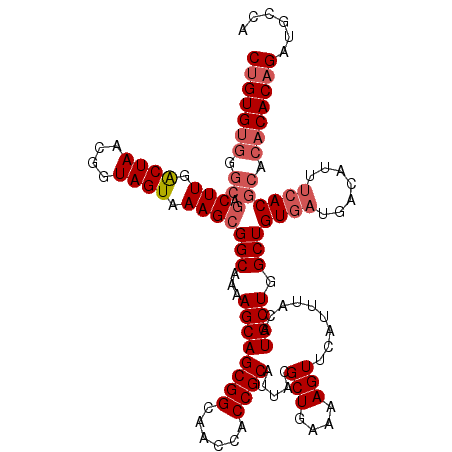
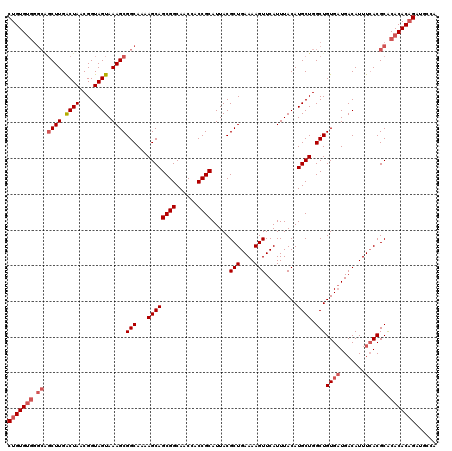
| Location | 21,121,217 – 21,121,330 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.17 |
| Mean single sequence MFE | -38.46 |
| Consensus MFE | -33.48 |
| Energy contribution | -36.34 |
| Covariance contribution | 2.86 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.67 |
| SVM RNA-class probability | 0.971365 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21121217 113 + 23771897 CUGUG-------CUUGACUAACGGUAGUAAAGCGGCAAAAGCAGCGGCAACCACCGCAUUACGCUGAAAAGUUCAUUUACAUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCA ....(-------(((.((((....)))).))))((((..((((((((......)))).....(((....))).........)))).((.((((.......)))))).........)))). ( -31.70) >DroSec_CAF1 171904 120 + 1 CUGUGUGGGCGGCUUGGCUAACGGUAGUAAAGCGGCAAAAGCAGCGGCAACCACCGCAUUACGCUGAAAAGUUCAUUUACAUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCA (((((((.((.((((.((((....)))).))))(((...((((((((......)))).....(((....))).........)))).)))((((.......)))))).)))))))...... ( -41.40) >DroSim_CAF1 83611 120 + 1 CUGUGUGGGCGGCUUGGCUAACGGUAGUAAAGCGGCAAAAGCAGCGGCAACCACCGCAUUACGCUGAAAAGUUCAUUUACAUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCA (((((((.((.((((.((((....)))).))))(((...((((((((......)))).....(((....))).........)))).)))((((.......)))))).)))))))...... ( -41.40) >DroEre_CAF1 94480 120 + 1 CUGUGUGGGCAGCUUGACUAACGGUAGUAAAGCGGCAAAAGCAGCGGCAACCACCGCAUUACGCUGAAAAGUUCAUUUACAUGCUGGCUGUGCUGACAUUUAACGCGCACACAGAUGCCA (((((((.((.((((.((((....)))).))))((((..((((((((......)))).....(((....))).........)))).....))))..........)).)))))))...... ( -40.60) >DroYak_CAF1 65290 120 + 1 CUGUGUGGGCAGCUUGACUAACGGUAGUAAAGCGGCAAAAGCAGCGGCAACCACCGCAUUACGCUGAAAAGUUCAUUUACAUGCUGGCUGUGAUGACAUUUCACGCACACACUGACGCCA ..(((((.((.((((.((((....)))).))))(((...((((((((......)))).....(((....))).........)))).)))((((.......)))))).)))))........ ( -37.20) >consensus CUGUGUGGGCAGCUUGACUAACGGUAGUAAAGCGGCAAAAGCAGCGGCAACCACCGCAUUACGCUGAAAAGUUCAUUUACAUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCA (((((((.((.((((.((((....)))).))))(((...((((((((......)))).....(((....))).........)))).)))((((.......)))))).)))))))...... (-33.48 = -36.34 + 2.86)

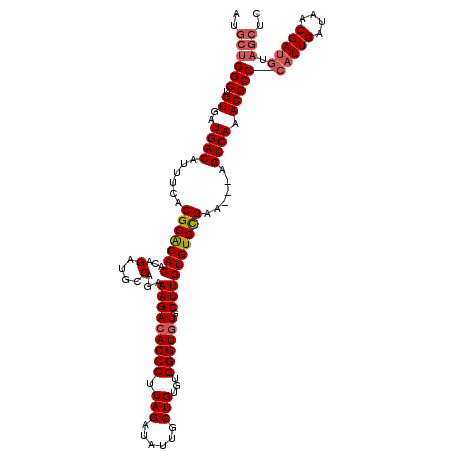
| Location | 21,121,290 – 21,121,410 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.89 |
| Mean single sequence MFE | -35.78 |
| Consensus MFE | -30.54 |
| Energy contribution | -31.34 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.43 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.795504 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21121290 120 + 23771897 AUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCAGAAAGACACCCUUAGAUAUUGCUGUGUGGGUGUGCUUGUGUGCGAAAGAAGUCAAACGCCCAUUGAUAACGAUGUAGCUC ..((((((.((..((((.(((..(((((((...(....)...(((((((((.(((......)))...)))))).))))))))))..))).)))).)))))(((((....))))).))).. ( -37.90) >DroSec_CAF1 171984 117 + 1 AUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCAGAAAGACACCCCUAGAUAUUGCUGUGUGGGUCUGCUUGUGUGCGAA---AGUCAAACGCCCAUUGAUAACGAUGUAGCUC ..((((((.((..((((.((((.((((((..(((((.(((.(.((.((...........)))).).))))))))..)))))).)))---))))).)))))(((((....))))).))).. ( -36.10) >DroSim_CAF1 83691 117 + 1 AUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCAGAAAGACACCCCUAGAUAUUGCUGUGUGGGUGUGCUUGUGUGCGAA---AGUCAAACGCCCAUUGAUAACGAUGUAGCUC ..((((((.((..((((.((((..((((((...(....)...(((((((((((((......))).).)))))).))))))))))))---))))).)))))(((((....))))).))).. ( -35.90) >DroEre_CAF1 94560 109 + 1 AUGCUGGCUGUGCUGACAUUUAACGCGCACACAGAUGCCAGAAAGACACCCUUAGAUAUUGCUGUGUGGGUGUGCUUGUGUGUGAA---AGUCAAACGCCCAUUGAUAACGA-------- .....(((.((..((((.(((....(((((((((..((.......((((((.(((......)))...)))))))))))))))))))---))))).)))))............-------- ( -29.21) >DroYak_CAF1 65370 117 + 1 AUGCUGGCUGUGAUGACAUUUCACGCACACACUGACGCCAUAAAGACACCCUUAGAUAUUGCUGUGUGGGUGUGCUUGUGUGUGAA---AGUCAAACGCCCAUUGAUAACGAUGUAGCUC ..((((((.((..((((.((((((((((....((....))..(((((((((.(((......)))...)))))).))))))))))))---))))).)))))(((((....))))).))).. ( -39.80) >consensus AUGCUGGCUGUGAUGACAUUUCACGCACACACAGAUGCCAGAAAGACACCCUUAGAUAUUGCUGUGUGGGUGUGCUUGUGUGCGAA___AGUCAAACGCCCAUUGAUAACGAUGUAGCUC ..((((((.((..((((......(((((((...(....)...(((((((((.(((......)))...)))))).))))))))))......)))).)))))(((((....))))).))).. (-30.54 = -31.34 + 0.80)


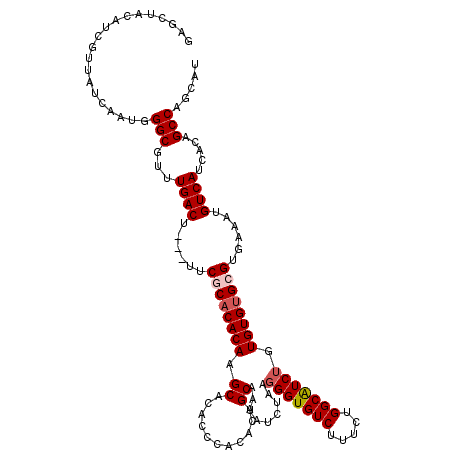
| Location | 21,121,290 – 21,121,410 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.89 |
| Mean single sequence MFE | -34.04 |
| Consensus MFE | -26.98 |
| Energy contribution | -27.62 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.22 |
| SVM RNA-class probability | 0.640290 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21121290 120 - 23771897 GAGCUACAUCGUUAUCAAUGGGCGUUUGACUUCUUUCGCACACAAGCACACCCACACAGCAAUAUCUAAGGGUGUCUUUCUGGCAUCUGUGUGUGCGUGAAAUGUCAUCACAGCCAGCAU ..(((.(((.(....).)))(((...(((((((...((((((((.((...........)).........(((((((.....))))))).)))))))).)))..)))).....)))))).. ( -35.30) >DroSec_CAF1 171984 117 - 1 GAGCUACAUCGUUAUCAAUGGGCGUUUGACU---UUCGCACACAAGCAGACCCACACAGCAAUAUCUAGGGGUGUCUUUCUGGCAUCUGUGUGUGCGUGAAAUGUCAUCACAGCCAGCAU ..(((.(((.(....).)))(((...(((((---(((((.((((.((((((((...............))).((((.....))))))))).)))).)))))).)))).....)))))).. ( -38.06) >DroSim_CAF1 83691 117 - 1 GAGCUACAUCGUUAUCAAUGGGCGUUUGACU---UUCGCACACAAGCACACCCACACAGCAAUAUCUAGGGGUGUCUUUCUGGCAUCUGUGUGUGCGUGAAAUGUCAUCACAGCCAGCAU ..(((.(((.(....).)))(((...(((((---(((((.(((..((((((((...............)))(((((.....))))).)))))))).)))))).)))).....)))))).. ( -35.06) >DroEre_CAF1 94560 109 - 1 --------UCGUUAUCAAUGGGCGUUUGACU---UUCACACACAAGCACACCCACACAGCAAUAUCUAAGGGUGUCUUUCUGGCAUCUGUGUGCGCGUUAAAUGUCAGCACAGCCAGCAU --------............((((((((((.---..((((((...((((((((................)))))).......))...))))))...)))))))))).((.......)).. ( -27.60) >DroYak_CAF1 65370 117 - 1 GAGCUACAUCGUUAUCAAUGGGCGUUUGACU---UUCACACACAAGCACACCCACACAGCAAUAUCUAAGGGUGUCUUUAUGGCGUCAGUGUGUGCGUGAAAUGUCAUCACAGCCAGCAU ..(((.(((.(....).)))(((...(((((---(((((.(((..((((((((................))))(((.....)))....))))))).)))))).)))).....)))))).. ( -34.19) >consensus GAGCUACAUCGUUAUCAAUGGGCGUUUGACU___UUCGCACACAAGCACACCCACACAGCAAUAUCUAAGGGUGUCUUUCUGGCAUCUGUGUGUGCGUGAAAUGUCAUCACAGCCAGCAU ....................(((...((((......((((((((.((...........)).........(((((((.....))))))).))))))))......)))).....)))..... (-26.98 = -27.62 + 0.64)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:03:32 2006