
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 21,119,378 – 21,119,538 |
| Length | 160 |
| Max. P | 0.999937 |

| Location | 21,119,378 – 21,119,498 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.42 |
| Mean single sequence MFE | -37.75 |
| Consensus MFE | -35.20 |
| Energy contribution | -35.04 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.47 |
| SVM RNA-class probability | 0.999904 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21119378 120 + 23771897 UGCAAGUGGAAAGCACAAAAAUCGAUUAAGUUUUUUUCCAAGUCGAUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGUCAGUCUCCCCUCGGACUC (((..(((.....)))..(((((((((.............))))))))))))(((((((((((((.....(((((((.....)))))))))))))))))))).(((((......))))). ( -37.92) >DroSec_CAF1 170027 120 + 1 UGCAAGUGGAGAGCACAAAAAUCGAUUUAGUUUUUUUCCAAGUCGUUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGCCAGUCUCCCCUCGGACUC ..(.((.((((((((...(((.((((((.(.......).)))))))))))).(((((((((((((.....(((((((.....))))))))))))))))))))...))))).)).)..... ( -39.00) >DroSim_CAF1 81742 120 + 1 UGCAAGUGGAGAGCACAAAAAUCGAUUUAGUUUUUUUCCAAGUCGUUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGCCAGUCUCCCCUCGGACUC ..(.((.((((((((...(((.((((((.(.......).)))))))))))).(((((((((((((.....(((((((.....))))))))))))))))))))...))))).)).)..... ( -39.00) >DroEre_CAF1 92525 117 + 1 UGCAAGUGGAAAGCACAAAAAUCGAUAUAGUUU-UUUCCAAGCCGUUUUGCAGCGGCA-AGUUGUUACGCUCAAGUAGUA-GUACUUGAACAACUUUGCUGCCAGUCUCCCCACGGACUC (((((((((((((.((.............)).)-))))))......))))))((((((-((((((.....(((((((...-.)))))))))))).))))))).(((((......))))). ( -33.42) >DroYak_CAF1 63429 120 + 1 UGCAAGUGGAAAGCACAAAAAUCGAUUUAGUUUUUUUCCAAGCUGUUCUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGGGUACUUGAACAACUUUGCUGCCAGUCCCCCCUCGGACUC ((((.(((.....))).......((..((((((......)))))).))))))(((((((((((((.....(((((((.....)))))))))))))))))))).(((((......))))). ( -39.40) >consensus UGCAAGUGGAAAGCACAAAAAUCGAUUUAGUUUUUUUCCAAGUCGUUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGCCAGUCUCCCCUCGGACUC ((((((((((((..((.............))...))))))......))))))(((((((((((((.....(((((((.....)))))))))))))))))))).(((((......))))). (-35.20 = -35.04 + -0.16)

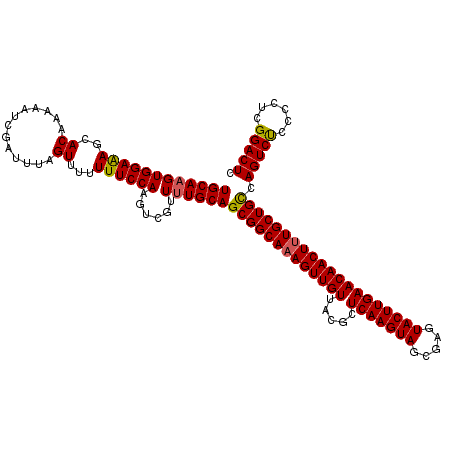

| Location | 21,119,378 – 21,119,498 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.42 |
| Mean single sequence MFE | -38.02 |
| Consensus MFE | -34.40 |
| Energy contribution | -34.80 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.90 |
| SVM decision value | 3.77 |
| SVM RNA-class probability | 0.999602 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21119378 120 - 23771897 GAGUCCGAGGGGAGACUGACAGCAAAGUUGUUCAAGUACUCGCUACUUGAGCGUAACAACUUUGCCGCUGCAAAUCGACUUGGAAAAAAACUUAAUCGAUUUUUGUGCUUUCCACUUGCA .....((((.(((((......(((((((((((((((((.....))))))).....)))))))))).((.(((((((((.((((........)))))))))..))))))))))).)))).. ( -33.70) >DroSec_CAF1 170027 120 - 1 GAGUCCGAGGGGAGACUGGCAGCAAAGUUGUUCAAGUACUCGCUACUUGAGCGUAACAACUUUGCCGCUGCAAAACGACUUGGAAAAAAACUAAAUCGAUUUUUGUGCUCUCCACUUGCA .....((((.(((((..(((.(((((((((((((((((.....))))))).....)))))))))).)))(((((((((.((((.......)))).)))..))))))..))))).)))).. ( -40.40) >DroSim_CAF1 81742 120 - 1 GAGUCCGAGGGGAGACUGGCAGCAAAGUUGUUCAAGUACUCGCUACUUGAGCGUAACAACUUUGCCGCUGCAAAACGACUUGGAAAAAAACUAAAUCGAUUUUUGUGCUCUCCACUUGCA .....((((.(((((..(((.(((((((((((((((((.....))))))).....)))))))))).)))(((((((((.((((.......)))).)))..))))))..))))).)))).. ( -40.40) >DroEre_CAF1 92525 117 - 1 GAGUCCGUGGGGAGACUGGCAGCAAAGUUGUUCAAGUAC-UACUACUUGAGCGUAACAACU-UGCCGCUGCAAAACGGCUUGGAAA-AAACUAUAUCGAUUUUUGUGCUUUCCACUUGCA ..((..((((..((((..(((((.((((((((((((((.-...))))))).....))))))-)...)))))....(((..(((...-...)))..)))......)).))..))))..)). ( -37.50) >DroYak_CAF1 63429 120 - 1 GAGUCCGAGGGGGGACUGGCAGCAAAGUUGUUCAAGUACCCGCUACUUGAGCGUAACAACUUUGCCGCUGCAGAACAGCUUGGAAAAAAACUAAAUCGAUUUUUGUGCUUUCCACUUGCA .(((((......))))).((((((((((((((((((((.....))))))).....)))))))))).((((.....)))).((((((...((.(((.....))).))..))))))..))). ( -38.10) >consensus GAGUCCGAGGGGAGACUGGCAGCAAAGUUGUUCAAGUACUCGCUACUUGAGCGUAACAACUUUGCCGCUGCAAAACGACUUGGAAAAAAACUAAAUCGAUUUUUGUGCUUUCCACUUGCA ..((..(.((((((...(((.(((((((((((((((((.....))))))).....)))))))))).)))(((((((((.((((.......)))).)))..)))))).)))))).)..)). (-34.40 = -34.80 + 0.40)

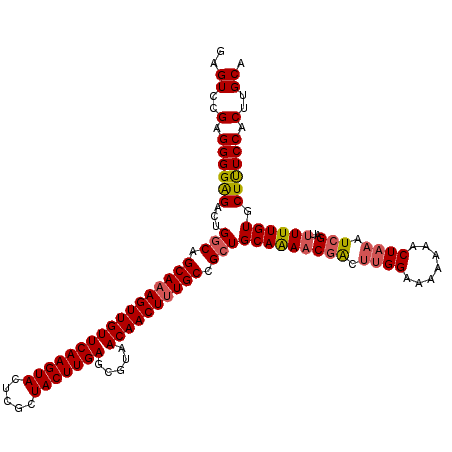

| Location | 21,119,418 – 21,119,538 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.24 |
| Mean single sequence MFE | -35.06 |
| Consensus MFE | -33.94 |
| Energy contribution | -33.82 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.50 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.68 |
| SVM RNA-class probability | 0.999937 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21119418 120 + 23771897 AGUCGAUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGUCAGUCUCCCCUCGGACUCUCUGCACCCUACUAUCAUCCCCAUUCCCCGACAGCUGGCA .((((...(((((((((((((((((.....(((((((.....)))))))))))))))))))..(((((......)))))..))))).....((.((.............)).)).)))). ( -32.42) >DroSec_CAF1 170067 120 + 1 AGUCGUUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGCCAGUCUCCCCUCGGACUCUCUGCACCCCACUAUCAUCCCCAUUCUCCGCUAGCUGGCA .((((((..((.(((((((((((((.....(((((((.....)))))))))))))))))))).(((((......)))))..............................)).))).))). ( -35.10) >DroSim_CAF1 81782 120 + 1 AGUCGUUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGCCAGUCUCCCCUCGGACUCUCUGCACCCCACUAUCAUCCCCAUUCUCCGCCAGCUGGCA (((.(...(((((((((((((((((.....(((((((.....)))))))))))))))))))).(((((......)))))...))))..).)))................(((....))). ( -37.40) >DroEre_CAF1 92564 116 + 1 AGCCGUUUUGCAGCGGCA-AGUUGUUACGCUCAAGUAGUA-GUACUUGAACAACUUUGCUGCCAGUCUCCCCACGGACUCUAC--ACCCCACUAUCAUCCCCAUUCCCCGCCAGCUGGCA .((((.((.((.((((((-((((((.....(((((((...-.)))))))))))).))))))).(((((......)))))....--........................)).)).)))). ( -32.60) >DroYak_CAF1 63469 117 + 1 AGCUGUUCUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGGGUACUUGAACAACUUUGCUGCCAGUCCCCCCUCGGACUCCAU--AC-ACACUAUCAUCCCCAUUUCCCGCCAGCUGGCA (((((....((.(((((((((((((.....(((((((.....)))))))))))))))))))).(((((......)))))....--..-.....................))))))).... ( -37.80) >consensus AGUCGUUUUGCAGCGGCAAAGUUGUUACGCUCAAGUAGCGAGUACUUGAACAACUUUGCUGCCAGUCUCCCCUCGGACUCUCUGCACCCCACUAUCAUCCCCAUUCCCCGCCAGCUGGCA .((((.((.((.(((((((((((((.....(((((((.....)))))))))))))))))))).(((((......)))))..............................)).)).)))). (-33.94 = -33.82 + -0.12)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:03:19 2006