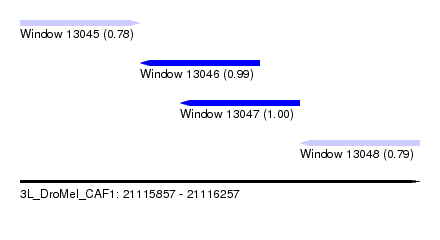
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 21,115,857 – 21,116,257 |
| Length | 400 |
| Max. P | 0.999936 |

| Location | 21,115,857 – 21,115,977 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.33 |
| Mean single sequence MFE | -38.64 |
| Consensus MFE | -33.54 |
| Energy contribution | -33.62 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.783717 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21115857 120 + 23771897 CAUGCGAAUGCUGCUUAACAUUCUGGAGGCGGAGCAUACGGACGUGGUCAUCGCUGGCCUGCACGUGCUCAGUAAGAUUAUGAGGAGCAACAAAAUGCGUCACAACUGGAUGCACUUUCU .....(((((((.((((....(((....((.((((..(((..((.(((((....)))))))..))))))).)).)))...)))).))))......((((((.(....)))))))..))). ( -35.30) >DroSec_CAF1 166457 120 + 1 CAUGCGAAUGCUCCUUAACAUUCUGGAGGCGGAGCACACGGACGUGGUCAUCGCUGGUCUGCAUGUGCUCAGUAAGAUUAUGAGGAGCAACAAAAUGCGUCACAACUGGAUGCACUUCCU ........(((((((((....(((....((.((((((((((((.((((....)))))))))..))))))).)).)))...)))))))))......((((((.(....)))))))...... ( -41.90) >DroSim_CAF1 78166 120 + 1 CAUGCGAAUGCUCCUUAACAUUCUGGAGGCGGAGCACACGGACUUGGUCAUCGCUGGCCUGCAUGUGCUCAGUAAGAUUAUGAGGAGCAACAAAAUGCGUCACAACUGGAUGCACUUCCU ........(((((((((....(((....((.(((((((..(....(((((....)))))..).))))))).)).)))...)))))))))......((((((.(....)))))))...... ( -40.50) >DroEre_CAF1 88982 120 + 1 CAUGCGAAUGCUUCUCAACAUUCUGGAGGCGGAGCACACCGACGUGGUCAUCGCCGGCCUGCACGUGCUCAGUAAGAUAAUGAGGAGCAACAAAAUGCGUCACAACUGGAUGCACUUCCU ........(((((((((....(((....((.((((((...(.((.((((......)))))))..)))))).)).)))...)))))))))......((((((.(....)))))))...... ( -37.60) >DroYak_CAF1 59835 120 + 1 CAUGCGAAUGCUUCUCAACAUUCUGGAGGCGGAUCACACGGACGUGGUCAUCGCCGGCCUGCACGUACUCAGUAAAAUAAUGAGGAGCAACAAAAUGCGUCACAACUGGAUGCAUUUCCU ........(((((((((.......((.((((((((((......))))))..))))..))...((.......)).......)))))))))...(((((((((.(....))))))))))... ( -37.90) >consensus CAUGCGAAUGCUCCUUAACAUUCUGGAGGCGGAGCACACGGACGUGGUCAUCGCUGGCCUGCACGUGCUCAGUAAGAUUAUGAGGAGCAACAAAAUGCGUCACAACUGGAUGCACUUCCU ........(((((((((....(((....((.((((((...(....((((......))))..)..)))))).)).)))...)))))))))......((((((.(....)))))))...... (-33.54 = -33.62 + 0.08)



| Location | 21,115,977 – 21,116,097 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.83 |
| Mean single sequence MFE | -40.12 |
| Consensus MFE | -34.80 |
| Energy contribution | -34.64 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.11 |
| SVM RNA-class probability | 0.988298 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21115977 120 - 23771897 AUGUUGAUGGACAGAUCCAGAGGCAAGGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCCCGCAAAGCCUCCUUGCUGUGUUGAUAGCACUGGAUGAUCUUCAGCAAAAUCAGCUCU .((((((.(((...((((((((((...((((((((((....))))))))))..(.((......)))...)))))..((((((....)))))).)))))..)))))))))........... ( -39.70) >DroSec_CAF1 166577 120 - 1 AUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCAAUAUCCCGCAAAGCCUCCUUGCUGUGUUGAUAGCACUGGAUGAUCUUCAGCAAAAUCAGCUCC .((((((.(((...((((((..((...((((((((((....))))))))))..((((((((...((((.......)))).)))))))).)).))))))..)))))))))........... ( -39.50) >DroSim_CAF1 78286 120 - 1 AUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCAAUAUCCCGCAAAGCCUCCUUGCUGUGUUGAUAGCACUGGAUGAUCUUCAGCAAAAUCAGCUCC .((((((.(((...((((((..((...((((((((((....))))))))))..((((((((...((((.......)))).)))))))).)).))))))..)))))))))........... ( -39.50) >DroEre_CAF1 89102 120 - 1 AUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCUCGCAAAGCCUCUUUGCUGUGCUGAUAGCACUGGAUGAUCUUGAGCAGGAUCAGCUCC .(((((.((((....)))).)))))..((((((((((....))))))))))((((....((((.((((((...)))))).((((.....)))).))))((((((....)))))).)))). ( -42.90) >DroYak_CAF1 59955 120 - 1 AUAUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCCCGUAAAGCCUCCUUGCUGUGCUGAUAGCACUGGAUGAUCUUGAGCAGGAUCAGCUCC .......((((....))))(((((...((((((((((....))))))))))....((......))....)))(((.((((((....))))))..)))(((((((....)))))))))... ( -39.00) >consensus AUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCCCGCAAAGCCUCCUUGCUGUGUUGAUAGCACUGGAUGAUCUUCAGCAAAAUCAGCUCC ..(((((((((....))).(((((...((((((((((....))))))))))..)))..(((((.((((.......)))).((((.....)))).))))).......))...))))))... (-34.80 = -34.64 + -0.16)


| Location | 21,116,017 – 21,116,137 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.50 |
| Mean single sequence MFE | -42.38 |
| Consensus MFE | -39.12 |
| Energy contribution | -39.16 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.92 |
| SVM decision value | 4.67 |
| SVM RNA-class probability | 0.999936 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21116017 120 - 23771897 AUUUGUGGGAAACUCACCCGUCGCAAUUACCGGAUUGACAAUGUUGAUGGACAGAUCCAGAGGCAAGGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCCCGCAAAGCCUCCUUGCU .(((((((((.......(((..........)))((((((..((((..((((....))))..))))..((((((((((....))))))))))..)))))).)))))))))((......)). ( -41.30) >DroSec_CAF1 166617 120 - 1 AUUUGUGGGAAACUCACCAGUCGCAAUCACUGGAUUGACAAUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCAAUAUCCCGCAAAGCCUCCUUGCU .(((((((((......(((((.......)))))((((((..(((((.((((....)))).)))))..((((((((((....))))))))))..)))))).)))))))))((......)). ( -47.90) >DroSim_CAF1 78326 120 - 1 AUUUGUGGGAAACUCACCAGUCGCAAUCACUGGAUUGACAAUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCAAUAUCCCGCAAAGCCUCCUUGCU .(((((((((......(((((.......)))))((((((..(((((.((((....)))).)))))..((((((((((....))))))))))..)))))).)))))))))((......)). ( -47.90) >DroEre_CAF1 89142 120 - 1 AUUCGUGGGAAACUCGCCCGUCGCAAUCACAGGAUUGACAAUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCUCGCAAAGCCUCUUUGCU ...((((((.......)))).)).......(((((((((..(((((.((((....)))).)))))..((((((((((....))))))))))..)))))).))).((((((...)))))). ( -39.10) >DroYak_CAF1 59995 120 - 1 AUUUGUGGGAAACUCUCCCGUCGCAAUAACAGGAUUGACAAUAUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCCCGUAAAGCCUCCUUGCU .(((((((((.((((....(((((.......(......)........((((....)))))))))...((((((((((....)))))))))))))).....)))))))))((......)). ( -35.70) >consensus AUUUGUGGGAAACUCACCCGUCGCAAUCACAGGAUUGACAAUGUUGAUGGACAGGUCCAGCGGCAAAGAUGGUGCUAUCCUUGGUAUCAUCGAGUCGAUAUCCCGCAAAGCCUCCUUGCU .(((((((((......((.((.......)).))((((((..(((((.((((....)))).)))))..((((((((((....))))))))))..)))))).)))))))))((......)). (-39.12 = -39.16 + 0.04)



| Location | 21,116,137 – 21,116,257 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -45.56 |
| Consensus MFE | -39.26 |
| Energy contribution | -40.86 |
| Covariance contribution | 1.60 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.44 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.790473 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 21116137 120 - 23771897 CAUCGAUUGCGUGUCGUCCGCCGAUCGCGCCAGAUUGGGGAACACAAUGUCCAAGUGGGCGUCUGUGAUCUCCGAACCAUGGUGUUCGGUCACCUCCAGCAGGAUCUUGAUGGCGCACAA ......(((.((((((((.(((((..((((((..(((((((.((((((((((....)))))).)))).)))))))....)))))))))))....(((....)))....)))))))).))) ( -50.20) >DroSec_CAF1 166737 120 - 1 CAUCGAUUGCGUGUCGUCCGCCGAACGCGCCAGAUUGGGGAACACAAUGUCCAAGUGGGCGUCUGUGAUCUCCGAGCCAUGGUGUUCGGUCACCUCCAGCAGGAUCUUGAUGGCGCACAA ......(((.((((((((.(((((..((((((..(((((((.((((((((((....)))))).)))).)))))))....)))))))))))....(((....)))....)))))))).))) ( -51.30) >DroSim_CAF1 78446 120 - 1 CAUCGAUUGCGUAUCGUCCGCCGAUCGCGCCAGAUUGGGGAACACAAUGUCCAAGUGGGCGUCUGUGAUCUCCGAGCCAUGGUGUUCGGUCACCUCCAGCAGGAUCUUGAUGGCGCACAA .......(((((..((((.(((((..((((((..(((((((.((((((((((....)))))).)))).)))))))....)))))))))))....(((....)))....)))))))))... ( -47.50) >DroEre_CAF1 89262 120 - 1 CAUCGAUUGCGUGUCGUCCGCUGAUCGCGCGAGGUUCGGGAACACAAUAUCCAAGUGAGCGUCAGUGAUCUCCGAGCCAUGGUGUUCGGUCACCUCCAGCAGGAUCUUGAUGGCGCACAA ......(((.((((((((....((((..((..(((((((((.(((....((.....))......))).)).)))))))..((((......))))....))..))))..)))))))).))) ( -40.10) >DroYak_CAF1 60115 120 - 1 CAUCGAUUGCGUGUCGUCCGCUGAUCGCGCAAGGUUAGGAAACACAAUAUCCAAGUGGGCAUCUGUGAUCUCCGAGCCAUGGUGUUCGGUCACCUCCAGCAGGAUCUUGAUGGCGCACAA ......(((.((((((((....((((..((..((((.(((..((((((..((....))..)).))))...))).))))..((((......))))....))..))))..)))))))).))) ( -38.70) >consensus CAUCGAUUGCGUGUCGUCCGCCGAUCGCGCCAGAUUGGGGAACACAAUGUCCAAGUGGGCGUCUGUGAUCUCCGAGCCAUGGUGUUCGGUCACCUCCAGCAGGAUCUUGAUGGCGCACAA ......(((.((((((((.(((((..((((((..(((((((.((((((((((....)))))).)))).)))))))....)))))))))))....(((....)))....)))))))).))) (-39.26 = -40.86 + 1.60)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:03:10 2006