
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 20,864,625 – 20,864,761 |
| Length | 136 |
| Max. P | 0.994221 |

| Location | 20,864,625 – 20,864,740 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 90.79 |
| Mean single sequence MFE | -36.88 |
| Consensus MFE | -29.32 |
| Energy contribution | -29.32 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.80 |
| SVM decision value | 2.46 |
| SVM RNA-class probability | 0.994221 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20864625 115 + 23771897 UUUCCAUAGCCGUUUUUCCAAUUAUUCGCGCUGCUGCAGCAGCAGCUGCUGCCAUAUAGCAUAUA---UGUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAU ..((((((((.(((..(((........(((((((((...))))))).)).....((((((...((---(((((....))))))))))))))))..)))....)))...)))))..... ( -35.40) >DroSec_CAF1 121260 115 + 1 UUUCCAUAGCAGUUUUUCCAAUUAUUCGCGCUGCUGCAGCAGCAGCUGCUGCCAUAUAGCAUAUG---UAUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAU ..((((((.((((...((.........(((((((((...))))))).))..((.((((((.((((---((((....)))))))))))))))).......)).)))).))))))..... ( -39.80) >DroSim_CAF1 123623 115 + 1 UUUCCAUAGCCGUUUUUCCAAUUAUUCGCGCUGCUGCAGCAGCAGCUGCUGCCAUAUAGCAUAUG---UAUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAU ..((((((((.(((..(((........(((((((((...))))))).)).....((((((.((((---((((....)))))))))))))))))..)))....)))...)))))..... ( -38.00) >DroEre_CAF1 121615 118 + 1 UUUCCAUAGCCGUUUUUCCAAUUAUUCGCGCUGCUGCAGCAGCUGCCACUGCCACAUAGCACAUAGCACAUAUAUGUGUACAUGGCUAUAGGUUUAACCGAUGCUGAUGUGGGCAGAU ...........................(((((((....))))).))..(((((((((((((((((((.(((.((....)).)))))))).((.....))).)))).)))).))))).. ( -34.50) >DroYak_CAF1 121652 110 + 1 UUUCCAUAGCCGUUUUUCCAAUUAUUCGCGCUGCUGCAGCAGCUGCCUCUGCCAUAUAGCAUAUA---CAUA-----GCAUAUAGCUAUGGGUUUAACCGAUGCUGAUGUGGGCAGAU ...........................(((((((....))))).)).(((((((((((((((...---((((-----((.....))))))((.....)).))))).)))).)))))). ( -36.70) >consensus UUUCCAUAGCCGUUUUUCCAAUUAUUCGCGCUGCUGCAGCAGCAGCUGCUGCCAUAUAGCAUAUA___UAUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAU ...........................(((((((....))))).))..(((((.....(((((((.......)))))))(((((((....((.....))...))).)))).))))).. (-29.32 = -29.32 + 0.00)

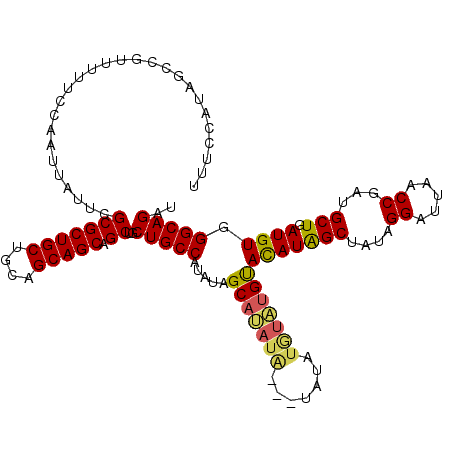
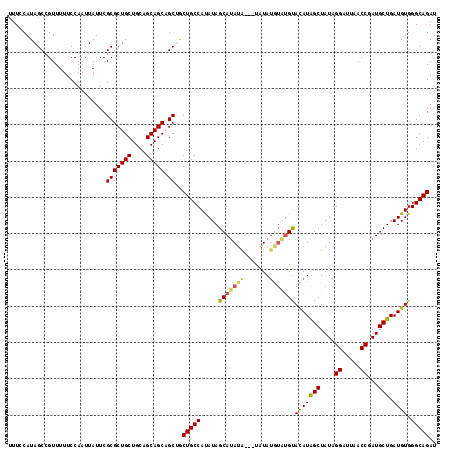
| Location | 20,864,625 – 20,864,740 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 90.79 |
| Mean single sequence MFE | -34.08 |
| Consensus MFE | -27.06 |
| Energy contribution | -27.18 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.56 |
| Structure conservation index | 0.79 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.986835 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20864625 115 - 23771897 AUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUACA---UAUAUGCUAUAUGGCAGCAGCUGCUGCUGCAGCAGCGCGAAUAAUUGGAAAAACGGCUAUGGAAA .........(((.(((....(((..(((....((((((((((((......---..)))).)))))))).((.(((((((...)))))))))........)))..))).)))))).... ( -32.40) >DroSec_CAF1 121260 115 - 1 AUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUAUA---CAUAUGCUAUAUGGCAGCAGCUGCUGCUGCAGCAGCGCGAAUAAUUGGAAAAACUGCUAUGGAAA .........(((.(((((((((((..((((((((((((((........))---)))).)))))).))..((.(((((((...)))))))))...)))))))......))))))).... ( -33.20) >DroSim_CAF1 123623 115 - 1 AUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUAUA---CAUAUGCUAUAUGGCAGCAGCUGCUGCUGCAGCAGCGCGAAUAAUUGGAAAAACGGCUAUGGAAA .........(((.(((....(((..(((....((((((((((((......---..)))).)))))))).((.(((((((...)))))))))........)))..))).)))))).... ( -32.40) >DroEre_CAF1 121615 118 - 1 AUCUGCCCACAUCAGCAUCGGUUAAACCUAUAGCCAUGUACACAUAUAUGUGCUAUGUGCUAUGUGGCAGUGGCAGCUGCUGCAGCAGCGCGAAUAAUUGGAAAAACGGCUAUGGAAA ..(((((.((((.(((((.((.....)).(((((((((((....)))))).)))))))))))))))))))((((.(((((....))))).(((....)))........))))...... ( -39.10) >DroYak_CAF1 121652 110 - 1 AUCUGCCCACAUCAGCAUCGGUUAAACCCAUAGCUAUAUGC-----UAUG---UAUAUGCUAUAUGGCAGAGGCAGCUGCUGCAGCAGCGCGAAUAAUUGGAAAAACGGCUAUGGAAA ...(((........)))..........((((((((...(((-----((((---((.....)))))))))...((.(((((....)))))))................))))))))... ( -33.30) >consensus AUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUAUA___UAUAUGCUAUAUGGCAGCAGCUGCUGCUGCAGCAGCGCGAAUAAUUGGAAAAACGGCUAUGGAAA .........(((.(((....(((..(((....((((((((((((...........)))).))))))))....((.(((((....)))))))........)))..))).)))))).... (-27.06 = -27.18 + 0.12)

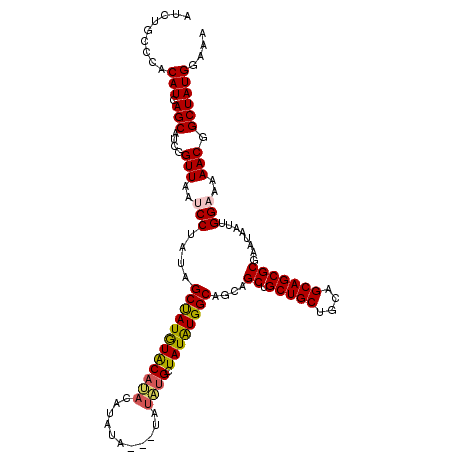
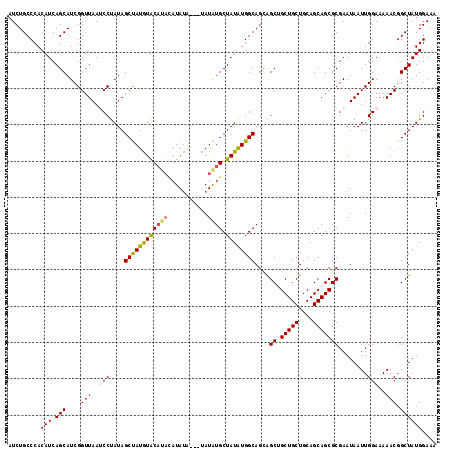
| Location | 20,864,663 – 20,864,761 |
|---|---|
| Length | 98 |
| Sequences | 5 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 89.62 |
| Mean single sequence MFE | -34.12 |
| Consensus MFE | -25.88 |
| Energy contribution | -25.88 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.79 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.79 |
| SVM RNA-class probability | 0.977565 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20864663 98 + 23771897 GCAGCAGCUGCUGCCAUAUAGCAUAUA---UGUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAUUUCGAUCAGUUCGCCAGCUGG ....((((((((((((((((((((.((---(((((....)))))))......((.....)).))))).)))).)))))....(((.....))).)))))). ( -32.20) >DroSec_CAF1 121298 98 + 1 GCAGCAGCUGCUGCCAUAUAGCAUAUG---UAUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAUUUCGAUCAGUUCGCCAGCUGG (((((....)))))..((((((.((((---((((....))))))))))))))((.....))...((((..((((((.(((....))).))))))))))... ( -33.60) >DroSim_CAF1 123661 98 + 1 GCAGCAGCUGCUGCCAUAUAGCAUAUG---UAUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAUUUCGAUCAGUUCGCCAGCUGG (((((....)))))..((((((.((((---((((....))))))))))))))((.....))...((((..((((((.(((....))).))))))))))... ( -33.60) >DroEre_CAF1 121653 101 + 1 GCAGCUGCCACUGCCACAUAGCACAUAGCACAUAUAUGUGUACAUGGCUAUAGGUUUAACCGAUGCUGAUGUGGGCAGAUUUCGAUCAGUUCGCCAGCUGG .((((((.(.(((((((((((((((((((.(((.((....)).)))))))).((.....))).)))).)))).))))).....((.....))).)))))). ( -34.50) >DroYak_CAF1 121690 93 + 1 GCAGCUGCCUCUGCCAUAUAGCAUAUA---CAUA-----GCAUAUAGCUAUGGGUUUAACCGAUGCUGAUGUGGGCAGAUUUCGAUCAGUUCGCCAGCUGG .((((((..(((((((((((((((...---((((-----((.....))))))((.....)).))))).)))).))))))...(((.....))).)))))). ( -36.70) >consensus GCAGCAGCUGCUGCCAUAUAGCAUAUA___UAUAUGUAUGUACAUAGCUAUAGGAUUAACCGAUGCUGAUGUGGGCAGAUUUCGAUCAGUUCGCCAGCUGG .((((.....(((((.....(((((((.......)))))))(((((((....((.....))...))).)))).)))))....(((.....)))...)))). (-25.88 = -25.88 + 0.00)

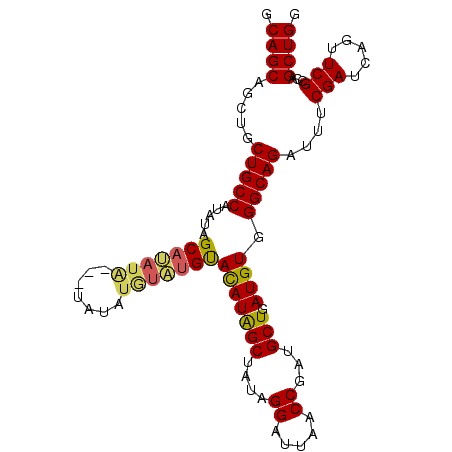
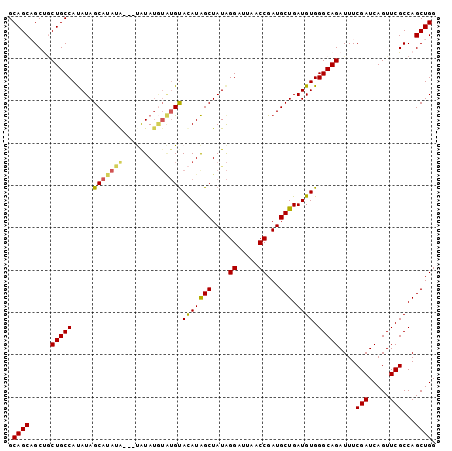
| Location | 20,864,663 – 20,864,761 |
|---|---|
| Length | 98 |
| Sequences | 5 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 89.62 |
| Mean single sequence MFE | -31.00 |
| Consensus MFE | -23.16 |
| Energy contribution | -23.28 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.06 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.88 |
| SVM RNA-class probability | 0.981106 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20864663 98 - 23771897 CCAGCUGGCGAACUGAUCGAAAUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUACA---UAUAUGCUAUAUGGCAGCAGCUGCUGC .((((((.(((.....)))....(((((............((.....))((((((((((((......))))---))...)))))).))))))))))).... ( -26.90) >DroSec_CAF1 121298 98 - 1 CCAGCUGGCGAACUGAUCGAAAUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUAUA---CAUAUGCUAUAUGGCAGCAGCUGCUGC .((((((.(((.....)))....(((((............((.....))((((((((((((........))---)))).)))))).))))))))))).... ( -28.70) >DroSim_CAF1 123661 98 - 1 CCAGCUGGCGAACUGAUCGAAAUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUAUA---CAUAUGCUAUAUGGCAGCAGCUGCUGC .((((((.(((.....)))....(((((............((.....))((((((((((((........))---)))).)))))).))))))))))).... ( -28.70) >DroEre_CAF1 121653 101 - 1 CCAGCUGGCGAACUGAUCGAAAUCUGCCCACAUCAGCAUCGGUUAAACCUAUAGCCAUGUACACAUAUAUGUGCUAUGUGCUAUGUGGCAGUGGCAGCUGC .((((((.(((.....)).....(((((.((((.(((((.((.....)).(((((((((((....)))))).))))))))))))))))))).).)))))). ( -38.10) >DroYak_CAF1 121690 93 - 1 CCAGCUGGCGAACUGAUCGAAAUCUGCCCACAUCAGCAUCGGUUAAACCCAUAGCUAUAUGC-----UAUG---UAUAUGCUAUAUGGCAGAGGCAGCUGC .((((((.(((.....))....((((((......(((((.((.....))((((((.....))-----))))---...)))))....))))))).)))))). ( -32.60) >consensus CCAGCUGGCGAACUGAUCGAAAUCUGCCCACAUCAGCAUCGGUUAAUCCUAUAGCUAUGUACAUACAUAUA___UAUAUGCUAUAUGGCAGCAGCUGCUGC .((((.(((....(((((((....(((........))))))))))........((((((((((((...........)))).))))))))....))))))). (-23.16 = -23.28 + 0.12)

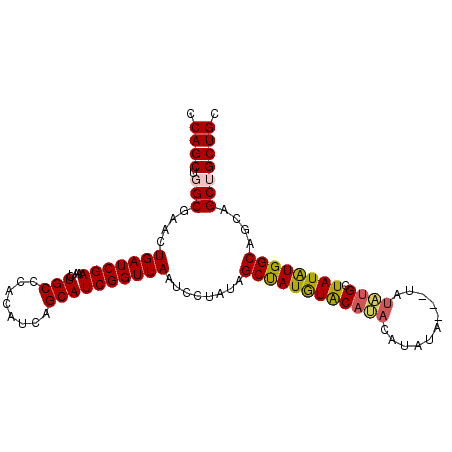
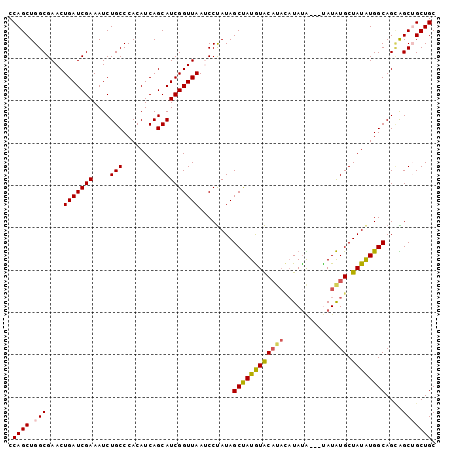
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:57:22 2006