
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 20,610,722 – 20,610,877 |
| Length | 155 |
| Max. P | 0.998256 |

| Location | 20,610,722 – 20,610,837 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.93 |
| Mean single sequence MFE | -33.80 |
| Consensus MFE | -27.78 |
| Energy contribution | -28.68 |
| Covariance contribution | 0.90 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967356 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20610722 115 + 23771897 -----CUCUACCCACCGCAUUAUUAAUAAUGCCUGAGCUGAGCUGAGCUGGGCCAGGCAGAGCGUUUUGACCACCGCCAAAGCGGUUUCAAAUGCUGGCCCAAUGCAAUUUUAUUAACGC -----...........(((((........((((((.(((.(((...))).))))))))).(((((((.((..(((((....))))).))))))))).....))))).............. ( -38.60) >DroSec_CAF1 55556 110 + 1 -----CUCUACCCACCGCAUUAUUAAUAAUGCCUGAGCU-----GAGCUGGGCCAGGCAGAGCGUUUUGACCACCGCCAAAGCGGUUUCAAAUGCUGGCCCAAUGCAAUUUUAUUAACGC -----...........(((((........((((((.(((-----......))))))))).(((((((.((..(((((....))))).))))))))).....))))).............. ( -33.30) >DroAna_CAF1 77393 110 + 1 UCGGCCUCUACCCACCGCAUUAUUAAUAAUGCCUGAG----------CUGCGCCAAGCCGGGCGUUUUGACCACCGCCAAAGCCGUUUCAAAUGCAGGCCCAAUGCAAUUUUAUUAACGC ..(((((.........(((((.........(((((.(----------((......))))))))((((((........)))))).......)))))))))).................... ( -28.20) >DroSim_CAF1 50448 110 + 1 -----CUCUACCCACCGCAUUAUUAAUAAUGCCUGAGCU-----GAGCUGGGCCAGGCAGAGCGUUUUGACCACCGCCAAAGCGGUUUCAAAUGCUGGCCCAAUGCAAUUUUAUUAACGC -----...........(((((........((((((.(((-----......))))))))).(((((((.((..(((((....))))).))))))))).....))))).............. ( -33.30) >DroEre_CAF1 48091 110 + 1 -----CUCUACCCACCGCAUUAUUAAUAAUGCUGGAGCU-----GAGCUGGGCCAGGCAGAGCGUUUUGACCACCGCCAAAGCGGUUUCAAAUGCUGGCCCAAUGCAAUUUUAUUAACGC -----(((..((((..((((((....))))))))).)..-----))).((((((((((...))..(((((..(((((....))))).)))))..)))))))).................. ( -35.60) >consensus _____CUCUACCCACCGCAUUAUUAAUAAUGCCUGAGCU_____GAGCUGGGCCAGGCAGAGCGUUUUGACCACCGCCAAAGCGGUUUCAAAUGCUGGCCCAAUGCAAUUUUAUUAACGC ................(((((..............((((......))))(((((((((...))..(((((..(((((....))))).)))))..)))))))))))).............. (-27.78 = -28.68 + 0.90)

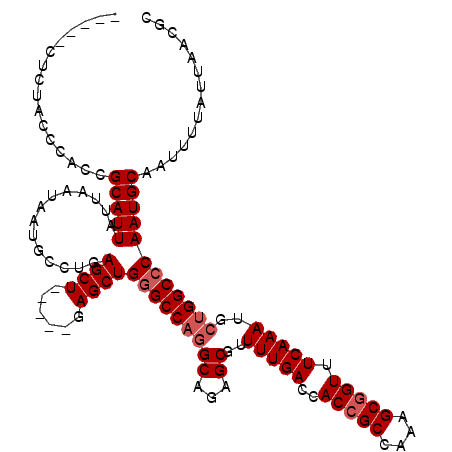

| Location | 20,610,722 – 20,610,837 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.93 |
| Mean single sequence MFE | -41.24 |
| Consensus MFE | -34.64 |
| Energy contribution | -35.54 |
| Covariance contribution | 0.90 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.67 |
| Structure conservation index | 0.84 |
| SVM decision value | 3.05 |
| SVM RNA-class probability | 0.998256 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20610722 115 - 23771897 GCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUCAGCUCAGCUCAGGCAUUAUUAAUAAUGCGGUGGGUAGAG----- (((..........))).((((((((((((((((.(((((....)))))..)))))......)).)))))))))(((.(((((.(....((((((....))))))).))))).)))----- ( -42.50) >DroSec_CAF1 55556 110 - 1 GCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC-----AGCUCAGGCAUUAUUAAUAAUGCGGUGGGUAGAG----- (((..........))).((((((((((((((((.(((((....)))))..)))))......)).)))))))))(((-----.(((((.((((((....))))))..))))).)))----- ( -42.10) >DroAna_CAF1 77393 110 - 1 GCGUUAAUAAAAUUGCAUUGGGCCUGCAUUUGAAACGGCUUUGGCGGUGGUCAAAACGCCCGGCUUGGCGCAG----------CUCAGGCAUUAUUAAUAAUGCGGUGGGUAGAGGCCGA ((((((.(((...(((.((((((.(((.(((((.((.((....)).))..)))))..(((......)))))))----------))))))))...))).))))))....(((....))).. ( -37.40) >DroSim_CAF1 50448 110 - 1 GCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC-----AGCUCAGGCAUUAUUAAUAAUGCGGUGGGUAGAG----- (((..........))).((((((((((((((((.(((((....)))))..)))))......)).)))))))))(((-----.(((((.((((((....))))))..))))).)))----- ( -42.10) >DroEre_CAF1 48091 110 - 1 GCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC-----AGCUCCAGCAUUAUUAAUAAUGCGGUGGGUAGAG----- (((..........))).((((((((((((((((.(((((....)))))..)))))......)).)))))))))(((-----.(((((.((((((....)))))).).)))).)))----- ( -42.10) >consensus GCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC_____AGCUCAGGCAUUAUUAAUAAUGCGGUGGGUAGAG_____ ((((((.(((...(((..(((((((((((((((.(((((....)))))..)))))......))).)))))))(((......)))....)))...))).))))))................ (-34.64 = -35.54 + 0.90)



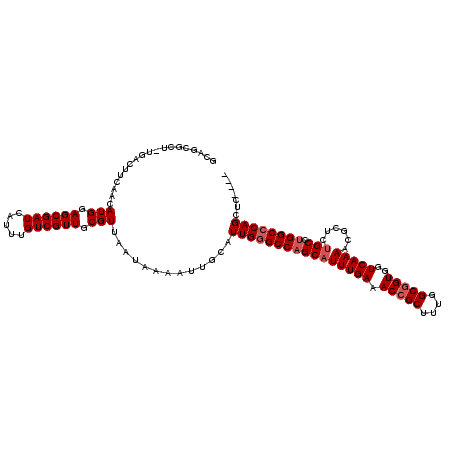
| Location | 20,610,757 – 20,610,877 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.26 |
| Mean single sequence MFE | -39.30 |
| Consensus MFE | -31.48 |
| Energy contribution | -32.28 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.880792 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20610757 120 - 23771897 GCAGCGCUAUGACUUCAACAUGGAGCGAUCAUUUGUCGUUGCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUCAGCU (((((((.((((((((.....))))...))))..).))))))..........(((..((((((((((((((((.(((((....)))))..)))))......)).)))))))))..))).. ( -43.00) >DroSec_CAF1 55590 115 - 1 GCAGCGCU-UGACUUCAACAUGGAGCGAUCAUUUGUCGUUGCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC---- (((((((.-(((((((.....))))...)))...).))))))............((..(((((((((((((((.(((((....)))))..)))))......)).))))))))))..---- ( -41.30) >DroAna_CAF1 77430 100 - 1 -------------CUCAACAUGGAGCGAUCAUUUGUCGUUGCGUUAAUAAAAUUGCAUUGGGCCUGCAUUUGAAACGGCUUUGGCGGUGGUCAAAACGCCCGGCUUGGCGCAG------- -------------......(((.((((((.....)))))).))).........(((.(..((((.((.(((((.((.((....)).))..)))))..))..))))..).))).------- ( -29.80) >DroSim_CAF1 50482 115 - 1 GCAGCGCU-UGACUUCAACAUGGAGCGAUCAUUUGUCGUUGCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC---- (((((((.-(((((((.....))))...)))...).))))))............((..(((((((((((((((.(((((....)))))..)))))......)).))))))))))..---- ( -41.30) >DroEre_CAF1 48125 115 - 1 GCAGAGCU-UGGCUUCAACAUGGAGCGAUCAUUUGUCGUUGCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC---- ...(((((-.((((.....(((.((((((.....)))))).)))...............((((.(((.(((((.(((((....)))))..)))))..)))..)))))))).)))))---- ( -41.10) >consensus GCAGCGCU_UGACUUCAACAUGGAGCGAUCAUUUGUCGUUGCGUUAAUAAAAUUGCAUUGGGCCAGCAUUUGAAACCGCUUUGGCGGUGGUCAAAACGCUCUGCCUGGCCCAGCUC____ ...................(((.((((((.....)))))).))).............((((((((((((((((.(((((....)))))..)))))......))).))))))))....... (-31.48 = -32.28 + 0.80)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:54:41 2006