
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 20,584,009 – 20,584,205 |
| Length | 196 |
| Max. P | 0.958626 |

| Location | 20,584,009 – 20,584,125 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.59 |
| Mean single sequence MFE | -27.09 |
| Consensus MFE | -20.77 |
| Energy contribution | -21.97 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.49 |
| SVM RNA-class probability | 0.958626 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20584009 116 - 23771897 UUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGAGGCGAUUUAAUUUGAA----AGGCUUCUCUAAUGAAUAUU .(((...(((((((((((....)))))))))))...)))(((((((((.((((........)))).)))))))))..((((((..(((......))----).))))))............ ( -29.50) >DroSec_CAF1 28721 116 - 1 UUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGAGGCGAUUAAAAUUGAA----AAGCUUCUCUAAUGAAUAUU .(((...(((((((((((....)))))))))))...)))(((((((((.((((........)))).)))))))))..((((((.............----..))))))............ ( -29.36) >DroAna_CAF1 50092 112 - 1 UUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCUCAAUAAUCAUAUAUAUUC-ACAAUU-UGGACUG------AAUUUCACUUGACAUUCAAGCUUUUCUAAUGAUUAUG ((((...(((((((((((....)))))))))))...))))......((((((((.....(((-(.....-)))).((------(((.((....)).)))))..........)))))))). ( -20.70) >DroSim_CAF1 22254 116 - 1 UUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGAGGCGAUUAAAAUUGAA----AAGCUUCUCUAAUGAAUAUU .(((...(((((((((((....)))))))))))...)))(((((((((.((((........)))).)))))))))..((((((.............----..))))))............ ( -29.36) >DroEre_CAF1 21845 116 - 1 UUGUCAUAAAGUUGAUGACUUGUUAUCGGUUUUUAAACAGACCGCAAUAGUCGUAUACAUCCGACGAUUGUGGGUUUGGGGGUAAUUAAUAAUGGA----ACGCCUUUCUAAUAAUAACU ..(((((.......)))))(((((((..(........(((((((((((.((((........)))).))))).))))))((.((.............----)).))...)..))))))).. ( -26.52) >consensus UUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGAGGCGAUUAAAAUUGAA____AAGCUUCUCUAAUGAAUAUU .(((...(((((((((((....)))))))))))...)))(((((((((.((((........)))).)))))))))..((((((...................))))))............ (-20.77 = -21.97 + 1.20)



| Location | 20,584,045 – 20,584,165 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.67 |
| Mean single sequence MFE | -33.94 |
| Consensus MFE | -28.12 |
| Energy contribution | -28.32 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.96 |
| SVM RNA-class probability | 0.889360 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20584045 120 - 23771897 GGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGAAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGA ((((.(((....))).)))).(((.(((((........))))).)))(((((((((((....))))))))))).....((((((((((.((((........)))).)))))))))).... ( -33.80) >DroSec_CAF1 28757 120 - 1 GGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGGAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGA (((((.(....(((((((...((((.(((((..(((....((((...(((((((((((....)))))))))))...))))..)))..)))))))))...)))))))..).)))))..... ( -34.00) >DroAna_CAF1 50129 115 - 1 GGGCUGAUAGGCAUCGGCUCGGUGUCGAUUGGACCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCUCAAUAAUCAUAUAUAUUC-ACAAUU-UGGACUG--- .(((..((.(((((((...))))))).))..).)).((((((((...(((((((((((....)))))))))))......(....).................-))))))-)).....--- ( -27.30) >DroSim_CAF1 22290 120 - 1 GGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGGAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGA (((((.(....(((((((...((((.(((((..(((....((((...(((((((((((....)))))))))))...))))..)))..)))))))))...)))))))..).)))))..... ( -34.00) >DroEre_CAF1 21881 119 - 1 GGGCAGAUAGGCAUCG-GUUGGUGUCGAUUGGAGCGCAGGUUGUCAUAAAGUUGAUGACUUGUUAUCGGUUUUUAAACAGACCGCAAUAGUCGUAUACAUCCGACGAUUGUGGGUUUGGG ...(((((..((((((-(...((((((((((..((((((((..(((......)))..))))))....(((((......)))))))..)))))).))))..))))....)))..))))).. ( -40.60) >consensus GGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGGAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAGACCGCAAUAAUCAUAUAUAUCUGAUGAUUGUGGUUUCAGA ((((.(((....))).)))).(((.(((((........))))).)))(((((((((((....))))))))))).....((((((((((.((((........)))).)))))))))).... (-28.12 = -28.32 + 0.20)

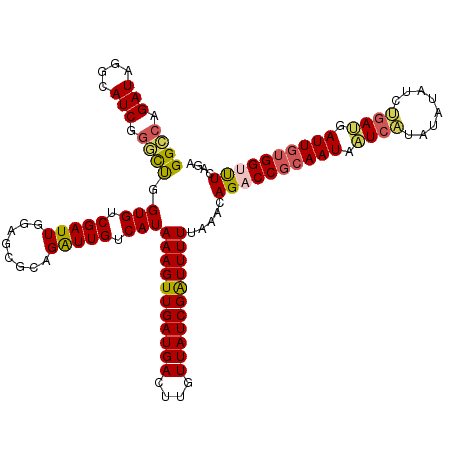
| Location | 20,584,085 – 20,584,205 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.50 |
| Mean single sequence MFE | -33.52 |
| Consensus MFE | -27.46 |
| Energy contribution | -27.22 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.33 |
| SVM RNA-class probability | 0.942423 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20584085 120 - 23771897 GGCAGAAUUAUGAUUUACUAUAAUGAAGCGCUAUAUAGUCGGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGAAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAG (((((.((((((......))))))...(((((...(((((((((((...(....)...)))))..)))))).)))))...)))))..(((((((((((....)))))))))))....... ( -32.80) >DroSec_CAF1 28797 120 - 1 GGCAAAAUUAUGAUUUACUAUAAUGAAGCGCUAUAUUGUCGGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGGAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAG (((((.((((((......))))))...(((((..((((((((((.(((....))).))))))...))))...)))))...)))))..(((((((((((....)))))))))))....... ( -34.60) >DroAna_CAF1 50164 120 - 1 GACAGAAUUAUGAUUUACUAUAAUGAAGCGUUAUAUAGUCGGGCUGAUAGGCAUCGGCUCGGUGUCGAUUGGACCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAG (((((.((((((......))))))...(((........((((((((((....)))))))))).(((.....))))))...)))))..(((((((((((....)))))))))))....... ( -32.30) >DroSim_CAF1 22330 120 - 1 GGCAGAAUUAUGAUUUACUAUAAUGAAGCGCUAUAUUGUCGGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGGAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAG (((((.((((((......))))))...(((((..((((((((((.(((....))).))))))...))))...)))))...)))))..(((((((((((....)))))))))))....... ( -34.30) >DroEre_CAF1 21921 119 - 1 GGAGGAAUUAUGAUUGACUAUAAUGGAGCGCUAUAUAGUCGGGCAGAUAGGCAUCG-GUUGGUGUCGAUUGGAGCGCAGGUUGUCAUAAAGUUGAUGACUUGUUAUCGGUUUUUAAACAG ..(((((((.((.(((((((((.(((....)))))))))))).))((((.((..((-((((....))))))..))((((((..(((......)))..)))))))))))))))))...... ( -33.60) >consensus GGCAGAAUUAUGAUUUACUAUAAUGAAGCGCUAUAUAGUCGGCCAGAUAGGCAUCGGGCUGGUGUCGAUUGGAGCGCAGAUUGUCAUAAAGUUGAUGACUUGUUAUCGAUUUUUAAACAG (((((.((((((......))))))...(((((.....(((.....))).((((((.....))))))......)))))...)))))..(((((((((((....)))))))))))....... (-27.46 = -27.22 + -0.24)

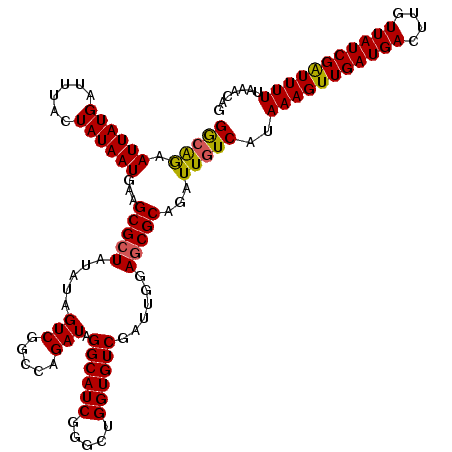

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:53:44 2006