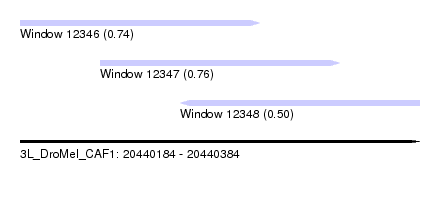
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 20,440,184 – 20,440,384 |
| Length | 200 |
| Max. P | 0.764383 |

| Location | 20,440,184 – 20,440,304 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.50 |
| Mean single sequence MFE | -38.19 |
| Consensus MFE | -34.18 |
| Energy contribution | -34.50 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.27 |
| Structure conservation index | 0.90 |
| SVM decision value | 0.46 |
| SVM RNA-class probability | 0.744204 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20440184 120 + 23771897 AUGAUGACCAUGGAUAACUGGUGGUUCUUUGUGAUAAGUUCGAUCAGUGGAGUAUUGGGUGUUAGCUUCUCAGUGGCUUUCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAGCGCUCU .((((..(((......((((((.(..(((......)))..).)))))).......)))..))))(((..((.((((..((((((.....)))))).....))))....))..)))..... ( -35.62) >DroSec_CAF1 49885 120 + 1 AUGAUGACCAUGGAUAACUGGUGGUUCUUUGUGAUCAGUUCGCUCAGUGGAGUCUUGGGCGUUAGCUUCUCAGUGGUUUCCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAUCGCUCU ..(((..((((.((.(((((((.(.......).)))))))...)).)))).)))..(((((....(((((..(((((.((((((.....))))))....)))))...)))))..))))). ( -43.90) >DroSim_CAF1 51640 120 + 1 AUGAUGACCAUGGAUAACUGGUGGUUCUUUGUGAUUAGUUCGCUCAGUGGAGUUUUGGGCGUUAGCUUCUCAGUGGUUUUCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAGCGCUCU ......((((.(.....))))).(((((((.(((..(((((((((((.......)))))))..))))..)))(((((.((((((.....))))))....)))))....)))))))..... ( -40.10) >DroEre_CAF1 44895 120 + 1 AUGAUGACCAUCGGAAACUGGUGGUUCUUUGUGAUCAGUUCGAUCAGUGGAGUUUUGGGCGUUAGCUUCUCCGUUGUUUUUGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAACGCUCU .....((((((((....).)))))))(((..(((((.....)))).)..)))....((((((...(((((..((.((.((((((.....))))))....)).))...))))).)))))). ( -37.30) >DroYak_CAF1 69812 120 + 1 AUGAUGACCAUCGAUAACUGGUGGUUCUUUGUGAUCAGUUCGACUAGUGGAGUUUUGGGCGUUAGCUUCUCGGUGGUUUUUGCCUAUGUAGCGGAUGUCACCACAAAGGACGAGCGCUCU .((((((((((((.....)))))))).......))))(((((.((..(((((....((((....)))))))(((((..(((((.......)))))..))))).))..)).)))))..... ( -34.01) >consensus AUGAUGACCAUGGAUAACUGGUGGUUCUUUGUGAUCAGUUCGAUCAGUGGAGUUUUGGGCGUUAGCUUCUCAGUGGUUUUCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAGCGCUCU .......((((.((.(((((((.(.......).)))))))...)).))))......((((((...(((((..(((((.((((((.....))))))....)))))...))))).)))))). (-34.18 = -34.50 + 0.32)



| Location | 20,440,224 – 20,440,344 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.42 |
| Mean single sequence MFE | -41.18 |
| Consensus MFE | -37.60 |
| Energy contribution | -37.64 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.764383 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20440224 120 + 23771897 CGAUCAGUGGAGUAUUGGGUGUUAGCUUCUCAGUGGCUUUCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAGCGCUCUAGAUCCUAUGAACUUGUAUCGGCCACCUUUGCUGCCAGCU ....(((..(((...((((((..((((.......))))..)))))).(((((.((((......((.(((((((....)))...)))).))......)))).))))))))..)))...... ( -39.50) >DroSec_CAF1 49925 120 + 1 CGCUCAGUGGAGUCUUGGGCGUUAGCUUCUCAGUGGUUUCCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAUCGCUCUAGAUCCUAUGGACUGGUAUCGGCCACCUUUGCUGCCAGUU ......((((..(((.(((((....(((((..(((((.((((((.....))))))....)))))...)))))..))))).)))..)))).((((((((.(((......))).)))))))) ( -49.40) >DroSim_CAF1 51680 120 + 1 CGCUCAGUGGAGUUUUGGGCGUUAGCUUCUCAGUGGUUUUCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAGCGCUCUAGAUCCUAUGGACUGGUAUCGGCCACCUUUGCUGCCAGUU ......((((..(((.((((((...(((((..(((((.((((((.....))))))....)))))...))))).)))))).)))..)))).((((((((.(((......))).)))))))) ( -45.30) >DroEre_CAF1 44935 120 + 1 CGAUCAGUGGAGUUUUGGGCGUUAGCUUCUCCGUUGUUUUUGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAACGCUCUAGAUCCUAUGGAUUGGUAUCGGCCACUUUCGCAGCCAGUA (((((.((((..(((.((((((...(((((..((.((.((((((.....))))))....)).))...))))).)))))).)))..)))).))))).....(((..(....)..))).... ( -35.90) >DroYak_CAF1 69852 120 + 1 CGACUAGUGGAGUUUUGGGCGUUAGCUUCUCGGUGGUUUUUGCCUAUGUAGCGGAUGUCACCACAAAGGACGAGCGCUCUAGAUCCUAUGGAUUGGUAUCGGCCACCUUUGCUGCCAGUU ......((((..(((.(((((((...((((..(((((.(((((.......)))))....)))))..))))..))))))).)))..)))).((((((((.(((......))).)))))))) ( -35.80) >consensus CGAUCAGUGGAGUUUUGGGCGUUAGCUUCUCAGUGGUUUUCGCCUAUGUGGCGGAUGUUACCACAAAGGAAGAGCGCUCUAGAUCCUAUGGACUGGUAUCGGCCACCUUUGCUGCCAGUU ...(((..(((.....((((((...(((((..(((((.((((((.....))))))....)))))...))))).))))))....)))..)))(((((((.(((......))).))))))). (-37.60 = -37.64 + 0.04)


| Location | 20,440,264 – 20,440,384 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.61 |
| Mean single sequence MFE | -36.55 |
| Consensus MFE | -23.33 |
| Energy contribution | -24.45 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.21 |
| Structure conservation index | 0.64 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20440264 120 - 23771897 CGAUCCAUUAUGAGAUUACCCAUUGCUGGGGCGAUCACCAAGCUGGCAGCAAAGGUGGCCGAUACAAGUUCAUAGGAUCUAGAGCGCUCUUCCUUUGUGGUAACAUCCGCCACAUAGGCG .(((((..((((((....((((....)))).((..((((..((.....))...))))..)).......)))))))))))............(((.((((((.......)))))).))).. ( -37.10) >DroPse_CAF1 40018 94 - 1 AUAUUCAUCAGUAUGUUACCCAAGGCCGGAGAUACGACCAGGCUGGCGGCAAAGGUGGCGGACAACAUCCCGUGCGACCGCGAACGCUCCUCUG-------------------------- ...(((.......(((..((...((((((........)).))))))..)))...((((((.((........)).)).)))))))..........-------------------------- ( -22.90) >DroSec_CAF1 49965 120 - 1 AGAUCCAUUAUGAGAUUACCCAUUGCCGGGGCGAUCACCAAACUGGCAGCAAAGGUGGCCGAUACCAGUCCAUAGGAUCUAGAGCGAUCUUCCUUUGUGGUAACAUCCGCCACAUAGGCG ((((((..((((.((((.....(((((((((......))...)))))))....((((.....))))))))))))))))))...........(((.((((((.......)))))).))).. ( -36.20) >DroSim_CAF1 51720 120 - 1 AGAUCCAUUAUGAGAUUACCCAUUGCCGGAGCGAUCACCAAACUGGCAGCAAAGGUGGCCGAUACCAGUCCAUAGGAUCUAGAGCGCUCUUCCUUUGUGGUAACAUCCGCCACAUAGGCG ((((((..((((.((((.....(((((((.............)))))))....((((.....))))))))))))))))))...........(((.((((((.......)))))).))).. ( -34.72) >DroEre_CAF1 44975 120 - 1 AGAUCCAUUAUGAGAUUGCCCAAAGCUGGGGCGAUCACCAUACUGGCUGCGAAAGUGGCCGAUACCAAUCCAUAGGAUCUAGAGCGUUCUUCCUUUGUGGUAACAUCCGCCACAUAGGCA ((((((..((((.((((((((.......)))))))).......((((..(....)..)))).........))))))))))...........(((.((((((.......)))))).))).. ( -46.60) >DroYak_CAF1 69892 120 - 1 AGAUCCAUGAUGAGAUUGCCCAAAAAUGGGGCGAUCACCAAACUGGCAGCAAAGGUGGCCGAUACCAAUCCAUAGGAUCUAGAGCGCUCGUCCUUUGUGGUGACAUCCGCUACAUAGGCA (((((((((....((((((((.......)))))))).......((((.((....)).)))).........))).)))))).........(.(((.(((((((.....))))))).)))). ( -41.80) >consensus AGAUCCAUUAUGAGAUUACCCAAUGCCGGGGCGAUCACCAAACUGGCAGCAAAGGUGGCCGAUACCAGUCCAUAGGAUCUAGAGCGCUCUUCCUUUGUGGUAACAUCCGCCACAUAGGCG .(((((.......((((.(((.......))).)))).......((((.((....)).)))).............)))))............(((.((((((.......)))))).))).. (-23.33 = -24.45 + 1.12)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:51:17 2006