
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 20,328,163 – 20,328,300 |
| Length | 137 |
| Max. P | 0.883781 |

| Location | 20,328,163 – 20,328,262 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 79.30 |
| Mean single sequence MFE | -24.88 |
| Consensus MFE | -15.98 |
| Energy contribution | -16.45 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.883781 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20328163 99 + 23771897 ACACGAAAUAGUCAGCAACUGCACAUUUUGGGGACAAAAGUCUAAGAUAUGUAUACGA-UUUACGCACAUAAAUAUGCGGC-GUGCGUACGACCACGUACU .((((....((((.((....))((((.((..((((....))))..)).))))....))-))..((((........)))).)-))).(((((....))))). ( -24.30) >DroSec_CAF1 23807 95 + 1 ACACGAAAUAGUCCGCAACUGCACAUUUUGGGGACAAAAGUCUAAAAU----AUACGA-UUUACGCACAUAAAUAUGCGGC-GUGCGUACGCACACGUACU ..(((.....(.(((((........(((((....)))))((.((((..----......-)))).)).........))))))-((((....)))).)))... ( -22.60) >DroSim_CAF1 23987 95 + 1 ACACGAAAUAGUCCGCAACUGCACAUUUUGGGGACAAAAGUCUAAAAU----AUACGA-UUUACGCACAUAAAUGUGCGGC-GUGCGUACGACCACGUACU .((((.....((((.(((.........))).))))..(((((......----....))-))).((((((....)))))).)-))).(((((....))))). ( -26.70) >DroEre_CAF1 24048 96 + 1 ACACGAAAUAGACAGGAACUGCACAUUUUGGGGACAAAAGUCUGAAGU----GUACAGUUUUACGCACAUAAAUAUGCGGC-GUGCGUACGAGCACGUACU ..............(((((((.(((((((..((((....)))))))))----)).))))))).((((........))))((-((((......))))))... ( -30.00) >DroYak_CAF1 32465 96 + 1 AUUCGAAAUAGACAGGAACUGCACAUUUUGGGGACAAAAGUGUGAAGU----AUACAAUUUUACGCACAUAAAUAUGCGGC-GUGCGUACGACCACGGUCU .........((((.....(((((..(((((....)))))(((((((((----.....))))))))).........)))))(-(((.(.....))))))))) ( -26.40) >DroPer_CAF1 25683 88 + 1 ---AUAAAUAAUGAGGAACUGAAAAUUUUGGGGACAGUGGUCCCGAAU----AUAUAU-AUU--GUAUAUAUAAUUGCGUCUGUGCGUACA---GUGCACC ---...........((.((((......((.(((((....))))).))(----((((((-(..--.))))))))..(((((....)))))))---))...)) ( -19.30) >consensus ACACGAAAUAGUCAGCAACUGCACAUUUUGGGGACAAAAGUCUAAAAU____AUACAA_UUUACGCACAUAAAUAUGCGGC_GUGCGUACGACCACGUACU ....................((((.(((((....)))))........................((((........))))...))))(((((....))))). (-15.98 = -16.45 + 0.47)


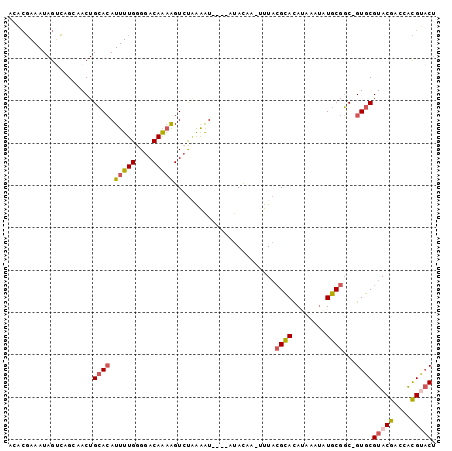
| Location | 20,328,163 – 20,328,262 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 79.30 |
| Mean single sequence MFE | -24.95 |
| Consensus MFE | -14.97 |
| Energy contribution | -14.95 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.844147 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20328163 99 - 23771897 AGUACGUGGUCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAA-UCGUAUACAUAUCUUAGACUUUUGUCCCCAAAAUGUGCAGUUGCUGACUAUUUCGUGU .(((((....)))))((((-(..((((((((.((((((....-.)))))).........(((....)))....))))))))((((...))))....))))) ( -25.90) >DroSec_CAF1 23807 95 - 1 AGUACGUGUGCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAA-UCGUAU----AUUUUAGACUUUUGUCCCCAAAAUGUGCAGUUGCGGACUAUUUCGUGU .(((((....)))))((((-((((((.((((((....)))))-).((((----(((((.(((....)))...)))))))))...))))).......))))) ( -28.21) >DroSim_CAF1 23987 95 - 1 AGUACGUGGUCGUACGCAC-GCCGCACAUUUAUGUGCGUAAA-UCGUAU----AUUUUAGACUUUUGUCCCCAAAAUGUGCAGUUGCGGACUAUUUCGUGU ..((((..((.((.((((.-((.(((((((((((((((....-.)))))----))....(((....)))....)))))))).)))))).)).))..)))). ( -29.70) >DroEre_CAF1 24048 96 - 1 AGUACGUGCUCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAAACUGUAC----ACUUCAGACUUUUGUCCCCAAAAUGUGCAGUUCCUGUCUAUUUCGUGU .(((((....)))))((((-(.((((((....))))))...((((((((----(.....(((....))).......)))))))))...........))))) ( -28.30) >DroYak_CAF1 32465 96 - 1 AGACCGUGGUCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAAAUUGUAU----ACUUCACACUUUUGUCCCCAAAAUGUGCAGUUCCUGUCUAUUUCGAAU ((((.(..((..(((((((-(...........))))))))..))..)..----(((.((((.(((((....))))))))).)))....))))......... ( -21.80) >DroPer_CAF1 25683 88 - 1 GGUGCAC---UGUACGCACAGACGCAAUUAUAUAUAC--AAU-AUAUAU----AUUCGGGACCACUGUCCCCAAAAUUUUCAGUUCCUCAUUAUUUAU--- .((((..---.....)))).(((.....((((((((.--..)-))))))----)...(((((....)))))...........))).............--- ( -15.80) >consensus AGUACGUGGUCGUACGCAC_GCCGCAUAUUUAUGUGCGUAAA_UCGUAU____AUUUUAGACUUUUGUCCCCAAAAUGUGCAGUUCCUGACUAUUUCGUGU .(((((....)))))........((((((((..(((((......)))))..........(((....)))....)))))))).................... (-14.97 = -14.95 + -0.02)



| Location | 20,328,201 – 20,328,300 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 80.53 |
| Mean single sequence MFE | -24.27 |
| Consensus MFE | -14.45 |
| Energy contribution | -15.65 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.726615 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20328201 99 - 23771897 CAGUGUUCUAAAUAACUUUAGAUUGCUGCAAUUGUAUGAGUACGUGGUCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAA-UCGUAUACAUAUCUUAGACU ((((..((((((....))))))..))))....((((((((((((....)))))..((-((.((((....))))))))...-)))))))............. ( -25.20) >DroSec_CAF1 23845 95 - 1 CAGUGUUCUAAAUAACUUUAGAUUGCUGCAAUUGUAUGAGUACGUGUGCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAA-UCGUAU----AUUUUAGACU ((((..((((((....))))))..))))....(((((((.(((((((((....))))-)).(((((....))))))))..-))))))----)......... ( -28.10) >DroSim_CAF1 24025 95 - 1 CAGUGUUCUAAAUAACUUUAGAUUGCUGCAAUUGUAUGAGUACGUGGUCGUACGCAC-GCCGCACAUUUAUGUGCGUAAA-UCGUAU----AUUUUAGACU ((((..((((((....))))))..))))....((((((((((((....)))))....-..((((((....))))))....-))))))----)......... ( -26.40) >DroEre_CAF1 24086 96 - 1 CAGUGUUCUAAAUAGCUUUAGAUUGCUGCAAUUGUAUGAGUACGUGCUCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAAACUGUAC----ACUUCAGACU ((((..((((((....))))))..)))).....((((((((....))))))))..((-((.((((....))))))))....(((...----....)))... ( -25.50) >DroYak_CAF1 32503 96 - 1 CAGUGUUCUAAAUAACUUUAGAUUGCUGCAAUUGUAUGAGACCGUGGUCGUACGCAC-GCCGCAUAUUUAUGUGCGUAAAAUUGUAU----ACUUCACACU .(((((((((((....))))))....(((((((......(((....))).(((((((-(...........)))))))).))))))).----.....))))) ( -23.70) >DroPer_CAF1 25718 88 - 1 CUGUGUACUAAAUAACUGCAAACUUUU---AUAGUAUAGGUGCAC---UGUACGCACAGACGCAAUUAUAUAUAC--AAU-AUAUAU----AUUCGGGACC ((((((.....((((..(....)..))---)).((((((.....)---))))))))))).(.(...((((((((.--..)-))))))----)...).)... ( -16.70) >consensus CAGUGUUCUAAAUAACUUUAGAUUGCUGCAAUUGUAUGAGUACGUGGUCGUACGCAC_GCCGCAUAUUUAUGUGCGUAAA_UCGUAU____AUUUUAGACU (((((.((((((....)))))).))))).....(((((((((((....))))).......((((((....)))))).....)))))).............. (-14.45 = -15.65 + 1.20)

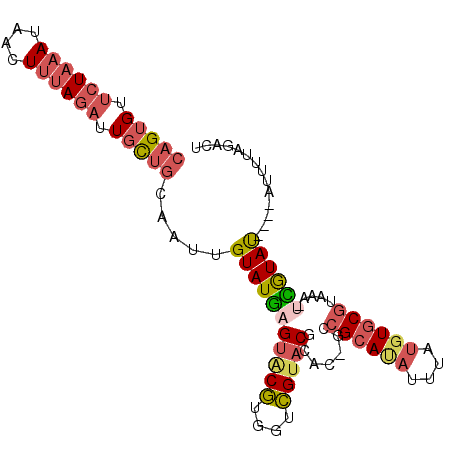
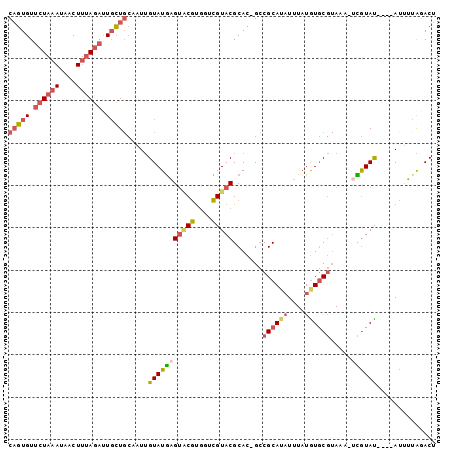
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:50:19 2006