
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 20,022,865 – 20,023,027 |
| Length | 162 |
| Max. P | 0.999948 |

| Location | 20,022,865 – 20,022,976 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 80.71 |
| Mean single sequence MFE | -37.72 |
| Consensus MFE | -27.71 |
| Energy contribution | -29.52 |
| Covariance contribution | 1.81 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.31 |
| SVM RNA-class probability | 0.681363 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
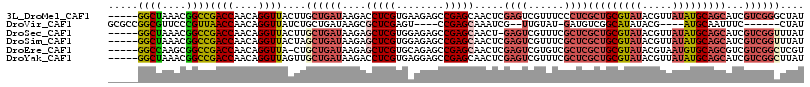
>3L_DroMel_CAF1 20022865 111 + 23771897 -----GGCUAAACGGCCGACCAACAGGUUACUUGCUGAUAAGACCUCGUGAAGAGCCGAGCAACUCGAGUCGUUUCCCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGGCUAU -----((((....))))((((....))))...........((.(((..(((.(((..((((.((....)).))))..)))((((((((((...)))))))))).)))..))))).. ( -33.90) >DroVir_CAF1 15239 99 + 1 GCGCCGGCGUUCCCGUUAACCAACAGGUUAUCUGCUGAUAAGCGCUCGAGU----CCGAGCAAAUCG--UUGUAU-GAUGUCGGCAUAUACG----AUGCAAUUUC------CUAU ..((((((((...(((....((((.(.(((((....))))).)(((((...----.))))).....)--))).))-))))))))).......----..........------.... ( -29.50) >DroSec_CAF1 14507 110 + 1 -----GGCUAAACGGCCGACCAACAGGUUACUUGCUGAUAAGAGCUCGUGGAGAGCCGAGCAACU-GAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGUUUAU -----((((....))))((((....))))....((((((..(.((((.....)))))(((((((.-.....)))..))))((((((((((...))))))))))...)))))).... ( -38.40) >DroSim_CAF1 9201 111 + 1 -----GGCUAAACGGCCGACCAACAGGUUACUAGCUGAUAAGAGCUCGUGGAGAGCCGAGCAACUCGAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGUUUAU -----((((....))))((((.((....(((.((((......)))).)))((((((.((((.((....)).)))).))))((((((((((...)))))))))).)))).))))... ( -39.60) >DroEre_CAF1 13986 110 + 1 -----GGCCAAGCGGCCGACCAACAGGUUA-CUGCUGAUAAGAGCUCGUGCAGAGCCGAGCAACUCGAGUCGUGUCGCUCGCUGCGUAUACGUAAUGUGCAGCGUCGUCGGCUCGU -----((((....))))...((.(((....-))).))......(((((.((...)))))))....(((((((...((..((((((((((.....)))))))))).)).))))))). ( -44.70) >DroYak_CAF1 16109 111 + 1 -----GGCUAAACGGCCGACCAACAGGUUAGUUGCUGAUAAGACCUCGUGAGGAGCCGAGCAACUCGAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGCUUAU -----((((....))))((((....))))....((((((.......(....)((((.((((.((....)).)))).))))((((((((((...))))))))))...)))))).... ( -40.20) >consensus _____GGCUAAACGGCCGACCAACAGGUUACUUGCUGAUAAGAGCUCGUGAAGAGCCGAGCAACUCGAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGCUUAU .....((((....))))((((....))))....((((((....(((((........))))).....((((......))))(((((((((.....)))))))))...)))))).... (-27.71 = -29.52 + 1.81)
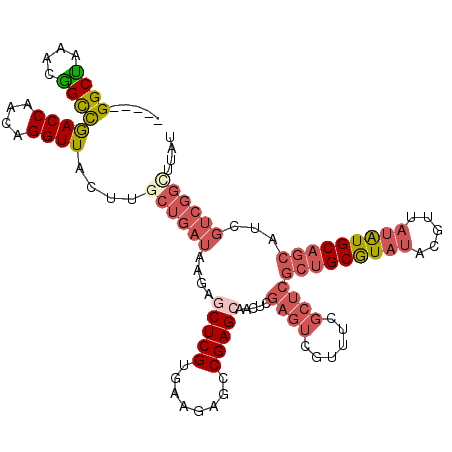

| Location | 20,022,900 – 20,023,001 |
|---|---|
| Length | 101 |
| Sequences | 5 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 91.49 |
| Mean single sequence MFE | -39.68 |
| Consensus MFE | -35.50 |
| Energy contribution | -35.54 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.89 |
| SVM decision value | 4.44 |
| SVM RNA-class probability | 0.999898 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20022900 101 + 23771897 AGACCUCGUGAAGAGCCGAGCAACUCGAGUCGUUUCCCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGGCUAUUACGUAUACGCAGCGUUUAACUCAC .((((((.....))).((((...)))).)))(((.....((((((((((((((.(((.((..(......).))))).))))))))))))))...))).... ( -35.40) >DroSec_CAF1 14542 100 + 1 AGAGCUCGUGGAGAGCCGAGCAACU-GAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGUUUAUUACGUAUACGCAGCGUUUCACUCAC .......(((((((((.((((.((.-..)).)))).))))(((((((((((((.(((.((..........)).))).)))))))))))))..))))).... ( -37.40) >DroSim_CAF1 9236 101 + 1 AGAGCUCGUGGAGAGCCGAGCAACUCGAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGUUUAUUACGUAUACGCCGCGUUUCACUCAC .(((..(((((.(((((((.......((((......))))((((((((((...))))))))))....)))))))....((....))))))).....))).. ( -35.20) >DroEre_CAF1 14020 101 + 1 AGAGCUCGUGCAGAGCCGAGCAACUCGAGUCGUGUCGCUCGCUGCGUAUACGUAAUGUGCAGCGUCGUCGGCUCGUUACGUAUACGCAGCGUUGCGCUCAC .(((((((.((...)))))(((((..((((......))))(((((((((((((((((.((..((....)))).)))))))))))))))))))))))))).. ( -52.50) >DroYak_CAF1 16144 101 + 1 AGACCUCGUGAGGAGCCGAGCAACUCGAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGCUUAUUACGUAUACGCAACGUUGCGCUCAC .......((((((((((((.......((((......))))((((((((((...))))))))))....)))))))..........((((....))))))))) ( -37.90) >consensus AGAGCUCGUGAAGAGCCGAGCAACUCGAGUCGUUUCGCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGCUUAUUACGUAUACGCAGCGUUUCACUCAC .......(((((((((.((((.((....)).)))).))))(((((((((((((.(((.((..........)).))).)))))))))))))..))))).... (-35.50 = -35.54 + 0.04)

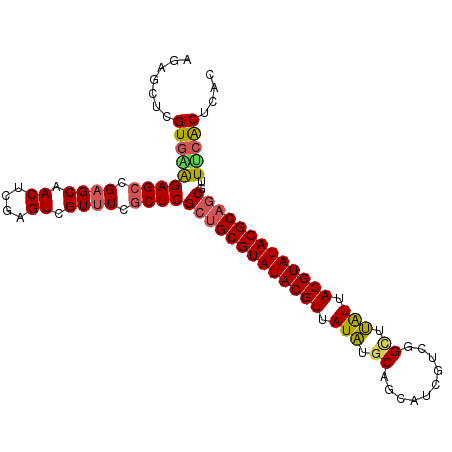
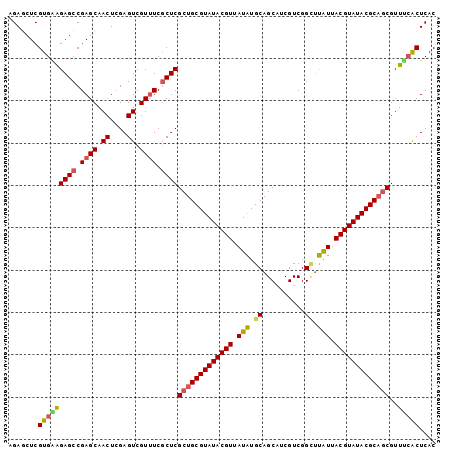
| Location | 20,022,900 – 20,023,001 |
|---|---|
| Length | 101 |
| Sequences | 5 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 91.49 |
| Mean single sequence MFE | -39.48 |
| Consensus MFE | -31.72 |
| Energy contribution | -31.80 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.09 |
| Structure conservation index | 0.80 |
| SVM decision value | 2.83 |
| SVM RNA-class probability | 0.997263 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20022900 101 - 23771897 GUGAGUUAAACGCUGCGUAUACGUAAUAGCCCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGGGAAACGACUCGAGUUGCUCGGCUCUUCACGAGGUCU .((((((...((((((((((((((.(((((...........)).))).))))))))))))))..(....))))))).....((((........)))).... ( -39.20) >DroSec_CAF1 14542 100 - 1 GUGAGUGAAACGCUGCGUAUACGUAAUAAACCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGCGAAACGACUC-AGUUGCUCGGCUCUCCACGAGCUCU ..(((..(...(((((((((((((.(((..(.(......).)..))).)))))))))))))(((((...)).)))-..)..)))(((((.....))))).. ( -36.90) >DroSim_CAF1 9236 101 - 1 GUGAGUGAAACGCGGCGUAUACGUAAUAAACCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGCGAAACGACUCGAGUUGCUCGGCUCUCCACGAGCUCU ..(((..(..(((.((((((((((.(((..(.(......).)..))).)))))))))).))(((((...)).))))..)..)))(((((.....))))).. ( -32.90) >DroEre_CAF1 14020 101 - 1 GUGAGCGCAACGCUGCGUAUACGUAACGAGCCGACGACGCUGCACAUUACGUAUACGCAGCGAGCGACACGACUCGAGUUGCUCGGCUCUGCACGAGCUCU ..((((((((((((((((((((((((.(.((.(......).)).).)))))))))))))))(((((...)).)))..)))))(((((...)).))))))). ( -46.70) >DroYak_CAF1 16144 101 - 1 GUGAGCGCAACGUUGCGUAUACGUAAUAAGCCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGCGAAACGACUCGAGUUGCUCGGCUCCUCACGAGGUCU (((((.((...(((((((((((((.(((.((.(......).)).))).)))))))))))))((((((..((...))..)))))).))..)))))....... ( -41.70) >consensus GUGAGUGAAACGCUGCGUAUACGUAAUAAACCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGCGAAACGACUCGAGUUGCUCGGCUCUCCACGAGCUCU ..((((..((((((((((((((((.(((..(.(......).)..))).)))))))))))))(((........)))..)))))))(((((.....))))).. (-31.72 = -31.80 + 0.08)

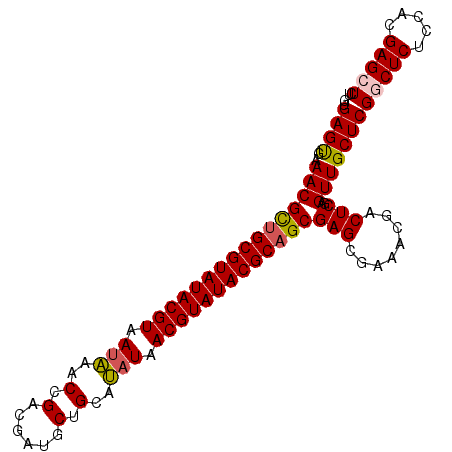

| Location | 20,022,936 – 20,023,027 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 91 |
| Reading direction | forward |
| Mean pairwise identity | 90.00 |
| Mean single sequence MFE | -40.64 |
| Consensus MFE | -34.46 |
| Energy contribution | -34.30 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.14 |
| Mean z-score | -4.55 |
| Structure conservation index | 0.85 |
| SVM decision value | 4.02 |
| SVM RNA-class probability | 0.999760 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20022936 91 + 23771897 CCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGGCUAUUACGUAUACGCAGCGUUUAACUCACUCAAUUGCAAGGGCUUUGAGUGGGCG ...((((((((((((((.(((.((..(......).))))).)))))))))))))).....(((((((((..((....)).))))))))).. ( -40.30) >DroSec_CAF1 14577 91 + 1 GCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGUUUAUUACGUAUACGCAGCGUUUCACUCACUCAAUUGCAAGGGCUUUGAGUGGGCG ...((((((((((((((.(((.((..........)).))).)))))))))))))).....(((((((((..((....)).))))))))).. ( -39.00) >DroSim_CAF1 9272 91 + 1 GCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGUUUAUUACGUAUACGCCGCGUUUCACUCACUCAAUUGCAAGGGCUUUGAGUGGGCG ...(((.((((((((((.(((.((..........)).))).)))))))))).))).....(((((((((..((....)).))))))))).. ( -34.40) >DroEre_CAF1 14056 90 + 1 GCUCGCUGCGUAUACGUAAUGUGCAGCGUCGUCGGCUCGUUACGUAUACGCAGCGUUGCGCUCACUCAAUUGCAGGG-UGUUGAGUGGACG ((.((((((((((((((((((.((..((....)))).))))))))))))))))))..))(..((((((((.......-.))))))))..). ( -48.50) >DroYak_CAF1 16180 91 + 1 GCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGCUUAUUACGUAUACGCAACGUUGCGCUCACUCAGUUGCAAGGGAUCUGAGUGGGCG ((.((.(((((((((((...(((.(((.......)))))).))))))))))).))..))((((((((((...(....)..)))))))))). ( -41.00) >consensus GCUCGCUGCGUAUACGUUAUAUGCAGCAUCGUCGGCUUAUUACGUAUACGCAGCGUUUCACUCACUCAAUUGCAAGGGCUUUGAGUGGGCG ...((((((((((((((.(((.((..........)).))).)))))))))))))).....(((((((((...........))))))))).. (-34.46 = -34.30 + -0.16)

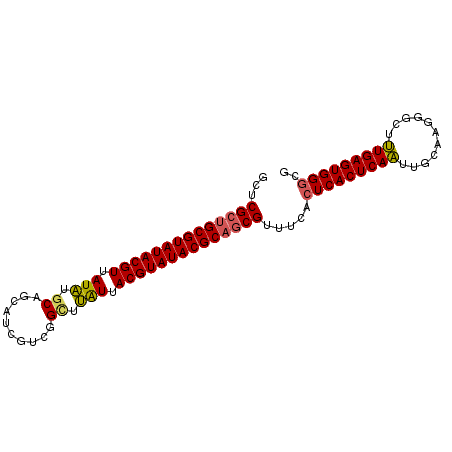
| Location | 20,022,936 – 20,023,027 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 91 |
| Reading direction | reverse |
| Mean pairwise identity | 90.00 |
| Mean single sequence MFE | -36.70 |
| Consensus MFE | -33.32 |
| Energy contribution | -32.72 |
| Covariance contribution | -0.60 |
| Combinations/Pair | 1.17 |
| Mean z-score | -4.48 |
| Structure conservation index | 0.91 |
| SVM decision value | 4.77 |
| SVM RNA-class probability | 0.999948 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 20022936 91 - 23771897 CGCCCACUCAAAGCCCUUGCAAUUGAGUGAGUUAAACGCUGCGUAUACGUAAUAGCCCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGG .((.(((((((.((....))..))))))).))....((((((((((((((.(((((...........)).))).))))))))))))))... ( -36.00) >DroSec_CAF1 14577 91 - 1 CGCCCACUCAAAGCCCUUGCAAUUGAGUGAGUGAAACGCUGCGUAUACGUAAUAAACCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGC (((.(((((((.((....))..))))))).)))...((((((((((((((.(((..(.(......).)..))).))))))))))))))... ( -36.30) >DroSim_CAF1 9272 91 - 1 CGCCCACUCAAAGCCCUUGCAAUUGAGUGAGUGAAACGCGGCGUAUACGUAAUAAACCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGC (((.(((((((.((....))..))))))).)))...(((.((((((((((.(((..(.(......).)..))).)))))))))).)))... ( -31.70) >DroEre_CAF1 14056 90 - 1 CGUCCACUCAACA-CCCUGCAAUUGAGUGAGCGCAACGCUGCGUAUACGUAACGAGCCGACGACGCUGCACAUUACGUAUACGCAGCGAGC ((..(((((((..-........)))))))..))...((((((((((((((((.(.((.(......).)).).))))))))))))))))... ( -39.90) >DroYak_CAF1 16180 91 - 1 CGCCCACUCAGAUCCCUUGCAACUGAGUGAGCGCAACGUUGCGUAUACGUAAUAAGCCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGC (((.(((((((...........))))))).)))...((((((((((((((.(((.((.(......).)).))).))))))))))))))... ( -39.60) >consensus CGCCCACUCAAAGCCCUUGCAAUUGAGUGAGUGAAACGCUGCGUAUACGUAAUAAACCGACGAUGCUGCAUAUAACGUAUACGCAGCGAGC (((.(((((((...........))))))).)))...((((((((((((((.(((..(.(......).)..))).))))))))))))))... (-33.32 = -32.72 + -0.60)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:47:46 2006