
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 19,918,800 – 19,918,958 |
| Length | 158 |
| Max. P | 0.999984 |

| Location | 19,918,800 – 19,918,896 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.49 |
| Mean single sequence MFE | -43.97 |
| Consensus MFE | -26.22 |
| Energy contribution | -26.03 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.863769 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
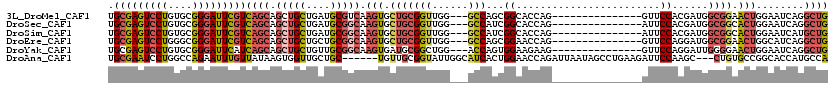
>3L_DroMel_CAF1 19918800 96 + 23771897 CGGCCAGCAGGCGAUUGAAGGACAGGUCGUACUGCA------------------CCAAUUGCUGCAACGG------CAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCG .........((((((((...(.(((......)))).------------------.))))))))(((.(((------((((((((((((((....)))))))....)))))))))).))). ( -44.80) >DroSec_CAF1 54548 96 + 1 CGGACAGCAGGCGGUUGAAGGACAGGUCGUACUGCA------------------CCAAUUGCUGCAGCGG------CAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCG ......(((((((((((...(.(((......)))).------------------.)))))))).((((((------(.((((((((((((....)))))))).)))).))))))).))). ( -47.40) >DroSim_CAF1 38873 96 + 1 CGGACAGCAGGCGGUUGAAGGAGAGGUCGUACUGCA------------------CCAAUUGCUGCAGCGG------CAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCG ......(((((((((((..(.((........)).).------------------.)))))))).((((((------(.((((((((((((....)))))))).)))).))))))).))). ( -45.30) >DroEre_CAF1 53883 96 + 1 CGGCCAGCAGGCGGUUGAAGGACAGGUCAUACUGCA------------------CCAGUUGCUGCAUCGG------CAGCUGCGAGUCCUGGGCGGGAUUCGUCAGCAGCUGCUGCUGCG ..((.(((((.((((((...(((.((((.....).)------------------)).((.(((((....)------)))).))(((((((....))))))))))..))))))))))))). ( -45.00) >DroYak_CAF1 61697 96 + 1 CGGUCAGCAGGCGGUUGAAGGACAGGUCAUACUGCA------------------CCAGUUGCUGCAUCGG------CAGCUGCGAGUCCUGUGCGGGAUUCAUCAGCAGCUGCUGUUGCG .((.((((..((((((((........))).))))).------------------...))))))(((.(((------((((((((((((((....)))))))....)))))))))).))). ( -43.40) >DroAna_CAF1 45277 119 + 1 CAGUGAGGAGGCGGGAAAAGGACAGGUCACAGGGCAGACGUAGGAAUUGGGACGCCUGCUGCUGCUGCGGCGACAACUGCUGCGAAUCCUGGCCAGAAUUUGUUAUAAGUGGUUGCUGC- ((((.((.(((((....((...((.(((........))).).)...))....))))).))))))..((((((((.(((...((((((.((....)).))))))....))).))))))))- ( -37.90) >consensus CGGACAGCAGGCGGUUGAAGGACAGGUCAUACUGCA__________________CCAAUUGCUGCAGCGG______CAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCG (((.((((.((((((((......................................))))))))...............((((((((((((....)))))))).)))).)))))))..... (-26.22 = -26.03 + -0.19)
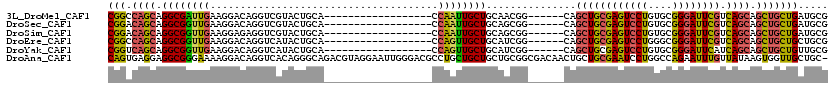
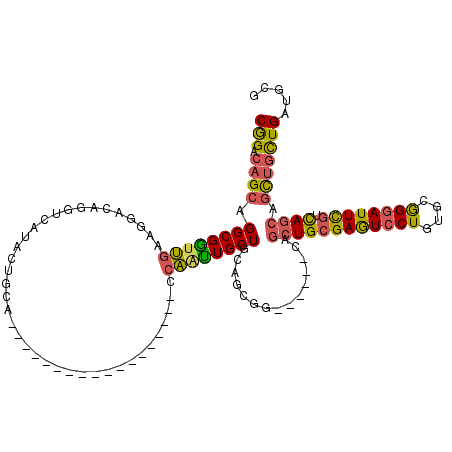
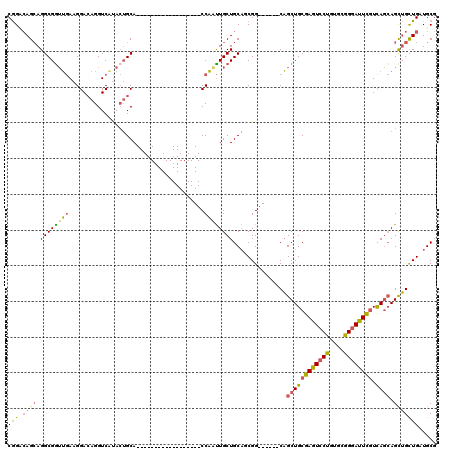
| Location | 19,918,836 – 19,918,927 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 91 |
| Reading direction | forward |
| Mean pairwise identity | 92.42 |
| Mean single sequence MFE | -49.86 |
| Consensus MFE | -46.52 |
| Energy contribution | -47.76 |
| Covariance contribution | 1.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -4.05 |
| Structure conservation index | 0.93 |
| SVM decision value | 5.36 |
| SVM RNA-class probability | 0.999984 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
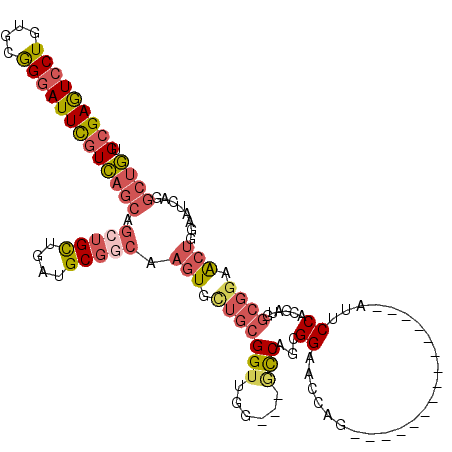
>3L_DroMel_CAF1 19918836 91 + 23771897 CCAAUUGCUGCAACGGCAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGUCAAGUGCUGCGGUUGGGCCAGCGGCACCAG ....(((.((((.(((((((((((((((((....)))))))....)))))))))).)))).)))(((((((((.....)).)))))))... ( -48.70) >DroSec_CAF1 54584 91 + 1 CCAAUUGCUGCAGCGGCAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGGCAAGUGCUGCGGUUGGGCCAUCGGCACCAG ....((((((((.(((((((((((((((((....)))))))....)))))))))).))))))))((((((.((.....))..))))))... ( -51.10) >DroSim_CAF1 38909 91 + 1 CCAAUUGCUGCAGCGGCAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGGCAAGUGCUGCGGUUGGGCCAUCGGCACCAG ....((((((((.(((((((((((((((((....)))))))....)))))))))).))))))))((((((.((.....))..))))))... ( -51.10) >DroEre_CAF1 53919 91 + 1 CCAGUUGCUGCAUCGGCAGCUGCGAGUCCUGGGCGGGAUUCGUCAGCAGCUGCUGCUGCGGCAAGUGCUGCGGUUGGGCCAGCGGAACCAG .((.((((((((.(((((((((((((((((....)))))))....)))))))))).)))))))).))((((((.....)).))))...... ( -50.50) >DroYak_CAF1 61733 91 + 1 CCAGUUGCUGCAUCGGCAGCUGCGAGUCCUGUGCGGGAUUCAUCAGCAGCUGCUGUUGCGGCAAGUGAUGCGGCUGGACCAGUGGAAGAAG .((.((((((((.(((((((((((((((((....)))))))....)))))))))).)))))))).))...(.((((...)))).)...... ( -47.90) >consensus CCAAUUGCUGCAGCGGCAGCUGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGGCAAGUGCUGCGGUUGGGCCAGCGGCACCAG ....((((((((.(((((((((((((((((....)))))))....)))))))))).))))))))(((((((((.....)).)))))))... (-46.52 = -47.76 + 1.24)

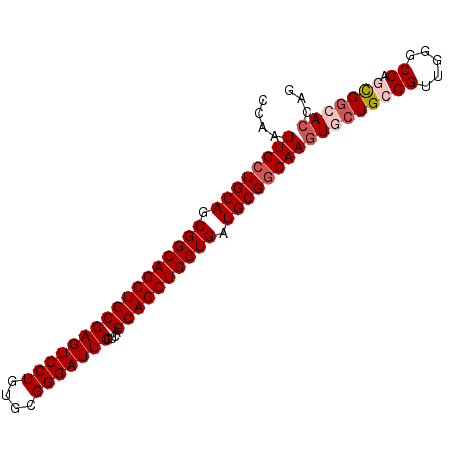
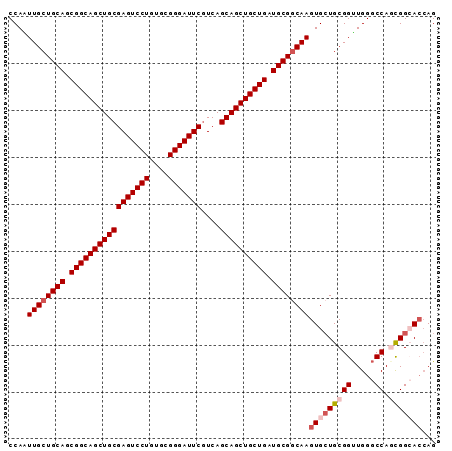
| Location | 19,918,856 – 19,918,958 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.99 |
| Mean single sequence MFE | -43.18 |
| Consensus MFE | -22.49 |
| Energy contribution | -23.83 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.538715 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19918856 102 + 23771897 UGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGUCAAGUGCUGCGGUUGG---GCCAGCGGCACCAG---------------GUUCCACGAUGGCGGAACUGGAAUCAGGCUG .(((((((((....)))))))((((((....))))))))(((..(((((((((....---.)).)))))))..(---------------(((((.(....).)))))).......))).. ( -44.80) >DroSec_CAF1 54604 102 + 1 UGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGGCAAGUGCUGCGGUUGG---GCCAUCGGCACCAG---------------AUUCCACGAUGGCGGCACUGGAAUCAGGCUG .(((((((((....)))))))))((((.(((((....))))).((((((((.(((((---(..(((.......)---------------)).)).)))).))))))))........)))) ( -47.80) >DroSim_CAF1 38929 102 + 1 UGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGGCAAGUGCUGCGGUUGG---GCCAUCGGCACCAG---------------AUUCCACGAUGGCGGCACUGGAAUCAUGCUG .(((((((((....)))))))))((((((((((....))))).((((((((.(((((---(..(((.......)---------------)).)).)))).)))))))).......))))) ( -48.80) >DroEre_CAF1 53939 102 + 1 UGCGAGUCCUGGGCGGGAUUCGUCAGCAGCUGCUGCUGCGGCAAGUGCUGCGGUUGG---GCCAGCGGAACCAG---------------GUUCCAGGAUGGCGGAACUGGCAUCAGGCUG .(((((((((....)))))))))((((.((((((((((((((....))))))))..)---).))))(((((...---------------)))))..((((.((....)).))))..)))) ( -48.20) >DroYak_CAF1 61753 102 + 1 UGCGAGUCCUGUGCGGGAUUCAUCAGCAGCUGCUGUUGCGGCAAGUGAUGCGGCUGG---ACCAGUGGAAGAAG---------------GUUCCAGGAUUGGGGAACUGGAAUCAGGCUG ...(((((((....)))))))..((((..(..(((.(.((((.(....)...)))).---).)))..).....(---------------(((((((..........))))))))..)))) ( -38.60) >DroAna_CAF1 45357 111 + 1 UGCGAAUCCUGGCCAGAAUUUGUUAUAAGUGGUUGCUGC------UGUUGCGGUAUUGGCAUCACUGGAACCAGAUUAAUAGCCUGAAGAUUCCAAGC---CUGUGCCGGCACCAUGCCA .(((((((..(((...((((((.....((((((((((((------....)))))).....)))))).....))))))....)))....)))))...((---(......))).....)).. ( -30.90) >consensus UGCGAGUCCUGUGCGGGAUUCGUCAGCAGCUGCUGAUGCGGCAAGUGCUGCGGUUGG___GCCAGCGGAACCAG_______________AUUCCACGAUGGCGGAACUGGAAUCAGGCUG .(((((((((....)))))))))((((.(((((....))))).(((.(((((((......)))...((........................))......)))).)))........)))) (-22.49 = -23.83 + 1.34)
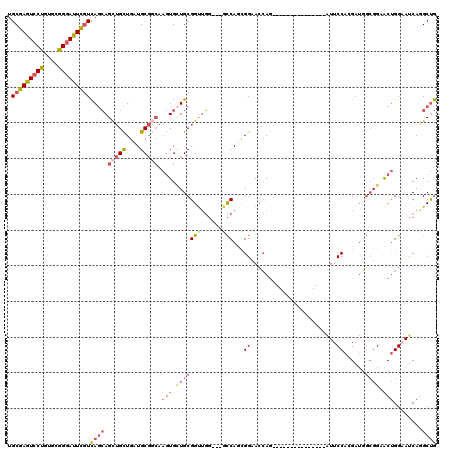
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:46:08 2006