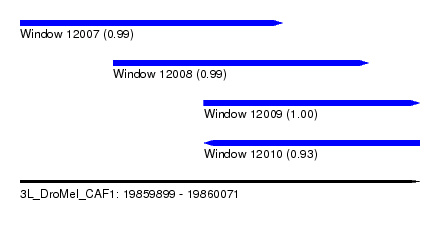
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 19,859,899 – 19,860,071 |
| Length | 172 |
| Max. P | 0.998445 |

| Location | 19,859,899 – 19,860,012 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 78.45 |
| Mean single sequence MFE | -53.27 |
| Consensus MFE | -28.46 |
| Energy contribution | -28.38 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.33 |
| Mean z-score | -3.43 |
| Structure conservation index | 0.53 |
| SVM decision value | 2.27 |
| SVM RNA-class probability | 0.991477 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19859899 113 + 23771897 GAGCUCUGGGCUGCGGCAUUGGCUUCGGCUAUUUGCAUCGCAUUCCCGUGCAGGCAGAGCCAGUGACCAGCA-GUUCCGCUGCUGUUCCUGCUGCUGCCGCCGCGGAGUCCACU ......((((((((((((.((((....))))..)))..)))....(((((..((((((((.((.((.(((((-(.....)))))).))))))).)))))..))))))))))).. ( -55.30) >DroPse_CAF1 6390 104 + 1 GUGCUCUCGGCUGUGGCAUUGGCUUCGGCUAUCUGCAUCGCAUCCCGGUGCAGGCAGUCCCCGUCC-CAGCCUCUUCGGCCUCGGUU------GCUACUGCAGCCGAGGCC--- ........(((((.(((..((((....))))(((((((((.....)))))))))........))).-))))).....((((((((((------((....))))))))))))--- ( -56.70) >DroSec_CAF1 5850 113 + 1 GAGCACUGGGCUGCGGCAUUGGCUUCGGCUAUCUGCAUCGUAUUCCCGUGCAGGCAGAGCCAGUGACCAGCA-GCUCAGCUGCUGCUCCUGCUGCUGCCUCCGCGGAGUCCUCU .......(((((((((...((((....)))).)))).........(((((.(((((((((.((.((.(((((-((...))))))).))))))).)))))).))))))))))... ( -55.90) >DroSim_CAF1 5453 113 + 1 GAGCACUGGGCUGCGGCAUUGGCUUCGGCUAUCUGCAUCGCAUUCCCGUGCAGGCAGAGCCAGUGACCAGCA-GCUCCGCUGCUGCCCCUGCUGCUGCCUCCGCGGAGUCCUCU .......(((((((((((.((((....))))..)))..)))....(((((.(((((((((.((.(..(((((-((...)))))))..)))))).)))))).))))))))))... ( -54.80) >DroYak_CAF1 5847 110 + 1 GAGCCCUGGGCUGCGGCAUUGGCUUCGGCUAUCUGCAUCGCAUUCCCGUGCAAGCAGAGCCAGUCCCUAGUA-GCACUGCGGCUGCUCCUGCUUCUGCCGUC---GAGUCCUCU .......(((((((((((..(((...(((..(((((...((((....))))..)))))))).......((((-((......))))))...)))..)))))).---.)))))... ( -42.40) >DroPer_CAF1 6395 104 + 1 GUGCUCUCGGCUGUGGCAUUGGCUUCGGCUAUCUGCAUCGCAUCCCGGUGCAGGCAGUCCCCGUCC-CAGCCUCUUCGGCCUCGGUU------GCUACUGCAGCUGAGGCC--- ........(((((.(((..((((....))))(((((((((.....)))))))))........))).-))))).....((((((((((------((....))))))))))))--- ( -54.50) >consensus GAGCUCUGGGCUGCGGCAUUGGCUUCGGCUAUCUGCAUCGCAUUCCCGUGCAGGCAGAGCCAGUCACCAGCA_GCUCCGCUGCUGCUCCUGCUGCUGCCGCCGCGGAGUCCUCU ..((....(((((.(((....))).))))).(((((...((((....))))..)))))...........))..(((((((.((.((..........)).)).)))))))..... (-28.46 = -28.38 + -0.08)

| Location | 19,859,939 – 19,860,049 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 71.39 |
| Mean single sequence MFE | -52.25 |
| Consensus MFE | -20.06 |
| Energy contribution | -21.07 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.67 |
| Structure conservation index | 0.38 |
| SVM decision value | 2.32 |
| SVM RNA-class probability | 0.992271 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19859939 110 + 23771897 CAUUCCCGUGCAGGCAGAGCCAGUGACCAGCA-GUUCCGCUGCUGUUCCUGCUGCUGCCGCCGCGGAGUCCACUGCUCCGCUGUUAACUGCCGGAGGAGCUGCUCCGGCGG .......(.((.((((((((.((.((.(((((-(.....)))))).))))))).))))))))(((((((.....)))))))......(((((((((......))))))))) ( -57.00) >DroPse_CAF1 6430 98 + 1 CAUCCCGGUGCAGGCAGUCCCCGUCC-CAGCCUCUUCGGCCUCGGUU------GCUACUGCAGCCGAGGCC---GCUCCAUUGCUGACGAGCGGAGGCG---GACCGGCAC ....(((((.(.(((.......))).-..((((((..((((((((((------((....))))))))))))---((((..........)))))))))))---.)))))... ( -52.90) >DroSec_CAF1 5890 110 + 1 UAUUCCCGUGCAGGCAGAGCCAGUGACCAGCA-GCUCAGCUGCUGCUCCUGCUGCUGCCUCCGCGGAGUCCUCUGCUCCACUGCUGACUGCCGGAGGAGCGGCACCGGCUG .....(((((.(((((((((.((.((.(((((-((...))))))).))))))).)))))).)))))((((..(((((((.((((.....).))).)))))))....)))). ( -55.20) >DroSim_CAF1 5493 110 + 1 CAUUCCCGUGCAGGCAGAGCCAGUGACCAGCA-GCUCCGCUGCUGCCCCUGCUGCUGCCUCCGCGGAGUCCUCUGCUCCGCUGCUGACUGCCGGAGGAGCGGCACCGGCUG .....(((((.(((((((((.((.(..(((((-((...)))))))..)))))).)))))).)))))((((..(((((((.((((.....).))).)))))))....)))). ( -53.50) >DroYak_CAF1 5887 107 + 1 CAUUCCCGUGCAAGCAGAGCCAGUCCCUAGUA-GCACUGCGGCUGCUCCUGCUUCUGCCGUC---GAGUCCUCUGCUCUUCUGCAGACAGCUGGAGGAGCGGCUCCAGCUG .((((.((.(((((((((((.((.((.(((..-...))).))))))).)))))..)))))..---))))..(((((......)))))(((((((((......))))))))) ( -44.20) >DroPer_CAF1 6435 98 + 1 CAUCCCGGUGCAGGCAGUCCCCGUCC-CAGCCUCUUCGGCCUCGGUU------GCUACUGCAGCUGAGGCC---GCUCCAUUGCUGACGAGCGGAGGCG---GACCGGCAC ....(((((.(.(((.......))).-..((((((..((((((((((------((....))))))))))))---((((..........)))))))))))---.)))))... ( -50.70) >consensus CAUUCCCGUGCAGGCAGAGCCAGUCACCAGCA_GCUCCGCUGCUGCUCCUGCUGCUGCCGCCGCGGAGUCCUCUGCUCCACUGCUGACUGCCGGAGGAGCGGCACCGGCUG .................((((.(((........(((((((.((.((..........)).)).)))))))...(((((((.(((........))).)))))))))).)))). (-20.06 = -21.07 + 1.00)


| Location | 19,859,978 – 19,860,071 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 66.86 |
| Mean single sequence MFE | -50.90 |
| Consensus MFE | -19.43 |
| Energy contribution | -19.47 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.45 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.38 |
| SVM decision value | 3.10 |
| SVM RNA-class probability | 0.998445 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19859978 93 + 23771897 UGCUGUUCCUGCUGCUGCCGCCGCGGAGUCCACUGCUCCGCUGUUAACUGCCGGAGGAGCUGCUCCGGCGGGUGCACAAGUAGCACAA------------GUAGU .((((((..(((((((...((((((((((.....)))))))......(((((((((......)))))))))).))...)))))))..)------------))))) ( -44.40) >DroPse_CAF1 6469 93 + 1 CUCGGUU------GCUACUGCAGCCGAGGCC---GCUCCAUUGCUGACGAGCGGAGGCG---GACCGGCACCUGCCCCUGUGGCAGUUGCCGGCUCUGCUGUCGU (((((((------((....))))))))).((---((((..........)))))).((((---((((((((.(((((.....))))).)))))).))))))..... ( -55.60) >DroSec_CAF1 5929 105 + 1 UGCUGCUCCUGCUGCUGCCUCCGCGGAGUCCUCUGCUCCACUGCUGACUGCCGGAGGAGCGGCACCGGCUGUUGCUCAAGCAGCCCAUGCAGGAUCCGCAGUGGU .(((((((((((((((.((((((.(.((((....((......)).)))).)))))))))))))...(((((((.....))))))).....))))...)))))... ( -52.20) >DroSim_CAF1 5532 105 + 1 UGCUGCCCCUGCUGCUGCCUCCGCGGAGUCCUCUGCUCCGCUGCUGACUGCCGGAGGAGCGGCACCGGCUGUUGCUCAAGCAGCCCAUGCAGGAUCCGCUGUGGU ..((((...(((((((.((((((((((((.....))))))).((.....)).))))))))))))..(((((((.....)))))))...))))............. ( -50.50) >DroYak_CAF1 5926 102 + 1 GGCUGCUCCUGCUUCUGCCGUC---GAGUCCUCUGCUCUUCUGCAGACAGCUGGAGGAGCGGCUCCAGCUGUUGCUCCAGCAGCUUAUGCAGGAUCUGCUGUGGU (((.(.((((((.(((((.(..---((((.....))))..).)))))..(((((((.((((((....)))))).))))))).......)))))).).)))..... ( -49.30) >DroPer_CAF1 6474 93 + 1 CUCGGUU------GCUACUGCAGCUGAGGCC---GCUCCAUUGCUGACGAGCGGAGGCG---GACCGGCACCUGCCCCUGUGGCAGUUGCCGGCUCUGCUGUCGU (((((((------((....))))))))).((---((((..........)))))).((((---((((((((.(((((.....))))).)))))).))))))..... ( -53.40) >consensus UGCUGCUCCUGCUGCUGCCGCCGCGGAGUCCUCUGCUCCACUGCUGACUGCCGGAGGAGCGGCACCGGCUGUUGCUCAAGCAGCACAUGCAGGAUCUGCUGUGGU ..((((.......(((((..............(((((((.(((........))).)))))))....(((....)))...)))))....))))............. (-19.43 = -19.47 + 0.03)

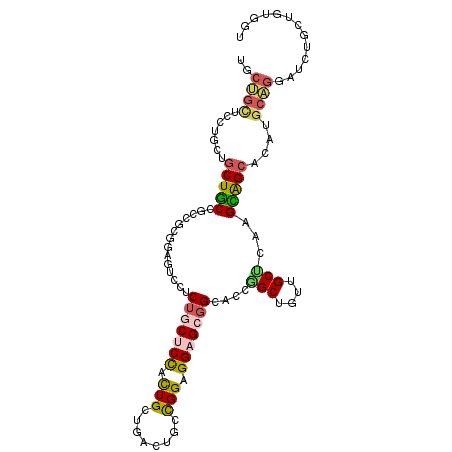

| Location | 19,859,978 – 19,860,071 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 66.86 |
| Mean single sequence MFE | -45.08 |
| Consensus MFE | -15.18 |
| Energy contribution | -15.85 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.34 |
| SVM decision value | 1.24 |
| SVM RNA-class probability | 0.934905 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19859978 93 - 23771897 ACUAC------------UUGUGCUACUUGUGCACCCGCCGGAGCAGCUCCUCCGGCAGUUAACAGCGGAGCAGUGGACUCCGCGGCGGCAGCAGCAGGAACAGCA ....(------------(((((((......((..(((((((((......)))))))........(((((((....).))))))))..)))))).))))....... ( -37.30) >DroPse_CAF1 6469 93 - 1 ACGACAGCAGAGCCGGCAACUGCCACAGGGGCAGGUGCCGGUC---CGCCUCCGCUCGUCAGCAAUGGAGC---GGCCUCGGCUGCAGUAGC------AACCGAG ......((.(..((((((.(((((.....))))).))))))..---)))..((((((..((....))))))---)).(((((.(((....))------).))))) ( -46.30) >DroSec_CAF1 5929 105 - 1 ACCACUGCGGAUCCUGCAUGGGCUGCUUGAGCAACAGCCGGUGCCGCUCCUCCGGCAGUCAGCAGUGGAGCAGAGGACUCCGCGGAGGCAGCAGCAGGAGCAGCA .((.(((((((((((((((.(((((.((....))))))).)))).(((((....((.....))...)))))..)))).))))))).))..((.((....)).)). ( -49.50) >DroSim_CAF1 5532 105 - 1 ACCACAGCGGAUCCUGCAUGGGCUGCUUGAGCAACAGCCGGUGCCGCUCCUCCGGCAGUCAGCAGCGGAGCAGAGGACUCCGCGGAGGCAGCAGCAGGGGCAGCA ......((...((((((((.(((((.((....))))))).))...((((((((.((.....)).(((((((....).))))))))))).))).))))))...)). ( -49.70) >DroYak_CAF1 5926 102 - 1 ACCACAGCAGAUCCUGCAUAAGCUGCUGGAGCAACAGCUGGAGCCGCUCCUCCAGCUGUCUGCAGAAGAGCAGAGGACUC---GACGGCAGAAGCAGGAGCAGCC ......((...((((((.....((((((((((..(((((((((......)))))))))(((((......))))).).)))---..))))))..))))))...)). ( -45.60) >DroPer_CAF1 6474 93 - 1 ACGACAGCAGAGCCGGCAACUGCCACAGGGGCAGGUGCCGGUC---CGCCUCCGCUCGUCAGCAAUGGAGC---GGCCUCAGCUGCAGUAGC------AACCGAG ((..((((.(..((((((.(((((.....))))).))))))..---)....((((((..((....))))))---)).....))))..))...------....... ( -42.10) >consensus ACCACAGCAGAUCCUGCAUGUGCUACUGGAGCAACAGCCGGUGCCGCUCCUCCGGCAGUCAGCAGUGGAGCAGAGGACUCCGCGGCGGCAGCAGCAGGAACAGCA .....................(((((((..((....(((((..........)))))...........(((.......))).))..)))))))............. (-15.18 = -15.85 + 0.67)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:45:51 2006