
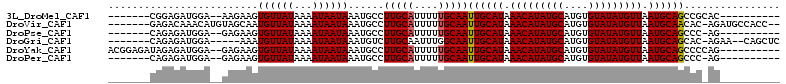
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 19,459,794 – 19,459,887 |
| Length | 93 |
| Max. P | 0.938356 |

| Location | 19,459,794 – 19,459,887 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 84.75 |
| Mean single sequence MFE | -24.87 |
| Consensus MFE | -20.02 |
| Energy contribution | -20.05 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.21 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.938356 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
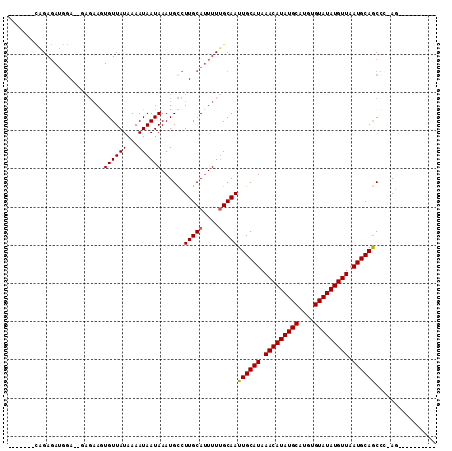
>3L_DroMel_CAF1 19459794 93 + 23771897 -------CGGAGAUGGA--AAGAAGUGUUAUAAAAUAAUAAAUGCCUUGCAUUUUUGCAAUUGCAUAAACAUAUGCAUGUGUAUAUGUUAAUGCAGCCGCAC---------- -------.(((((((.(--(.(...((((((...))))))....).)).)))))))((..((((((.(((((((((....))))))))).))))))..))..---------- ( -25.50) >DroVir_CAF1 52071 102 + 1 -------GAGACAAACAUGUAGCAAUGUUAUAAAAUAAUAAAUGCCUUGCAUUUUUGCAAUUGCAUAAACAUAUGCAUGUGUAUAUGUUAAUGCAACAC-AGAUGCCACC-- -------...........((.(((.(((..................(((((....)))))((((((.(((((((((....))))))))).)))))).))-)..))).)).-- ( -25.30) >DroPse_CAF1 59964 92 + 1 -------CAGAGAUGGA--GAGAAGUGUUAUAAAAUAAUAAAUGCCUUGCAUUUUUGCAAUUGCAUAAACAUAUGCAUGUGUAUAUGUUAAUGCAGCCC-AG---------- -------((((((((((--(.(...((((((...))))))....)))).))))))))...((((((.(((((((((....))))))))).))))))...-..---------- ( -24.70) >DroGri_CAF1 204 97 + 1 -------CAGAGAUGGA-----AAAUGUUAUAAAAUAAUAAAUGUCUUGCAAUUUGGCAAUUGCAUAAACAUAUGCAUGUGUAUAUGUUAAUGCAGCAC-AGAA--CAGCUC -------..((((((..-----...((((((...))))))..))))))((..((((((...(((((.(((((((((....))))))))).))))))).)-))).--..)).. ( -23.10) >DroYak_CAF1 140771 100 + 1 ACGGAGAUAGAGAUGGA--GAGAAGUGUUAUAAAAUAAUAAAUGCCUUGCAUUUUUGCAAUUGCAUAAACAUAUGCAUGUGUAUAUGUUAAUGCAGCCCCAG---------- ..((.(.((((((((((--(.(...((((((...))))))....)))).)))))))))..((((((.(((((((((....))))))))).))))))..))..---------- ( -25.90) >DroPer_CAF1 60246 92 + 1 -------CAGAGAUGGA--GAGAAGUGUUAUAAAAUAAUAAAUGCCUUGCAUUUUUGCAAUUGCAUAAACAUAUGCAUGUGUAUAUGUUAAUGCAGCCC-AG---------- -------((((((((((--(.(...((((((...))))))....)))).))))))))...((((((.(((((((((....))))))))).))))))...-..---------- ( -24.70) >consensus _______CAGAGAUGGA__GAGAAGUGUUAUAAAAUAAUAAAUGCCUUGCAUUUUUGCAAUUGCAUAAACAUAUGCAUGUGUAUAUGUUAAUGCAGCCC_AG__________ .........................((((((...))))))......(((((....)))))((((((.(((((((((....))))))))).))))))................ (-20.02 = -20.05 + 0.03)

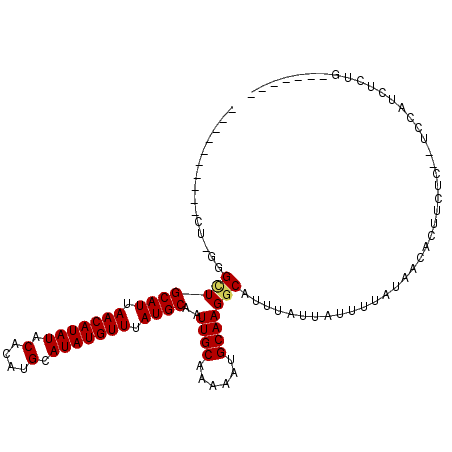
| Location | 19,459,794 – 19,459,887 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 84.75 |
| Mean single sequence MFE | -20.05 |
| Consensus MFE | -15.24 |
| Energy contribution | -15.30 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.51 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.756481 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
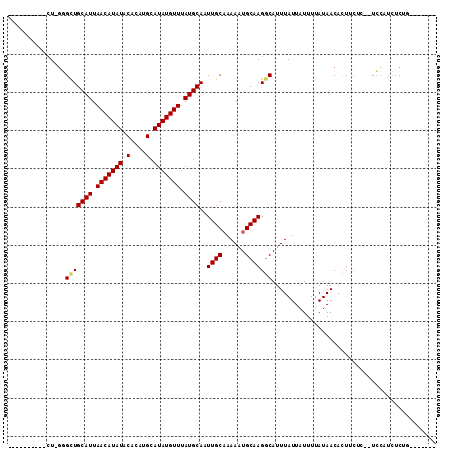
>3L_DroMel_CAF1 19459794 93 - 23771897 ----------GUGCGGCUGCAUUAACAUAUACACAUGCAUAUGUUUAUGCAAUUGCAAAAAUGCAAGGCAUUUAUUAUUUUAUAACACUUCUU--UCCAUCUCCG------- ----------.(((((.(((((.(((((((.(....).))))))).))))).))))).((((((...))))))....................--..........------- ( -20.10) >DroVir_CAF1 52071 102 - 1 --GGUGGCAUCU-GUGUUGCAUUAACAUAUACACAUGCAUAUGUUUAUGCAAUUGCAAAAAUGCAAGGCAUUUAUUAUUUUAUAACAUUGCUACAUGUUUGUCUC------- --.((((((..(-(((((((((.(((((((.(....).))))))).))))))).....((((((...))))))...........))).))))))...........------- ( -25.50) >DroPse_CAF1 59964 92 - 1 ----------CU-GGGCUGCAUUAACAUAUACACAUGCAUAUGUUUAUGCAAUUGCAAAAAUGCAAGGCAUUUAUUAUUUUAUAACACUUCUC--UCCAUCUCUG------- ----------.(-(((((((((.(((((((.(....).))))))).))))..(((((....))))))))..((((......))))........--.)))......------- ( -18.50) >DroGri_CAF1 204 97 - 1 GAGCUG--UUCU-GUGCUGCAUUAACAUAUACACAUGCAUAUGUUUAUGCAAUUGCCAAAUUGCAAGACAUUUAUUAUUUUAUAACAUUU-----UCCAUCUCUG------- (((.((--....-(((.(((((.(((((((.(....).))))))).))))).((((......))))...................)))..-----..)).)))..------- ( -17.70) >DroYak_CAF1 140771 100 - 1 ----------CUGGGGCUGCAUUAACAUAUACACAUGCAUAUGUUUAUGCAAUUGCAAAAAUGCAAGGCAUUUAUUAUUUUAUAACACUUCUC--UCCAUCUCUAUCUCCGU ----------.(((((.(((((.(((((((.(....).))))))).))))).(((((....)))))..........................)--))))............. ( -20.00) >DroPer_CAF1 60246 92 - 1 ----------CU-GGGCUGCAUUAACAUAUACACAUGCAUAUGUUUAUGCAAUUGCAAAAAUGCAAGGCAUUUAUUAUUUUAUAACACUUCUC--UCCAUCUCUG------- ----------.(-(((((((((.(((((((.(....).))))))).))))..(((((....))))))))..((((......))))........--.)))......------- ( -18.50) >consensus __________CU_GGGCUGCAUUAACAUAUACACAUGCAUAUGUUUAUGCAAUUGCAAAAAUGCAAGGCAUUUAUUAUUUUAUAACACUUCUC__UCCAUCUCUG_______ ...............(((((((.(((((((.(....).))))))).))))..((((......)))))))........................................... (-15.24 = -15.30 + 0.06)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:42:41 2006