
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 19,152,331 – 19,152,472 |
| Length | 141 |
| Max. P | 0.712822 |

| Location | 19,152,331 – 19,152,434 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.43 |
| Mean single sequence MFE | -26.38 |
| Consensus MFE | -13.01 |
| Energy contribution | -14.23 |
| Covariance contribution | 1.23 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.712822 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
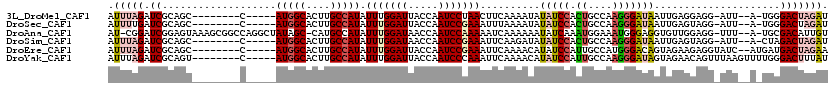
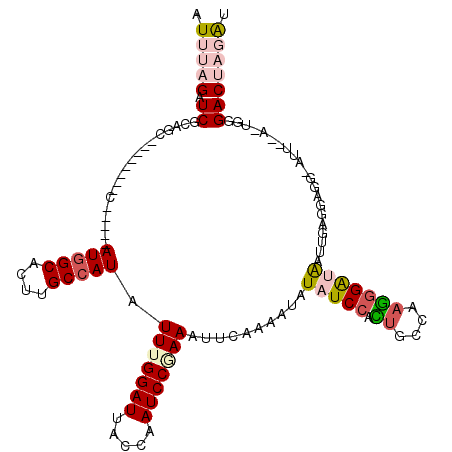
>3L_DroMel_CAF1 19152331 103 - 23771897 AUUUAGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCUAACUUCAAAAUAUAUCCACUGCCAAGGGAUAAUUGAGGAGG-AUU--A-UGGGACUAGAU (((((((((.(((.--------.-----(((((....)))))..))))))..((((((((..((((((....(((((.((....))))))).)))))))))-)).--.-)))..)))))) ( -30.10) >DroSec_CAF1 15150 103 - 1 AUUUUGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCGAAAUUUAAAAUAUAUCCACUGCCAAGGGAUAAUUGAGUAGG-AUU--A-UGGGACUAGAU ....(((((.(((.--------.-----(((((....)))))..))))))))(((((((...((((((....(((((.((....))))))).)))))).))-)).--.-)))........ ( -25.70) >DroAna_CAF1 8272 114 - 1 AU-CGGAUCGGAGUAAAGCGGCCAGGCUAUAGC-CAUGCCAUAUUUGGAUAACCAAUCCAAAAAUCAAAAAAUAUCAAAUGGAAAUGGGAGGUGUUGGAGG-UUU--A-UGCGACAUUGU ..-....(((..(((((.(..((((((....))-)(((((...(((((((.....)))))))............((..((....))..)))))))))).).-)))--)-).)))...... ( -26.30) >DroSim_CAF1 14823 103 - 1 AUUUAGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUAACCAAUCCGAAAUUCAAGAUAUAUCCACUGCCAAGGGAUAAUUGAGUAGG-AUU--A-CUAGACUAGAU (((((((((.(((.--------.-----(((((....)))))..))))))....(((((...((((((....(((((.((....))))))).)))))).))-)))--.-.....)))))) ( -28.00) >DroEre_CAF1 15798 105 - 1 AUUUAGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCGAAAUUCAAAACAUAUCCAUUGCCAUGGGACAGUAGAAGAGGUAUC--AUGAUGACUAGAA .(((((.((((((.--------.-----(((((....))))).(((((((.....)))))))................))))((((..((..(....)..))..)--)))..))))))). ( -22.90) >DroYak_CAF1 15897 107 - 1 AUUUAGAUCGCAGU--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCCAAAUUCAAAACAUAUCCAUUGCCAAGGGAUAGUAGAACAGUUUAAGUUUUGGGACUUUAU .....((((.(((.--------.-----(((((....)))))..))))))).....(((((((....((((.(((((.........)))))(.....).))))....)))))))...... ( -25.30) >consensus AUUUAGAUCGCAGC________C_____AUGGCACUUGCCAUAUUUGGAUUACCAAUCCGAAAUUCAAAAUAUAUCCACUGCCAAGGGAUAAUUGAGGAGG_AUU__A_UGGGACUAGAU .(((((.((...................(((((....))))).(((((((.....)))))))..........(((((.((....))))))).....................))))))). (-13.01 = -14.23 + 1.23)

| Location | 19,152,367 – 19,152,472 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.11 |
| Mean single sequence MFE | -27.00 |
| Consensus MFE | -22.59 |
| Energy contribution | -23.40 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.581099 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19152367 105 - 23771897 UCCAUUGUGCGCUGGUGUCUAGUGCUU--UGGCACUCUAGAUUUAGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCUAACUUCAAAAUAUAUCCACU ((((.((((.((.((((((.(((((..--..)))))...(((....))).....--------.-----..)))))).))))))..))))............................... ( -24.60) >DroSec_CAF1 15186 106 - 1 UCCAUUGUGCACUGGUGUCUAGUGCUU-UUGGCACUCCAGAUUUUGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCGAAAUUUAAAAUAUAUCCACU ......(((..((((.....(((((..-...)))))))))(((((((.......--------.-----(((((....))))).(((((((.....)))))))..))))))).....))). ( -26.90) >DroAna_CAF1 8308 118 - 1 UCCAUUGUGUGCGGGUGUCUAGUGCUUUUUGGCACUCCAGAU-CGGAUCGGAGUAAAGCGGCCAGGCUAUAGC-CAUGCCAUAUUUGGAUAACCAAUCCAAAAAUCAAAAAAUAUCAAAU .....((((.((((.(((.....(((....)))(((((.(((-...))))))))...))).)).(((....))-)..))))))(((((((.....))))))).................. ( -30.00) >DroSim_CAF1 14859 106 - 1 UCCAUUGUGCGCUGGUGUCUGGUGCUU-UUGGCACUCCAGAUUUAGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUAACCAAUCCGAAAUUCAAGAUAUAUCCACU ....(((((..((((.(((((((((..-...)))..))))))))))..))))).--------.-----(((((....))))).(((((((.....))))))).................. ( -28.50) >DroEre_CAF1 15836 106 - 1 UCCAUUGUGCGCUGGUGUCUAGUGCUU-UUGGCACUCCAGAUUUAGAUCGCAGC--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCGAAAUUCAAAACAUAUCCAUU ....(((((..((((.(((((((((..-...)))))..))))))))..))))).--------.-----(((((....))))).(((((((.....))))))).................. ( -26.70) >DroYak_CAF1 15937 106 - 1 UCCAUUGUGCGCUGGUGUCUAGUGCUU-UUGGCACUCCAGAUUUAGAUCGCAGU--------C-----AUGGCACUUGCCAUAUUUGGAUUACCAAUCCCAAAUUCAAAACAUAUCCAUU ...((((((..((((.(((((((((..-...)))))..))))))))..))))))--------.-----(((((....))))).((((((((..........))))))))........... ( -25.30) >consensus UCCAUUGUGCGCUGGUGUCUAGUGCUU_UUGGCACUCCAGAUUUAGAUCGCAGC________C_____AUGGCACUUGCCAUAUUUGGAUUACCAAUCCGAAAUUCAAAAUAUAUCCACU ....(((((..((((.((((((((((....))))))..))))))))..)))))...............(((((....))))).(((((((.....))))))).................. (-22.59 = -23.40 + 0.81)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:37:40 2006