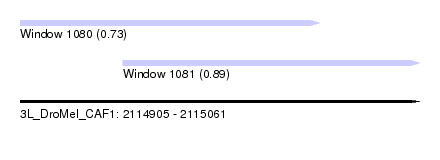
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 2,114,905 – 2,115,061 |
| Length | 156 |
| Max. P | 0.888812 |

| Location | 2,114,905 – 2,115,022 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.25 |
| Mean single sequence MFE | -46.87 |
| Consensus MFE | -28.05 |
| Energy contribution | -29.17 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.733866 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
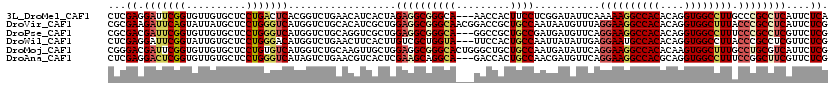
>3L_DroMel_CAF1 2114905 117 + 23771897 CUCGAGGAUUCGGUGUUGUGCUCCUGACUCACGGUCUGAACAUCACUAGAGGCGGGCA---AACCACUUCCUCGGAUAUUCAAAAAGGCCACACAGGUGGCCUUGCCCGCCUCAUUCUCA ...((((((..((((.(((..((..(((.....))).))))).)))).(((((((((.---..((........)).........((((((((....))))))))))))))))))))))). ( -51.00) >DroVir_CAF1 187185 120 + 1 CGCGAAGAUUCAGUAUUAUGCUCCUGGGUCAUGGUCUGCACAUCGCUGGAGGCGGGCAACGGACCGCUGCCAAUAAUGUUUAGGAAGGCCACACAGGUGGCUUUACCCGCCUCAUUCUCG .((((.(...(((.((((((((....)).)))))))))..).))))..((((((((((.((...)).))))...........((((((((((....)))))))).))))))))....... ( -44.70) >DroPse_CAF1 201696 117 + 1 CGCGACGAUUCGGUGUUGUGCUCCUGGGUCAUGGUCUGCAGGUCGCUGGAGGCGGGCA---GGCCGCUGCCGAUGAUGUUCAGGAAGGCCACACAGGUGGCCUUUCCCGCCUCGUUCUCG .((((((((((((.(.....).)).))))).((.....)).)))))..((((((((((---(....)))))...........((((((((((....)))))))))).))))))....... ( -51.70) >DroWil_CAF1 209691 117 + 1 CUCGAGGAUUCGGUAUUGUGCUCCUGGGACAUGGUCUGAACUUCACUUGUCGCUGGUA---UUCCACUGCCAAUUAUAUUGAGGAAUGCCACACAGGUGGCCUUACCCGCCUCGUUCUCG ..(((((.(((((.((..((.((....))))..)))))))..((((((((.(.(((((---((((.(.............).)))))))))))))))))).........)))))...... ( -38.52) >DroMoj_CAF1 165928 120 + 1 CGGGACGAUUCGGUGUUGUGCUCCUGUGUCAUGGUCUGCAAGUUGCUGGAGGCGGGCACUGGGCUGCUGCCAAUGAUAUUCAGGAAGGCCACACAAGUGGCUUUGCCUGCGUCAUUCUCG (((((......(((((...((.((.(((((...((((.((......)).)))).))))).)))).)).))).(((((...((((((((((((....)))))))).)))).)))))))))) ( -46.80) >DroAna_CAF1 167972 117 + 1 CUCGAGGACUCGGUGUUGUGCUCCUGGGUCAUAGUCUGAACGUCACUCGAAGCAGGCA---GACCACUGCCAACGAUGUUCAGGAAGGCCACGCAGGUGGCCUUUCCGGCUUCGUUCUCG ..(((((((((((...((((((....)).))))..)))).........(((((.((((---(....)))))...........((((((((((....))))))))))..)))))))))))) ( -48.50) >consensus CGCGAGGAUUCGGUGUUGUGCUCCUGGGUCAUGGUCUGAACGUCACUGGAGGCGGGCA___GACCACUGCCAAUGAUAUUCAGGAAGGCCACACAGGUGGCCUUACCCGCCUCAUUCUCG ...((.(((((((..........)))))))..................((((((((((.........))))...........((((((((((....)))))))).))))))))....)). (-28.05 = -29.17 + 1.12)
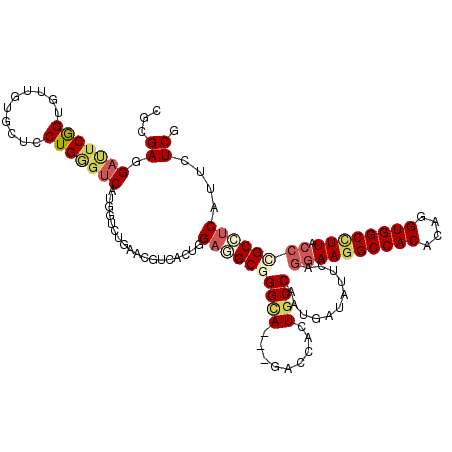
| Location | 2,114,945 – 2,115,061 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 80.19 |
| Mean single sequence MFE | -47.59 |
| Consensus MFE | -31.52 |
| Energy contribution | -33.05 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.888812 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 2114945 116 + 23771897 CAUCACUAGAGGCGGGCA---AACCACUUCCUCGGAUAUUCAAAAAGGCCACACAGGUGGCCUUGCCCGCCUCAUUCUCAGCCACACAGCUGAUCUUGCCAACGGCAUCGUGGGUUAGC ..((((..(((((((((.---..((........)).........((((((((....)))))))))))))))))....(((((......)))))...((((...))))..))))...... ( -49.10) >DroVir_CAF1 187225 119 + 1 CAUCGCUGGAGGCGGGCAACGGACCGCUGCCAAUAAUGUUUAGGAAGGCCACACAGGUGGCUUUACCCGCCUCAUUCUCGGCCAUGCAGCUGAUCUUGCCGACGGCAUCGUGGGUUAAC (((.((((((((((((((.((...)).))))...........((((((((((....)))))))).)))))))).....)))).))).((((.((..((((...))))..)).))))... ( -44.30) >DroPse_CAF1 201736 116 + 1 GGUCGCUGGAGGCGGGCA---GGCCGCUGCCGAUGAUGUUCAGGAAGGCCACACAGGUGGCCUUUCCCGCCUCGUUCUCGGCCACACAGCUGAUCUUGCCGACGGCGUCGUGGGUGAGC ........((((((((((---(....)))))...........((((((((((....)))))))))).))))))...((((.((((((.((((.((.....)))))))).)))).)))). ( -55.20) >DroWil_CAF1 209731 116 + 1 CUUCACUUGUCGCUGGUA---UUCCACUGCCAAUUAUAUUGAGGAAUGCCACACAGGUGGCCUUACCCGCCUCGUUCUCGGCAACGCAGCUUAUCUUACCCACUGCAUCGUGGGUUAGC ..((((((((.(.(((((---((((.(.............).)))))))))))))))))).((.((((((.........(....)((((.............))))...)))))).)). ( -36.54) >DroMoj_CAF1 165968 119 + 1 AGUUGCUGGAGGCGGGCACUGGGCUGCUGCCAAUGAUAUUCAGGAAGGCCACACAAGUGGCUUUGCCUGCGUCAUUCUCGGCCGUGCAGCUGAUCUUGCCAACGGCAUCGUGCGUUAGC .(.(((((..((((((...(.((((((.(((((((((...((((((((((((....)))))))).)))).))))))...)))...)))))).).))))))..))))).).......... ( -53.10) >DroAna_CAF1 168012 116 + 1 CGUCACUCGAAGCAGGCA---GACCACUGCCAACGAUGUUCAGGAAGGCCACGCAGGUGGCCUUUCCGGCUUCGUUCUCGGCCACGCAACUGAUCUUGCCGACGGCGUCGUGGGUCAGC .(((...((((((.((((---(....)))))...........((((((((((....))))))))))..)))))).....))).......(((((((...((((...)))).))))))). ( -47.30) >consensus CGUCACUGGAGGCGGGCA___GACCACUGCCAAUGAUAUUCAGGAAGGCCACACAGGUGGCCUUACCCGCCUCAUUCUCGGCCACGCAGCUGAUCUUGCCGACGGCAUCGUGGGUUAGC ........((((((((((.........))))...........((((((((((....)))))))).))))))))....(((((......))))).((.(((.(((....))).))).)). (-31.52 = -33.05 + 1.53)

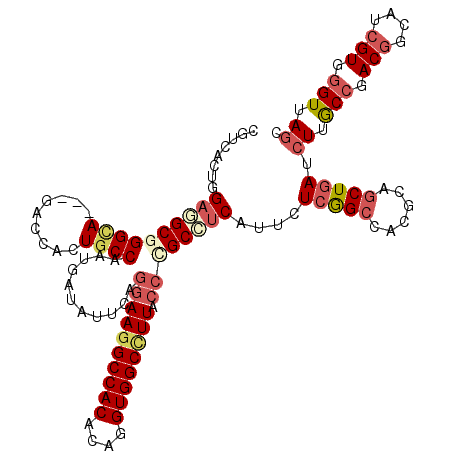
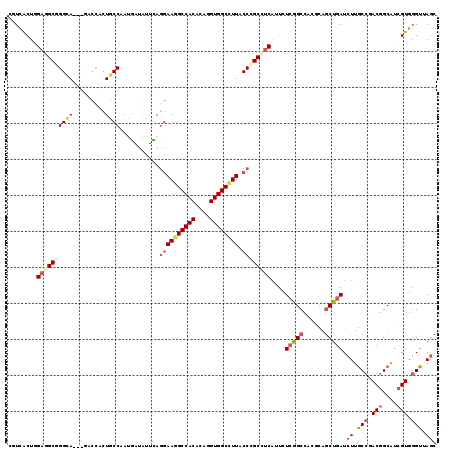
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:49:02 2006