
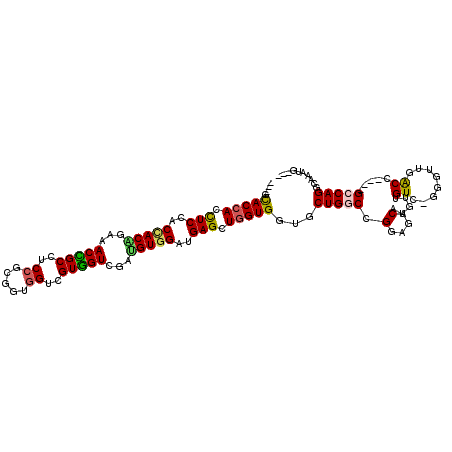
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 19,033,983 – 19,034,090 |
| Length | 107 |
| Max. P | 0.939077 |

| Location | 19,033,983 – 19,034,090 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 80.17 |
| Mean single sequence MFE | -52.38 |
| Consensus MFE | -29.06 |
| Energy contribution | -29.39 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.89 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.30 |
| SVM RNA-class probability | 0.939077 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19033983 107 + 23771897 ---GCACCAUUUCCACCACGGAAACCGCCUCCGCGGUGGUCGUGGUCGAUGUGGACGAGCUGGUGGUGCUGGCCGGAGACAUGUUGUCCGGGUUAACC----GCCAGGCACAUG--- ---((.((((.((.(((((((..(((((....)))))..))))))).)).)))).....((((((((...(.(((((.(.....).))))).)..)))----))))))).....--- ( -53.90) >DroPse_CAF1 175678 107 + 1 ---GCACCACCUCCACCACAGACACCGCCUCCGCGGUGGUUGUGGUCGAUGUGGAUGAGCUGGUGGUGCUGGCCGGCGACAUGUUGUC---GCUGGCC----GACAGAUAGAUGCAG ---((((((((((.(((((((.((((((....)))))).))))))).)).((......)).))))))))(((((((((((.....)))---)))))))----).............. ( -59.00) >DroEre_CAF1 169016 107 + 1 ---GCACCAUCUCCACUACAGAGACUGCCUCCGCAGUGGUCGUGGUCGAUGUGGACGAGCUGGUGGUGCUGGCCGGAGACAUGUUGUCUGGGUUUACC----GCCAGGCACAUG--- ---.(((((.(((..(((((..(((..(..((.....))..)..)))..)))))..))).)))))((((((((...((((.....))))((.....))----)))).))))...--- ( -46.20) >DroMoj_CAF1 192748 110 + 1 ---GCACCAGCUCCACCACUGAGACAGCCUCGGCCGUGGUCGUUGUUGAAGUGGAUGAGCUGGUGGUGCUGGCUGGUGACAUGUUGUCAGCAUU-ACCGCAUGCCAGCCCAAUC--- ---.(((((((((..(((((..((((((...(((....)))))))))..)))))..))))))))).....((((((((((.....))).((...-...))..))))))).....--- ( -52.20) >DroAna_CAF1 164733 110 + 1 CUCGUACAACUUCUACGACAGAAACCGCCUCCGCUGUGGUCGUGGUCGAGGUGGAUGAGCUGGUGGUGCUGGCCGGGGACAUGUUGUCCGGAUUCACG----GCCAGGGCAAUG--- .(((((.......)))))(((...(((((((.((..(....)..)).))))))).....)))...((.(((((((.((((.....))))(....).))----))))).))....--- ( -44.00) >DroPer_CAF1 176892 107 + 1 ---GCACCACCUCCACCACAGACACCGCCUCCGCGGUGGUUGUGGUCGAUGUGGAUGAGCUGGUGGUGCUGGCCGGCGACAUGUUGUC---GCUGGCC----GACAGGUAGAUGCAG ---((((((((((.(((((((.((((((....)))))).))))))).)).((......)).))))))))(((((((((((.....)))---)))))))----).............. ( -59.00) >consensus ___GCACCACCUCCACCACAGAAACCGCCUCCGCGGUGGUCGUGGUCGAUGUGGAUGAGCUGGUGGUGCUGGCCGGAGACAUGUUGUC_GGGUUGACC____GCCAGGCAAAUG___ ....(((((.(((..(((((...(((((..((.....))..)))))...)))))..))).)))))...(((((.((((((.....)))).......))....))))).......... (-29.06 = -29.39 + 0.34)

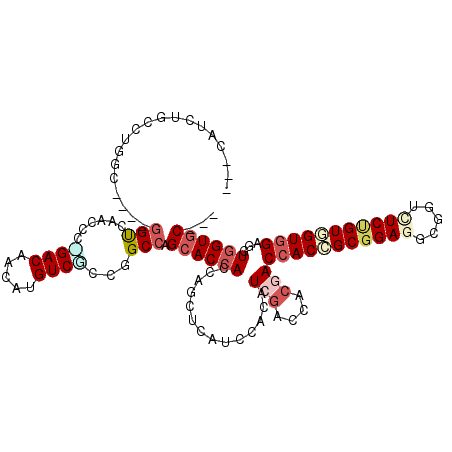
| Location | 19,033,983 – 19,034,090 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 80.17 |
| Mean single sequence MFE | -46.20 |
| Consensus MFE | -23.97 |
| Energy contribution | -25.42 |
| Covariance contribution | 1.45 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.894801 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 19033983 107 - 23771897 ---CAUGUGCCUGGC----GGUUAACCCGGACAACAUGUCUCCGGCCAGCACCACCAGCUCGUCCACAUCGACCACGACCACCGCGGAGGCGGUUUCCGUGGUGGAAAUGGUGC--- ---...((((.((((----((....))((((.........))))))))))))(((((....(((......))).....(((((((((((.....)))))))))))...))))).--- ( -44.00) >DroPse_CAF1 175678 107 - 1 CUGCAUCUAUCUGUC----GGCCAGC---GACAACAUGUCGCCGGCCAGCACCACCAGCUCAUCCACAUCGACCACAACCACCGCGGAGGCGGUGUCUGUGGUGGAGGUGGUGC--- ...............----((((.((---(((.....))))).)))).((((((((.(........).((.((((((..((((((....))))))..)))))).))))))))))--- ( -56.30) >DroEre_CAF1 169016 107 - 1 ---CAUGUGCCUGGC----GGUAAACCCAGACAACAUGUCUCCGGCCAGCACCACCAGCUCGUCCACAUCGACCACGACCACUGCGGAGGCAGUCUCUGUAGUGGAGAUGGUGC--- ---...((((.((((----((....)).((((.....))))...))))))))(((((.((((((......)))......((((((((((.....))))))))))))).))))).--- ( -43.40) >DroMoj_CAF1 192748 110 - 1 ---GAUUGGGCUGGCAUGCGGU-AAUGCUGACAACAUGUCACCAGCCAGCACCACCAGCUCAUCCACUUCAACAACGACCACGGCCGAGGCUGUCUCAGUGGUGGAGCUGGUGC--- ---....((((((((..(((..-..)))((((.....))))...)))))).))((((((((..(((((((......))..(((((....)))))...)))))..))))))))..--- ( -47.60) >DroAna_CAF1 164733 110 - 1 ---CAUUGCCCUGGC----CGUGAAUCCGGACAACAUGUCCCCGGCCAGCACCACCAGCUCAUCCACCUCGACCACGACCACAGCGGAGGCGGUUUCUGUCGUAGAAGUUGUACGAG ---.......(((((----((.(.....((((.....))))))))))))..................((((((.((..(.((.((((((.....)))))).)).)..)).)).)))) ( -29.60) >DroPer_CAF1 176892 107 - 1 CUGCAUCUACCUGUC----GGCCAGC---GACAACAUGUCGCCGGCCAGCACCACCAGCUCAUCCACAUCGACCACAACCACCGCGGAGGCGGUGUCUGUGGUGGAGGUGGUGC--- ...............----((((.((---(((.....))))).)))).((((((((.(........).((.((((((..((((((....))))))..)))))).))))))))))--- ( -56.30) >consensus ___CAUCUGCCUGGC____GGUCAACCC_GACAACAUGUCGCCGGCCAGCACCACCAGCUCAUCCACAUCGACCACGACCACCGCGGAGGCGGUCUCUGUGGUGGAGGUGGUGC___ ...................(((......((((.....))))...))).((((((..............(((....)))(((((((((((.....)))))))))))...))))))... (-23.97 = -25.42 + 1.45)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:36:04 2006