
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 18,984,526 – 18,984,651 |
| Length | 125 |
| Max. P | 0.979533 |

| Location | 18,984,526 – 18,984,622 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 80.43 |
| Mean single sequence MFE | -24.22 |
| Consensus MFE | -12.05 |
| Energy contribution | -11.94 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.691368 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
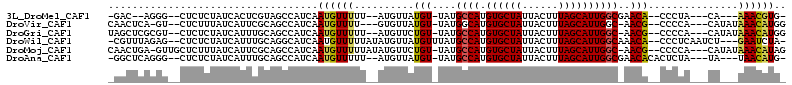
>3L_DroMel_CAF1 18984526 96 + 23771897 -GAC--AGGG--CUCUCUAUCACUCGUAGCCAUCAAUGUUUUU--AUGUUAUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGCGAACA--CCCUA---CA---AAACGUG- -((.--..((--((..(........).)))).)).((((((..--.(((..(((-(.(((((.((((((......)))))))))))))))--....)---))---)))))).- ( -23.70) >DroVir_CAF1 132899 100 + 1 CAACUCA-GU--CUCUUUAUCAUUCGCAGCCAUCAAUGUUUU---GUGUUAUGU-UAUGGCAUGUGCUAUUACUUUAGCAUUGGC-AACG--CCCCA---CAUAUAAACAUGG .......-..--.......................((((((.---....(((((-...(((..((((((......)))))).(..-..))--))..)---)))).)))))).. ( -18.30) >DroGri_CAF1 120054 102 + 1 UAGCUCGCGU--CUCUCUAUCAUUUGCAGCCAUCAAUGUUUUU--AUGUUCUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGC-AACG--CCCCA---CAUAUAAACAUGG ..(((.(((.--............)))))).....((((((.(--((((...((-..(((((.((((((......))))))))))-)..)--)...)---)))).)))))).. ( -26.92) >DroWil_CAF1 152559 104 + 1 -CGUUUAGAG--CUCUCUAUCAUUUGCAGGCAUCAAUGUUUUUAUAUGUUAUGUUUAUGCCAUGUGCUAUUACUUUAGCAUUGGCAAACA--CCCUCAAUCU---GAAUCUA- -.((((((((--(............))((((((.(((((......))))))))))).(((((.((((((......)))))))))))....--.......)))---))))...- ( -23.10) >DroMoj_CAF1 136140 105 + 1 CAACUGA-GUUGCUCUUUAUCAUUCGCAGCCAUCAAUGUUUUUAUAUGUUCUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGC-AACG--CCCCA---CAUAUAAACAUAG ....((.-(((((............)))))))....(((.(((((((((...((-..(((((.((((((......))))))))))-)..)--)...)---)))))))).))). ( -27.40) >DroAna_CAF1 116043 100 + 1 -GGCUCAGGG--CUCUCUAUCAUUUGCAGCCAUCAAUGUUUUU--AUGUUAUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGCGAACACACUCUA---UA---UAACAUG- -((((((((.--..........)))).))))...........(--(((((((((-...((((.((((((......))))))))))((......)).)---))---)))))))- ( -25.90) >consensus _AACUCAGGG__CUCUCUAUCAUUCGCAGCCAUCAAUGUUUUU__AUGUUAUGU_UAUGCCAUGUGCUAUUACUUUAGCAUUGGC_AACA__CCCCA___CA___AAACAUG_ ...................................((((((..........(((....((((.((((((......))))))))))..)))...............)))))).. (-12.05 = -11.94 + -0.11)


| Location | 18,984,556 – 18,984,651 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 78.50 |
| Mean single sequence MFE | -23.32 |
| Consensus MFE | -13.87 |
| Energy contribution | -13.71 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979533 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 18984556 95 + 23771897 AUGUUUUU--AUGUUAUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGCGAACA--CCCUA---CA---AAACGUG-CAGGGCAACCC--AACUUCGCCUAGAACCCC ((((((..--.(((..(((-(.(((((.((((((......)))))))))))))))--....)---))---)))))).-..((((.....--......))))........ ( -23.30) >DroGri_CAF1 120087 93 + 1 AUGUUUUU--AUGUUCUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGC-AACG--CCCCA---CAUAUAAACAUGGCACAUUACCCC--CUCUUUGCU-----GCCC ((((((.(--((((...((-..(((((.((((((......))))))))))-)..)--)...)---)))).))))))((((((.......--.....)).)-----))). ( -27.20) >DroSec_CAF1 118529 95 + 1 AUGUUUUU--AUGUUAUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGCGAACA--CCCUA---CA---AAACGUG-CAGGGCAACCC--AACUUCGCCUAGAACCCC ((((((..--.(((..(((-(.(((((.((((((......)))))))))))))))--....)---))---)))))).-..((((.....--......))))........ ( -23.30) >DroSim_CAF1 123305 95 + 1 AUGUUUUU--AUGUUAUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGCGAACA--CCCUA---CA---AAACGUG-CAGGGCAACCC--AACUUCGCCUAGAACCCC ((((((..--.(((..(((-(.(((((.((((((......)))))))))))))))--....)---))---)))))).-..((((.....--......))))........ ( -23.30) >DroWil_CAF1 152591 101 + 1 AUGUUUUUAUAUGUUAUGUUUAUGCCAUGUGCUAUUACUUUAGCAUUGGCAAACA--CCCUCAAUCU---GAAUCUA-CAGCACCCCCCCCCCCCCCGCCUA--ACCCC ............((((.((...(((((.((((((......)))))))))))....--........((---(......-)))................)).))--))... ( -16.60) >DroAna_CAF1 116075 89 + 1 AUGUUUUU--AUGUUAUGU-UAUGCCAUGUGCUAUUACUUUAGCAUUGGCGAACACACUCUA---UA---UAACAUG-CAUGGUG----------UGGCAUGGCUCCCC ........--..(((((((-((((((((((((((......))))..((.....)).......---..---......)-)))))))----------)))))))))..... ( -26.20) >consensus AUGUUUUU__AUGUUAUGU_UAUGCCAUGUGCUAUUACUUUAGCAUUGGCGAACA__CCCUA___CA___AAACGUG_CAGGGCAACCC__AACUUCGCCUAGAACCCC ((((((..........(((...(((((.((((((......))))))))))).)))...............))))))................................. (-13.87 = -13.71 + -0.16)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:35:42 2006