
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 18,689,943 – 18,690,043 |
| Length | 100 |
| Max. P | 0.974640 |

| Location | 18,689,943 – 18,690,043 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.63 |
| Mean single sequence MFE | -35.60 |
| Consensus MFE | -22.77 |
| Energy contribution | -22.97 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.847981 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
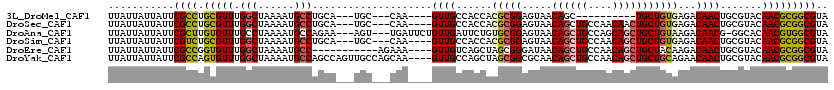
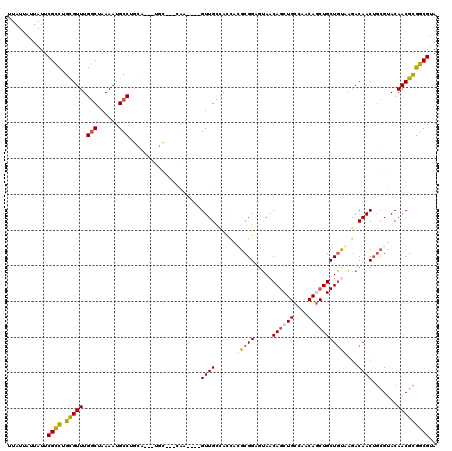
>3L_DroMel_CAF1 18689943 100 + 23771897 UUAUUAUUAUUCGCCUGCGUUUGGCUAAAAUGCCUGCA---UGC---CAA----GUUGCCACCACGCGGAGUAACAGC----------UGCUGUGAGACAACUGCGUACAACGCGGCGUA ...........((((.(((((.(((......))).(.(---(((---..(----((((((((...((((........)----------))).))).).))))))))).)))))))))).. ( -31.20) >DroSec_CAF1 19273 110 + 1 UUAUUAUUAUUCGCCUGCGUUUGGCUAAAAUGCCUGCA---UGC---CAA----GUUGCCACCACGCGGAGUAACAGCUGCCAACAACUGCUGUGAGACAACUGCGUACAACGCGGCGUA ...........((((.(((((.(((......))).(((---...---...----..)))....((((((.((.(((((...........)))))...))..))))))..))))))))).. ( -33.50) >DroAna_CAF1 17137 113 + 1 UUAUUAUUAUUCGCUUGUGUUUGCCUAAAAUGCCAGAA---AGU---UGAUUCUGUUGAUUCUGUGCGGAGUAACAGCUGCCAGCAGCUGCUGUAAGACAACG-GGCACAACGUGGCGUA ............((........)).....((((((...---.((---((..(((((((..(((.(((((.....((((((....)))))))))))))))))))-))..)))).)))))). ( -33.00) >DroSim_CAF1 19286 110 + 1 UUAUUAUUAUUCGUCUGCGUUUGGCUAAAAUGCCUGCA---UGC---CAA----GUUGCCACCACGCGGAGUAACAGCUGCCAACAGCUGCUGUGAGACAACUGCGUACAACGCGGCGUA ...........((.(((((((.(((......))).(((---...---...----..)))....((((((.((.(((((.((.....)).)))))...))..))))))..))))))))).. ( -34.30) >DroEre_CAF1 19262 105 + 1 UUAUUAUUAUUCGCCGGUGUUUGGCUAAAAUGCC-----------AGAAA----GUUGUCAGCUAGCGGGAUAACAGCUGCCAACAGCUGCUACAAGACAACUGCGUACAACGCGGCGUA ...........((((....((((((......)))-----------))).(----((((((...(((((......)(((((....)))))))))...)))))))(((.....))))))).. ( -36.70) >DroYak_CAF1 19959 116 + 1 UUAUUAUUAUUCGCCAGUGUUUGGCUAAAAUGCCAGCCAGUUGCCAGCAA----GUUGCCAGCUAGCGGCGCAACAGCUGCCAACAGCUGCUGCAGAACAACUGCGUACAACGCGGCGUA ...........((((.(((((((((......))))((((((((((.((.(----((.....))).)))))(((.((((((....)))))).))).....))))).))..))))))))).. ( -44.90) >consensus UUAUUAUUAUUCGCCUGCGUUUGGCUAAAAUGCCUGCA___UGC___CAA____GUUGCCACCACGCGGAGUAACAGCUGCCAACAGCUGCUGUAAGACAACUGCGUACAACGCGGCGUA ...........((((.(((((.(((......)))....................((((......(((((.....((((((....)))))))))))...)))).......))))))))).. (-22.77 = -22.97 + 0.20)
| Location | 18,689,943 – 18,690,043 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.63 |
| Mean single sequence MFE | -40.06 |
| Consensus MFE | -22.80 |
| Energy contribution | -24.50 |
| Covariance contribution | 1.70 |
| Combinations/Pair | 1.20 |
| Mean z-score | -3.15 |
| Structure conservation index | 0.57 |
| SVM decision value | 1.73 |
| SVM RNA-class probability | 0.974640 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
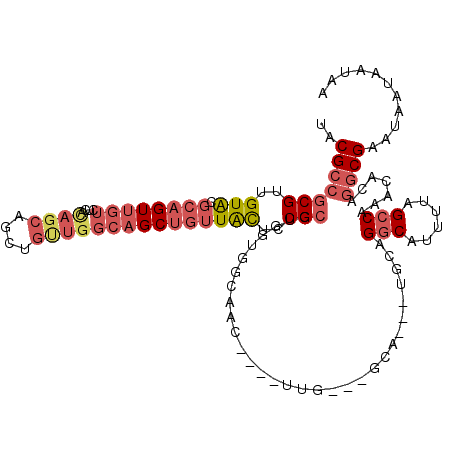
>3L_DroMel_CAF1 18689943 100 - 23771897 UACGCCGCGUUGUACGCAGUUGUCUCACAGCA----------GCUGUUACUCCGCGUGGUGGCAAC----UUG---GCA---UGCAGGCAUUUUAGCCAAACGCAGGCGAAUAAUAAUAA ..((((((((((((.((((((((......)))----------))))))))...(((((.((....)----..)---.))---))).(((......))).))))).))))........... ( -36.40) >DroSec_CAF1 19273 110 - 1 UACGCCGCGUUGUACGCAGUUGUCUCACAGCAGUUGUUGGCAGCUGUUACUCCGCGUGGUGGCAAC----UUG---GCA---UGCAGGCAUUUUAGCCAAACGCAGGCGAAUAAUAAUAA ..((((((((((((.(((((((((((((((((((((....)))))))).......)))).))))))----)..---)).---))).(((......))).))))).))))........... ( -42.41) >DroAna_CAF1 17137 113 - 1 UACGCCACGUUGUGCC-CGUUGUCUUACAGCAGCUGCUGGCAGCUGUUACUCCGCACAGAAUCAACAGAAUCA---ACU---UUCUGGCAUUUUAGGCAAACACAAGCGAAUAAUAAUAA ..(((....(((((..-..((((((...((((((((....)))))))).....((.(((((............---...---))))))).....)))))).))))))))........... ( -30.76) >DroSim_CAF1 19286 110 - 1 UACGCCGCGUUGUACGCAGUUGUCUCACAGCAGCUGUUGGCAGCUGUUACUCCGCGUGGUGGCAAC----UUG---GCA---UGCAGGCAUUUUAGCCAAACGCAGACGAAUAAUAAUAA ..(((.((((((((.(((((((((((((((((((((....)))))))).......)))).))))))----)..---)).---))).(((......))).))))).).))........... ( -38.71) >DroEre_CAF1 19262 105 - 1 UACGCCGCGUUGUACGCAGUUGUCUUGUAGCAGCUGUUGGCAGCUGUUAUCCCGCUAGCUGACAAC----UUUCU-----------GGCAUUUUAGCCAAACACCGGCGAAUAAUAAUAA ..(((((.(((((...(((((((...((((((((((....))))))))))...)).))))))))))----....(-----------(((......)))).....)))))........... ( -38.60) >DroYak_CAF1 19959 116 - 1 UACGCCGCGUUGUACGCAGUUGUUCUGCAGCAGCUGUUGGCAGCUGUUGCGCCGCUAGCUGGCAAC----UUGCUGGCAACUGGCUGGCAUUUUAGCCAAACACUGGCGAAUAAUAAUAA ......(((.....))).(((((((.((((((((((....))))))))))(((((((((.(....)----..))))))...(((((((....)))))))......))))))))))..... ( -53.50) >consensus UACGCCGCGUUGUACGCAGUUGUCUCACAGCAGCUGUUGGCAGCUGUUACUCCGCGUGGUGGCAAC____UUG___GCA___UGCAGGCAUUUUAGCCAAACACAGGCGAAUAAUAAUAA ..(((((((..(((.((((((((....((((....)))))))))))))))..)))...............................(((......))).......))))........... (-22.80 = -24.50 + 1.70)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:15 2006