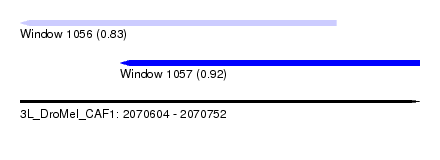
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 2,070,604 – 2,070,752 |
| Length | 148 |
| Max. P | 0.918798 |

| Location | 2,070,604 – 2,070,721 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 76.81 |
| Mean single sequence MFE | -37.47 |
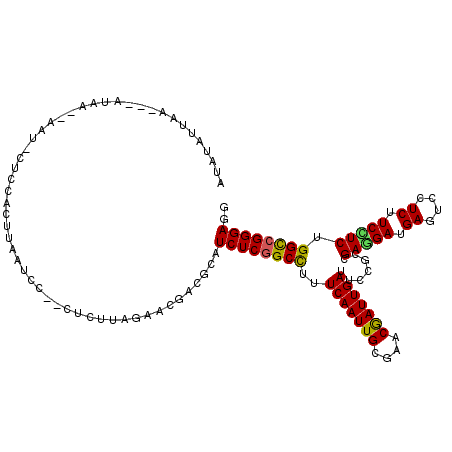
| Consensus MFE | -26.15 |
| Energy contribution | -25.65 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.830052 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
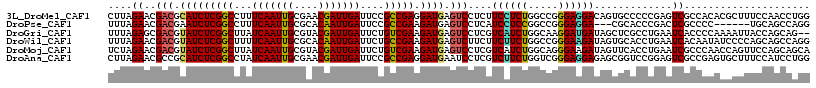
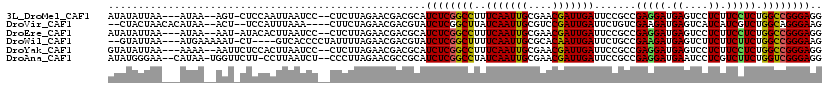
>3L_DroMel_CAF1 2070604 117 - 23771897 CUUAGAACGACGCAUCUCGGCCUUUCAAUUGCGAACGAUUGAUUCCGCCGAGGAUGAGUCCUCUUCCUCUGGCCGGGAGGACAGUGCCCCCGAGUCGCCACACGCUUUCCAACCUGG ..(((...(((.(((((((((...(((((((....)))))))....))))).)))).))).........((((((((.((......))))))....)))).............))). ( -36.60) >DroPse_CAF1 159113 108 - 1 UUUAGAACGACGAAUCUCGGCCUUUCAAUUGCGCACAAUUGAUUCCGCCGAAGAUGAGUCCUCAUCCUCCGGCCGGGAGGA---CGCACCCGACUCGCCCC------UGCAGCCAGG .............((((((((...(((((((....)))))))....)))).))))(((((..(.((((((.....))))))---.).....)))))...((------((....)))) ( -34.50) >DroGri_CAF1 123135 115 - 1 UUUAGAGCGACGUAUCUCGGCUUAUCAAUUGCGUACGAUUGAUUCUGUCGAAGAUGAGUCCUCGUCAUCUGGCAAGGAUGAUAGCUCGCCUGAAUCACCCCAAAAUUACCAGCAG-- ....(((((((.(((((((((..((((((((....))))))))...)))).))))).)))...(((((((.....))))))).))))((.((.................))))..-- ( -34.93) >DroWil_CAF1 166853 117 - 1 UUUAGAACGACGUAUCUCGGCUUUUCAAUUGCGCACAAUUGAUUCUGCCGAAGAUGAGUCUUCUUCUUCUGGCCGGGAAGAUAGUGCACCUGAAUCACAAUAUCCCCAGCAGCCAGG ...((((.(((.(((((((((...(((((((....)))))))....)))).))))).)))))))....(((((.(((..(((((((.........)))..)))))))....))))). ( -37.00) >DroMoj_CAF1 126714 117 - 1 UCUAGAACGACGUAUCUCGGCUUAUCAAUUGCGUACGAUUGAUUCUGUCGAAGAUGAGUCCUCGUCAUCUGGCAGGGAAGAUAGUUCACCUGAAUCGCCCAACCAGUUCCAGCAGCA .((.(((((((.(((((((((..((((((((....))))))))...)))).))))).))).........(((..(((......((((....))))..)))..))))))).))..... ( -33.00) >DroAna_CAF1 128849 117 - 1 CUUAGAACGCCGCAUCUCGGCCUAUCAAUUGCGAACGAUUGAUUCCGCCGAGGAUGAAUCCUCGUCUUCUGGUCGGGAGGAGAGCGGUCCGGAGUCGCCGAGUGCUUUCCAUCCUGG ........(((....((((((..((((((((....))))))))...))))))(((((....)))))....)))(((((((((((((.((.((.....)))).)))))))).))))). ( -48.80) >consensus UUUAGAACGACGUAUCUCGGCCUAUCAAUUGCGAACGAUUGAUUCCGCCGAAGAUGAGUCCUCGUCAUCUGGCCGGGAGGAUAGCGCACCUGAAUCGCCCCACCCUUUCCAGCCAGG ....((..(((.(((((((((...(((((((....)))))))....))))).)))).)))....((((((.....)))))).............))..................... (-26.15 = -25.65 + -0.50)
| Location | 2,070,641 – 2,070,752 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.58 |
| Mean single sequence MFE | -31.12 |
| Consensus MFE | -25.27 |
| Energy contribution | -24.83 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.918798 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
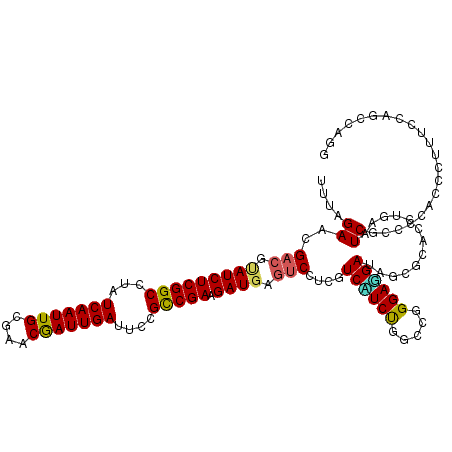
>3L_DroMel_CAF1 2070641 111 - 23771897 AUAUAUUAA---AUAA--AGU-CUCCAAUUAAUCC--CUCUUAGAACGACGCAUCUCGGCCUUUCAAUUGCGAACGAUUGAUUCCGCCGAGGAUGAGUCCUCUUCCUCUGGCCGGGAGG .........---....--...-..........(((--(..(((((..(((.(((((((((...(((((((....)))))))....))))).)))).))).......)))))..)))).. ( -30.30) >DroVir_CAF1 148525 109 - 1 --CUACUAACACAUAA--ACU--UCCAUUUAAA----CUUCUAGAACGACGUAUCUCGGCUUAUCAAUUGCGUCCGAUUGAUUCUGUCGAAGAUGAGUCAUCAUCGUCUGGCAGGGAAG --..............--.((--(((.......----((.(((((..(((.(((((((((..((((((((....))))))))...)))).))))).))).......))))).))))))) ( -27.61) >DroEre_CAF1 131579 111 - 1 AUAUAUUAA---AUAA--AAU-AUACACUUAAUCC--CUCUUAGAACGACGCAUCUCGGCCUUUCAAUUGCGAACGAUUGAUUCCGCCGAGGAUGAGUCCUCUUCCUCUGGCCGGGAGG .((((((..---....--)))-))).......(((--(..(((((..(((.(((((((((...(((((((....)))))))....))))).)))).))).......)))))..)))).. ( -31.40) >DroWil_CAF1 166890 109 - 1 --GUAUUAA---AUGAAAAAU-CU----GUCACCCCUAUUUUAGAACGACGUAUCUCGGCUUUUCAAUUGCGCACAAUUGAUUCUGCCGAAGAUGAGUCUUCUUCUUCUGGCCGGGAAG --.......---.........-..----....((((((....((((.(((.(((((((((...(((((((....)))))))....)))).))))).))))))).....)))..)))... ( -29.40) >DroYak_CAF1 125271 112 - 1 GUAUAUUAA---AAAA--AAUUCUCCACUUAAUCC--CUCUUAGAACGACGCAUCUCGGCCUUUCAAUUGCGAACGAUUGAUUCCGCCGAGGAUGAGUCCUCUUCCUCUGGCCGGGAGG .........---....--..............(((--(..(((((..(((.(((((((((...(((((((....)))))))....))))).)))).))).......)))))..)))).. ( -30.30) >DroAna_CAF1 128886 113 - 1 AUAUGGGAA--CAUAA-UGGUUCUU-CCUUAAUCU--CCCUUAGAACGCCGCAUCUCGGCCUAUCAAUUGCGAACGAUUGAUUCCGCCGAGGAUGAAUCCUCGUCUUCUGGUCGGGAGG ....(((((--(....-..))))))-.......((--(((((((((........((((((..((((((((....))))))))...))))))(((((....)))))))))))..))))). ( -37.70) >consensus AUAUAUUAA___AUAA__AAU_CUCCACUUAAUCC__CUCUUAGAACGACGCAUCUCGGCCUUUCAAUUGCGAACGAUUGAUUCCGCCGAGGAUGAGUCCUCUUCCUCUGGCCGGGAGG .....................................................((((((((..(((((((....))))))).......(((((.((....)).))))).)))))))).. (-25.27 = -24.83 + -0.44)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:48:38 2006