
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 18,305,933 – 18,306,032 |
| Length | 99 |
| Max. P | 0.963215 |

| Location | 18,305,933 – 18,306,032 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.73 |
| Mean single sequence MFE | -25.15 |
| Consensus MFE | -9.37 |
| Energy contribution | -10.90 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.37 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.577765 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
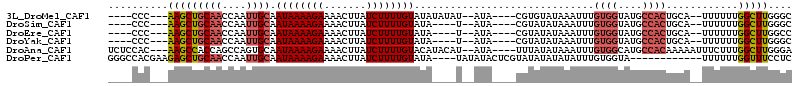
>3L_DroMel_CAF1 18305933 99 + 23771897 GCCCAAGCCAAAAAA--UGCAGUGGCAUACCACAAAUUUAUACACG----UAU--AUAUAUAUACAAAAGAUAAGUUUUCUUUUAUUGCAAUUGGUUGCAGCUU---GGG---- .((((((((((..((--((((((((....))))((((((((....(----(((--(....))))).....))))))))........))).)))..)))..))))---)))---- ( -26.80) >DroSim_CAF1 99196 95 + 1 GCCCAAGCCAAAAAA--UGCAGUGGCAUACCACAAAUUUAUAUACG----UAU--A----UAUACAAAAGAUAAGUUUUCUUUUAUUGCAAUUGGUUGCAGCUU---GGG---- .((((((((((..((--((((((((....)))).....((((((..----..)--)----)))).((((((.......))))))..))).)))..)))..))))---)))---- ( -25.00) >DroEre_CAF1 86473 95 + 1 GGCCAAGCCAAAAAA--UGCAGUGGCAUACCACAAAUUUAUAUACG----UAU--A----UAUACAAAAGAUAAGUUUUCUUUUAUUGCAAUUGGUUGCAGCUU---GGG---- ..(((((((((..((--((((((((....)))).....((((((..----..)--)----)))).((((((.......))))))..))).)))..)))..))))---)).---- ( -23.00) >DroYak_CAF1 96299 95 + 1 GCCCAAGCCAAAAAA--UGCAGUGGCAUACCACAAAUUUAUAUACG----UAU--A----UAUACAAAAGAUAAGUUUUCUUUUAUUGCAAUUGGUUGCAGCUU---GGG---- .((((((((((..((--((((((((....)))).....((((((..----..)--)----)))).((((((.......))))))..))).)))..)))..))))---)))---- ( -25.00) >DroAna_CAF1 87040 105 + 1 UCCCAAGCCAAAGAAAUUUUUGUGGCAUGCCACAAAUUUAUAUAAA----UAU--AUGUAUGUACAAAAGAUAAGUUUUCUUUUAUUGCACUGGCUGGUGGCUU---GUGGAGA ((((((((((.((.....(((((((....)))))))..((((((..----..)--)))))((((.((((((.......))))))..))))....))..))))))---).))).. ( -30.20) >DroPer_CAF1 109561 98 + 1 GAGGAAACCAAAAAA------------UACCACAAAUAUAUAUAUACGAGUAUAUA----UAUACAAAAGAUAAGUUUUCUUUUAUUGCAAUUGGUUGCAGCUCUUCGUGGCCC (.(....))......------------..((((...((((((((((....))))))----)))).((((((.......)))))).((((((....))))))......))))... ( -20.90) >consensus GCCCAAGCCAAAAAA__UGCAGUGGCAUACCACAAAUUUAUAUACG____UAU__A____UAUACAAAAGAUAAGUUUUCUUUUAUUGCAAUUGGUUGCAGCUU___GGG____ ((...((((((..........((((....))))................................((((((.......)))))).......))))))...))............ ( -9.37 = -10.90 + 1.53)


| Location | 18,305,933 – 18,306,032 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.73 |
| Mean single sequence MFE | -26.86 |
| Consensus MFE | -12.63 |
| Energy contribution | -13.38 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.77 |
| Structure conservation index | 0.47 |
| SVM decision value | 1.55 |
| SVM RNA-class probability | 0.963215 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
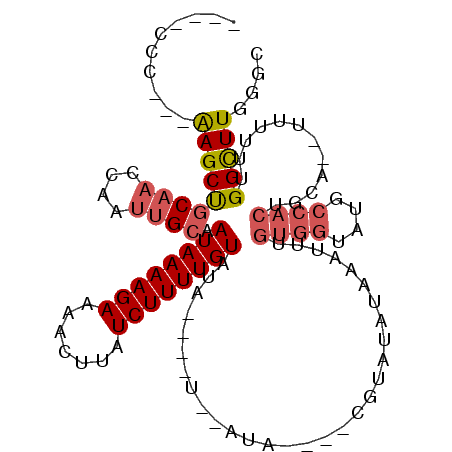
>3L_DroMel_CAF1 18305933 99 - 23771897 ----CCC---AAGCUGCAACCAAUUGCAAUAAAAGAAAACUUAUCUUUUGUAUAUAUAU--AUA----CGUGUAUAAAUUUGUGGUAUGCCACUGCA--UUUUUUGGCUUGGGC ----(((---(((((.........((((((((((((.......)))))))).(((((((--...----.))))))).....((((....))))))))--......)))))))). ( -28.16) >DroSim_CAF1 99196 95 - 1 ----CCC---AAGCUGCAACCAAUUGCAAUAAAAGAAAACUUAUCUUUUGUAUA----U--AUA----CGUAUAUAAAUUUGUGGUAUGCCACUGCA--UUUUUUGGCUUGGGC ----(((---(((((((((....))))).(((((((..........((((((((----(--...----.)))))))))...((((....))))....--)))))))))))))). ( -27.40) >DroEre_CAF1 86473 95 - 1 ----CCC---AAGCUGCAACCAAUUGCAAUAAAAGAAAACUUAUCUUUUGUAUA----U--AUA----CGUAUAUAAAUUUGUGGUAUGCCACUGCA--UUUUUUGGCUUGGCC ----.((---(((((((((....))))).(((((((..........((((((((----(--...----.)))))))))...((((....))))....--))))))))))))).. ( -24.40) >DroYak_CAF1 96299 95 - 1 ----CCC---AAGCUGCAACCAAUUGCAAUAAAAGAAAACUUAUCUUUUGUAUA----U--AUA----CGUAUAUAAAUUUGUGGUAUGCCACUGCA--UUUUUUGGCUUGGGC ----(((---(((((((((....))))).(((((((..........((((((((----(--...----.)))))))))...((((....))))....--)))))))))))))). ( -27.40) >DroAna_CAF1 87040 105 - 1 UCUCCAC---AAGCCACCAGCCAGUGCAAUAAAAGAAAACUUAUCUUUUGUACAUACAU--AUA----UUUAUAUAAAUUUGUGGCAUGCCACAAAAAUUUCUUUGGCUUGGGA ..(((.(---((((((.......((((((...((((.......))))))))))......--...----..........(((((((....)))))))........)))))))))) ( -29.40) >DroPer_CAF1 109561 98 - 1 GGGCCACGAAGAGCUGCAACCAAUUGCAAUAAAAGAAAACUUAUCUUUUGUAUA----UAUAUACUCGUAUAUAUAUAUUUGUGGUA------------UUUUUUGGUUUCCUC ((((((.((((.((..(((.........((((((((.......))))))))(((----((((((....)))))))))..)))..)).------------)))).))))..)).. ( -24.40) >consensus ____CCC___AAGCUGCAACCAAUUGCAAUAAAAGAAAACUUAUCUUUUGUAUA____U__AUA____CGUAUAUAAAUUUGUGGUAUGCCACUGCA__UUUUUUGGCUUGGGC ..........(((((((((....)))).((((((((.......))))))))..............................((((....))))............))))).... (-12.63 = -13.38 + 0.75)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:30:31 2006