
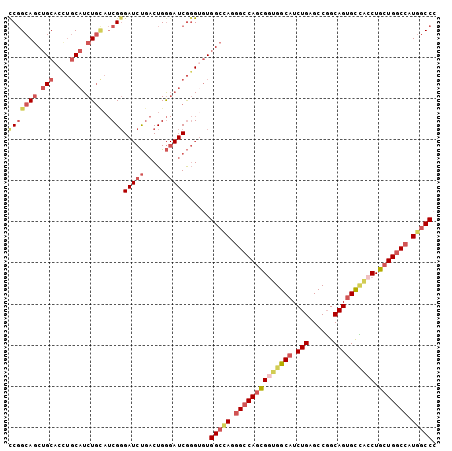
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 17,614,307 – 17,614,449 |
| Length | 142 |
| Max. P | 0.860344 |

| Location | 17,614,307 – 17,614,411 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 84.36 |
| Mean single sequence MFE | -53.33 |
| Consensus MFE | -41.71 |
| Energy contribution | -43.97 |
| Covariance contribution | 2.26 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.69 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.83 |
| SVM RNA-class probability | 0.860344 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 17614307 104 + 23771897 UCGGCAGCUGCACUUGCAUCUGUAUCGGGAUCUGACUGGGAUCGGGUGUGGCCAAGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCUACCUGCUGGCCAUGGCCA ..(((.((..((((((.((((...((((...))))...))))))))))..))((.((((((((((((((.(((......)))))))))).))))))).))))). ( -55.30) >DroSec_CAF1 26565 104 + 1 CCGGCAGCUGCACCUGCAUCUGCAUCGGGAUCUGACUGGGAUCGGGUGUGGCUAGGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCCACCUGCUGGCCAUGGCCC ..((((((..((((((.((((.((((((...)))).))))))))))))..)))..((((((((((((((.(((......)))))))))).)))))))...))). ( -60.70) >DroSim_CAF1 27790 104 + 1 CCGGCAGCUGCACCUGCAUCUGCAUCGGGAUCUGACUGGGAUCGGGUGUGGCUAGGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCCACCUGCUGGCCAUGGCCC ..((((((..((((((.((((.((((((...)))).))))))))))))..)))..((((((((((((((.(((......)))))))))).)))))))...))). ( -60.70) >DroEre_CAF1 27939 104 + 1 CCGGCAGCUGCACCUGCAUCUGCAUCGGGAUCUGACUGGGAUCGGGGUUGGCUAGGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCCACCUGCUGGCCAUGGCCC (((((((.(((....))).))))..)))(((((.....)))))......(((((.((((((((((((((.(((......)))))))))).))))))).))))). ( -57.40) >DroYak_CAF1 28406 104 + 1 CCGGCAGCUGCACCUGCAUCUGCAUCGGGAUCUGACUGGGAUCGGGUGUGGCCAGGGCCAGCGUAGGCAUCUGAGCCGGCAGUGCCACCUGCUGGCCAUGGCCC ..(((.((..((((((.((((.((((((...)))).))))))))))))..))((.((((((((..((((.(((......)))))))...)))))))).))))). ( -52.20) >DroPer_CAF1 29137 92 + 1 CCUGCAUCGGCCCCCCCAUCU----CGUG--CCCGCUGA---CAUGCGUGGUCAGCGACAGUGAGGGCAUCUGAGCCUGCAGCGUGCCCUG-UGGCCCU--CCC ........((((....(((.(----((((--(((((...---...))).)))..))))..)))((((((((((......))).))))))).-.))))..--... ( -33.70) >consensus CCGGCAGCUGCACCUGCAUCUGCAUCGGGAUCUGACUGGGAUCGGGUGUGGCCAGGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCCACCUGCUGGCCAUGGCCC (((((((.(((....))).))))..)))(((((.....)))))......(((((.((((((((((((((.(((......)))))))))).))))))).))))). (-41.71 = -43.97 + 2.26)


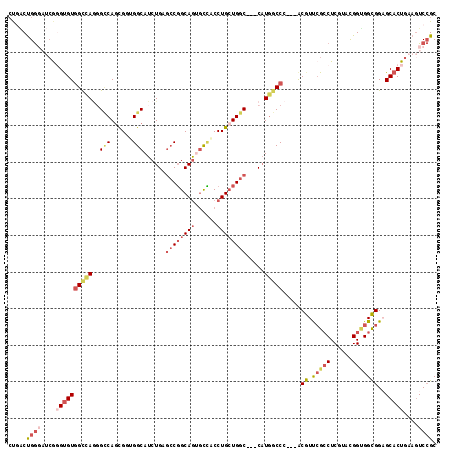
| Location | 17,614,338 – 17,614,449 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 78.50 |
| Mean single sequence MFE | -53.43 |
| Consensus MFE | -29.75 |
| Energy contribution | -31.53 |
| Covariance contribution | 1.78 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.613736 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 17614338 111 + 23771897 CUGACUGGGAUCGGGUGUGGCCAAGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCUACCUGCUGGC---CAUGGCCA---ACGUUCGCCUCGUACGGUGGCGGAGCACUGAAGUCCGC .......((((..((((((((((.((((((((((((((.(((......)))))))))).))))))---).))))).---...((((((.(.....).)))))))))))...)))).. ( -60.10) >DroGri_CAF1 31460 113 + 1 -UCCCCGGC---AUGUGUCGUCACCGUCAACGUGGGCAUCUGAUGCGGCAACAUCACCUGCUGACCCCCAUGACCCAGUGCAUCCGGCUCGUAUGGGGGUGGUGCACUAAAGUCCGC -.....(((---..(((....))).)))...((((((...(((((......)))))..((((.(((((((((.....(.((.....)).).))))))))).).))).....)))))) ( -37.20) >DroSec_CAF1 26596 111 + 1 CUGACUGGGAUCGGGUGUGGCUAGGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCCACCUGCUGGC---CAUGGCCC---ACGUUCGCCUCGUACGGCGGCGGAGCACUGAAGUCCGC .......((((((((((.(((((.((((((((((((((.(((......)))))))))).))))))---).))))))---))(((((((((....)).))).)))).))))..))).. ( -61.00) >DroSim_CAF1 27821 111 + 1 CUGACUGGGAUCGGGUGUGGCUAGGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCCACCUGCUGGC---CAUGGCCC---ACGUUCGCCUCGUACGGUGGCGGAGCACUGAAGUCCGC .......((((((((((.(((((.((((((((((((((.(((......)))))))))).))))))---).))))))---))(((((((.(.....).))).)))).))))..))).. ( -61.10) >DroYak_CAF1 28437 111 + 1 CUGACUGGGAUCGGGUGUGGCCAGGGCCAGCGUAGGCAUCUGAGCCGGCAGUGCCACCUGCUGGC---CAUGGCCC---ACGUUCGCCUCGUACGGUGGCGGAGCACUGAAGUCCGC .......((((((((((.(((((.((((((((..((((.(((......)))))))...)))))))---).))))))---))(((((((.(.....).))).)))).))))..))).. ( -55.60) >DroPer_CAF1 29162 105 + 1 CCCGCUGA---CAUGCGUGGUCAGCGACAGUGAGGGCAUCUGAGCCUGCAGCGUGCCCUG-UGGC---CCU--CCC---ACAUUGGCCUCGUAUGGCGGUGGUGCACUGAAAUCGGC .((((((.---((((((.((((((........((((((((((......))).)))))))(-(((.---...--.))---)).)))))).)))))).))))))....(((....))). ( -45.60) >consensus CUGACUGGGAUCGGGUGUGGCCAGGGCCAGCGGUGGCAUCUGAGCCGGCAGUGCCACCUGCUGGC___CAUGGCCC___ACGUUCGCCUCGUACGGUGGCGGAGCACUGAAGUCCGC .....((((....((((((((((..(((......)))......((((((((......)))))))).....))))).....((.(((((......)))))))..)))))....)))). (-29.75 = -31.53 + 1.78)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:25:53 2006