
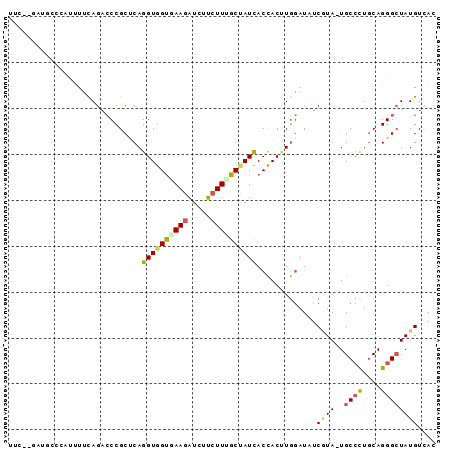
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 1,957,283 – 1,957,412 |
| Length | 129 |
| Max. P | 0.745690 |

| Location | 1,957,283 – 1,957,375 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 93 |
| Reading direction | forward |
| Mean pairwise identity | 77.22 |
| Mean single sequence MFE | -24.85 |
| Consensus MFE | -14.61 |
| Energy contribution | -14.70 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.552069 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
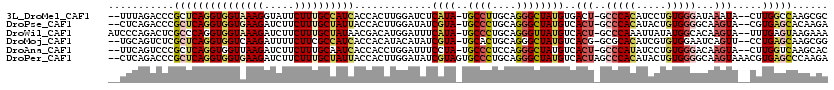
>3L_DroMel_CAF1 1957283 92 + 23771897 UGUUGAAUUCCGCCUUUUAGACCCGCUCAGGUGGUAAAGGUAUUCUUUGCCAUCACCACUUGGAUCUCAUA-UGCCUUGCAGGGCUAUGUGAC ..........................(((((((((...((((.....))))...)))))))))...(((((-(((((....)))).)))))). ( -26.30) >DroPse_CAF1 8235 90 + 1 UUC--UGUGUGCAUUCUCAGACCCGCUCAGGUGGUGAAGAUCUUCUUUGCUAUUACCACUUGGAUAUCGUA-UGCCCUGCAGGGCUAUGUCAC .((--((.(......).)))).....(((((((((((((..........)).)))))))))))....((((-.((((....)))))))).... ( -25.20) >DroGri_CAF1 7493 90 + 1 UUU--GUUGCUAUUUUGCAGAGCCGCCCAGGUGGCGAAAAUCUUCUUCGCUAUCACCACAUACAUAUCGUA-UGCGCUGCAGGGAUAUGUCAC ...--((.((((((((((((.((......(((((((((.......))))))))).....((((.....)))-))).))))).))))).)).)) ( -24.30) >DroEre_CAF1 8438 90 + 1 UUU--GAGACCCCUCUUCAGACCCGCGCAGGUGGUAAAGGUGUUCUUCGCCAUCACUACCUGGAUCUCAUA-UGCCUUGCAGGGCUAUGUGAC (((--((((....)).)))))......((((((((...((((.....))))...))))))))....(((((-(((((....)))).)))))). ( -28.80) >DroAna_CAF1 6955 87 + 1 UUC--C---UCCUCUUUCAGUCCCGCUCAGGUGGUUAAGAUCUUCUUUGCAAUCACCACCUGGAUUUCCUA-UGCCCUCCAGGGCUAUGUCAC ...--.---.................(((((((((.(((.....))).......)))))))))........-.((((....))))........ ( -20.00) >DroPer_CAF1 8956 91 + 1 UUC--UGUGUGCAUUCUCAGACCCGCUCAGGUGGUGAAGAUCUUCUUUGCUAUUACCACUUGGAUAUCGUAGUGCCCUGCAGGGCUAUGUCAC .((--((.(......).))))......((((((((((((..........)).))))))))))(((((......((((....)))).))))).. ( -24.50) >consensus UUC__GAUGCCCAUUUUCAGACCCGCUCAGGUGGUGAAGAUCUUCUUUGCUAUCACCACUUGGAUAUCGUA_UGCCCUGCAGGGCUAUGUCAC .............................((((((((((.....)))))))))).............((((..((((....)))))))).... (-14.61 = -14.70 + 0.09)


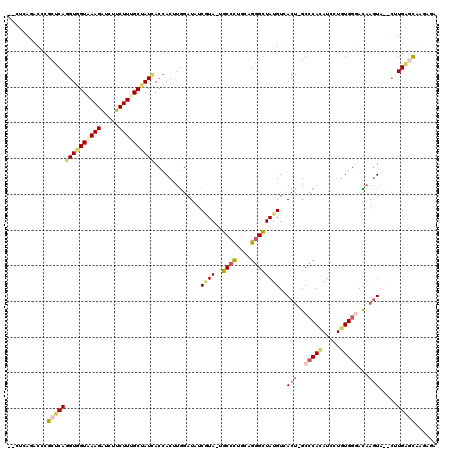
| Location | 1,957,298 – 1,957,412 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.62 |
| Mean single sequence MFE | -35.40 |
| Consensus MFE | -23.50 |
| Energy contribution | -24.70 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.46 |
| SVM RNA-class probability | 0.745690 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
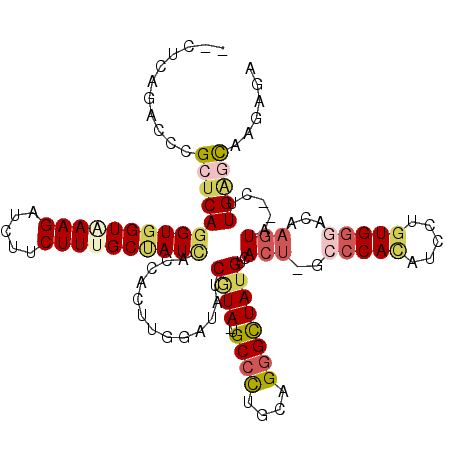
>3L_DroMel_CAF1 1957298 114 + 23771897 --UUUAGACCCGCUCAGGUGGUAAAGGUAUUCUUUGCCAUCACCACUUGGAUCUCAUA-UGCCUUGCAGGGCUAUGUGACU-GCCCACAUCCUGUGGGAUAAAUA--CUUGGCCAAGCGC --........(((...((((((((((.....)))))))))).....((((.(((((((-(((((....)))).))))))..-.(((((.....))))).......--...))))))))). ( -38.60) >DroPse_CAF1 8248 114 + 1 --CUCAGACCCGCUCAGGUGGUGAAGAUCUUCUUUGCUAUUACCACUUGGAUAUCGUA-UGCCCUGCAGGGCUAUGUCACU-GCCCACAUACUGUGGGGCAAGUA--CGUGAGCACAAGA --.........(((((.((((..(((.....)))..))......((((((((((....-.((((....)))).)))))...-.(((((.....))))).))))))--).)))))...... ( -39.90) >DroWil_CAF1 10208 116 + 1 AUCCCAGACUCGCCCAGGUGGUAAAGAUCUUCUUUGCUAUAACGACAUGGAUUUCAUA-UGCCCUGCAGGGUUAUGUCACU-GCCCAAAUUAUAUGGCACAAGUA--UUUGAGUAAGAAA .......((((......(((((((((.....))))))))).((((((((.........-.((((....))))))))))..(-(((..........))))...)).--...))))...... ( -26.70) >DroMoj_CAF1 8088 114 + 1 --UGCAGUCUCGCUCAGGUGGUCAAGAUUUUCUUCGCCAUCACCACAUACAUAUCGUA-UGCACUGCAGGGCUAUGUCACG-GCGCACAUCGUGUGGAAUCAGUU--CCUGAGCAAGCGG --....(.((.(((((((((((.(((.....))).))))...((((((((.....)).-(((.(((...(((...))).))-).)))....))))))........--))))))).))).. ( -33.50) >DroAna_CAF1 6965 114 + 1 --UUCAGUCCCGCUCAGGUGGUUAAGAUCUUCUUUGCAAUCACCACCUGGAUUUCCUA-UGCCCUCCAGGGCUAUGUCACU-GCCCAUAUCCUGUGGGACAAGUA--CUUGGUCAAGCAC --....((((((((((((((((.(((.....))).......)))))))))........-.........((((.........-)))).......))))))).....--............. ( -33.80) >DroPer_CAF1 8969 118 + 1 --CUCAGACCCGCUCAGGUGGUGAAGAUCUUCUUUGCUAUUACCACUUGGAUAUCGUAGUGCCCUGCAGGGCUAUGUCACUAGCCCACAUACUGUGGGGCAAGUAAACGUGAGCCCAAGA --.........((((((((((..(((.....)))..))))).((....))....(((..(((((..((((((((......))))))......))..))))).....))))))))...... ( -39.90) >consensus __CUCAGACCCGCUCAGGUGGUAAAGAUCUUCUUUGCUAUCACCACUUGGAUAUCGUA_UGCCCUGCAGGGCUAUGUCACU_GCCCACAUCCUGUGGGACAAGUA__CUUGAGCAAGAGA ...........(((((((((((((((.....)))))))))).............((((..((((....))))))))..(((..(((((.....)))))...))).....)))))...... (-23.50 = -24.70 + 1.20)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:47:51 2006