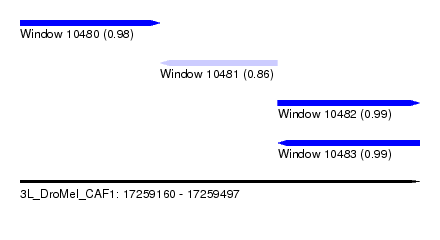
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 17,259,160 – 17,259,497 |
| Length | 337 |
| Max. P | 0.991964 |

| Location | 17,259,160 – 17,259,278 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.96 |
| Mean single sequence MFE | -35.20 |
| Consensus MFE | -29.52 |
| Energy contribution | -29.44 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.978951 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 17259160 118 + 23771897 GCUGGUAUUGUUUGCGCUGAGCAAUAAACAAC--AUUUCGUUGACUUUGGCAUUUAAAAUUUUAUGCGCAAUUAGGAGUGCAAGAUGAAUGUGUUGAAUGGCUGCCGGCGCAGUAAUUAC ....(((....((((((((.(((.....((((--.....)))).....(.(((((((.((((..(((((........)))))....))))...))))))).))))))))))))....))) ( -33.70) >DroSec_CAF1 142820 118 + 1 GCUGGUAUUGUUUGCGCUGAGCAAUAAACAAC--AUUUCGUUAACUUUGGCAUUUAAAAUGUUAUGCGCAAUUAGGAGUGCAAGAUGAAUGUGUUGAAUGGCUGCCGGCGCAGUAAUUAC ....(((....((((((((.(((.((..((((--((((((((.....((((((.....))))))(((((........))))).)))))).))))))..))..)))))))))))....))) ( -35.30) >DroSim_CAF1 137686 118 + 1 GCUGGUAUUGUUUGCGCUGAGCAAUAAACAAC--AUUUCGUUGACUUUGGCAUUUAAAAUGUUAUGCGCAAUUAGGAGUGCAAGAUGAAUGUGUUGAAUGGCUGCCGGCGCAGUAAUUAC ....(((....((((((((.(((.((..((((--((((((((.....((((((.....))))))(((((........))))).)))))).))))))..))..)))))))))))....))) ( -35.30) >DroEre_CAF1 145890 117 + 1 -CUGGCAUUGUUUGCGUCGAGCAAUAAACAAC--AUUUCGUUGACUUUGGCAUUUAAAAUGUUAUGCGCAAUUAGGAGUGCAAGAUGAAUGUGUUGAACGGCUGCCGACGCAGUAAUUAC -..........((((((((.(((.....((((--((((((((.....((((((.....))))))(((((........))))).)))))).))))))......)))))))))))....... ( -33.90) >DroYak_CAF1 150080 119 + 1 -CUGGCAUUGUUUGCGCUGAGCCAUAAACAACAAAUUUCGUUGACUUUGGCAUUUAAAAUGUUAUGCGCAAUUAGGAGUGCAAGAUGAAUGUGUUGAAUGGCUGCCGGCGCAGUAAUUAC -..........((((((((((((((...(((((...((((((.....((((((.....))))))(((((........))))).))))))..))))).))))))..))))))))....... ( -37.80) >consensus GCUGGUAUUGUUUGCGCUGAGCAAUAAACAAC__AUUUCGUUGACUUUGGCAUUUAAAAUGUUAUGCGCAAUUAGGAGUGCAAGAUGAAUGUGUUGAAUGGCUGCCGGCGCAGUAAUUAC ...........((((((((.(((.....((((....((((((.....((((((.....))))))(((((........))))).))))))...))))......)))))))))))....... (-29.52 = -29.44 + -0.08)



| Location | 17,259,278 – 17,259,377 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.14 |
| Mean single sequence MFE | -27.56 |
| Consensus MFE | -24.18 |
| Energy contribution | -24.18 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.858035 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 17259278 99 - 23771897 UUGGUAAAUCAGCAAAAUAAUUUCGCUUGUUUUGCU--------------GCUU-------GCAGAUUGCCAUUUCAUUUGGGUUUUCAUUAUGCGUGGUGCGUCACGUGUGCAGAGUUU .........(((((((((((......))))))))))--------------).((-------((.((..(((..........)))..))...((((((((....))))))))))))..... ( -25.90) >DroSec_CAF1 142938 99 - 1 UUGGUAAAUCAGCAAAAUAAUUUCGCUUGUUUUGCU--------------GCUU-------GCAGAUUGCCAUUUCAUUCGGAUUUUCAUUAUGCGUGGUGCGUCACGUGUGCAGAGUUU .((((((.((((((((((((......))))))))))--------------(...-------.).))))))))........(((((((...(((((((((....))))))))).))))))) ( -23.60) >DroSim_CAF1 137804 99 - 1 UUGGUAAAUCAGCAAAAUAAUUUCGCUUGUUUUGCU--------------GCUU-------GCAGAUUGCCAUUUCAUUCGGAUUUUCAUUAUGCGUGGUGCGUCACGUGUGCAGAGUUU .((((((.((((((((((((......))))))))))--------------(...-------.).))))))))........(((((((...(((((((((....))))))))).))))))) ( -23.60) >DroEre_CAF1 146007 106 - 1 UUGGUAAAUCAGCAAAAUAAUUUCGCUUGUUUUGCU--------------GCUUGCAGAUUGCAGAUUGCCAUUUCAUUCGGAUUUUCAUUAUGCGUGGUGCGUCACGUGUGCAGAGUUU .((((((..(((((((((((......))))))))))--------------).((((.....)))).))))))........(((((((...(((((((((....))))))))).))))))) ( -28.90) >DroYak_CAF1 150199 120 - 1 UUGGUAAAUCAGCAAAAUAAUUUCGCUUGUUUUGCUGCUUGCUGCUUGCUGCUUGCAGAUUGCAGAUUGCCAUUUCAUUUGGAUUUUCAUUAUGCGUGGUGCGUCACGUGUGCAGAGUUU .((((((..(((((((((((......)))))))))))....((((...(((....)))...)))).))))))........(((((((...(((((((((....))))))))).))))))) ( -35.80) >consensus UUGGUAAAUCAGCAAAAUAAUUUCGCUUGUUUUGCU______________GCUU_______GCAGAUUGCCAUUUCAUUCGGAUUUUCAUUAUGCGUGGUGCGUCACGUGUGCAGAGUUU .((((((.((((((((((((......))))))))))..............((.........)).)))))))).............(((..(((((((((....)))))))))..)))... (-24.18 = -24.18 + -0.00)

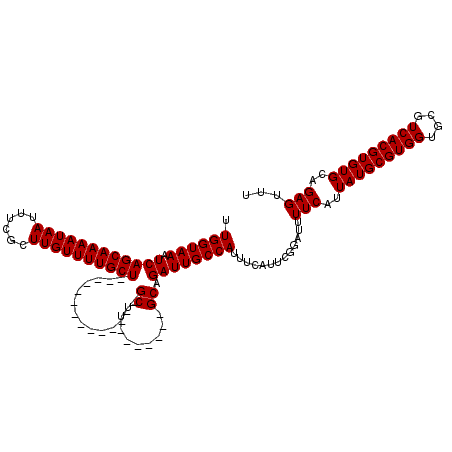

| Location | 17,259,377 – 17,259,497 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.83 |
| Mean single sequence MFE | -36.78 |
| Consensus MFE | -35.10 |
| Energy contribution | -35.50 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.95 |
| SVM decision value | 2.10 |
| SVM RNA-class probability | 0.988069 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 17259377 120 + 23771897 AGACAAACAUAAUUGAAUUACGGCAAGAGCGCACACGGACAUGGUUAUGAACACGUACACGUAUACGGACAAGGACUACGGUGCGUCUACGUAACGUGCUCCUUAGUGUUUGUGUGCACU ......................((....))((((((((((((.(((......(((....))).....)))(((((.((((.((((....)))).)))).))))).))))))))))))... ( -36.70) >DroSec_CAF1 143037 120 + 1 AGACAAACAUAAUUGAAUUACGGAAAGAGCGCACACGGACAUGGUUAUGAACACGGACACGUAUACGGACAAGGUCUACGGUGCGGCUACGUAACGUGCUCCUUAGUGUUUGUGUGCACU ...........................((.((((((((((((.(((......(((....))).....)))((((..((((.((((....)))).))))..)))).)))))))))))).)) ( -35.60) >DroSim_CAF1 137903 120 + 1 AGACAAACAUAAUUGAAUUACGGAAAGAGCGCACACGGACAUGGUUAUGAACACGGACACGUAUACGGACAAGGACUACGGUGCGGCUACGUAACGUGCUCCUUAGUGUUUGUGUGCACU ...........................((.((((((((((((.(((......(((....))).....)))(((((.((((.((((....)))).)))).))))).)))))))))))).)) ( -37.10) >DroEre_CAF1 146113 120 + 1 AGACAAACAUAAUUGAAUUACGGAAAGAGCGCACACGGACAUGGUUAUGAACACGGACACGUAUCCGGACAAGGACUACGGUGCGGCUACGUAACGUGCUCCUUAGUGUUUGUGUGCACU ...........................((.((((((((((((...........((((......))))...(((((.((((.((((....)))).)))).))))).)))))))))))).)) ( -38.50) >DroYak_CAF1 150319 120 + 1 AGACAAACAUAAUUGAAUUACGGAAAGAGCGCACACGGACAUGGUUAUGAACACGGACACGUAUCCGGACAAGGACUACGGUGCGGCUACGUAACAUGCUCCUUAGUGUUUGUGUGCACU ...........................((.((((((((((((.((((((....((.((.(((((((......))).)))))).))....))))))..........)))))))))))).)) ( -36.00) >consensus AGACAAACAUAAUUGAAUUACGGAAAGAGCGCACACGGACAUGGUUAUGAACACGGACACGUAUACGGACAAGGACUACGGUGCGGCUACGUAACGUGCUCCUUAGUGUUUGUGUGCACU ...........................((.((((((((((((.(((......(((....))).....)))(((((.((((.((((....)))).)))).))))).)))))))))))).)) (-35.10 = -35.50 + 0.40)

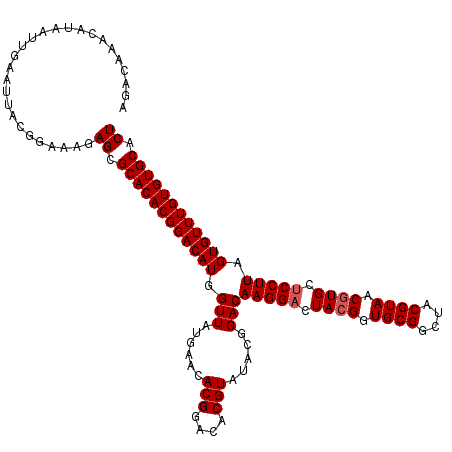
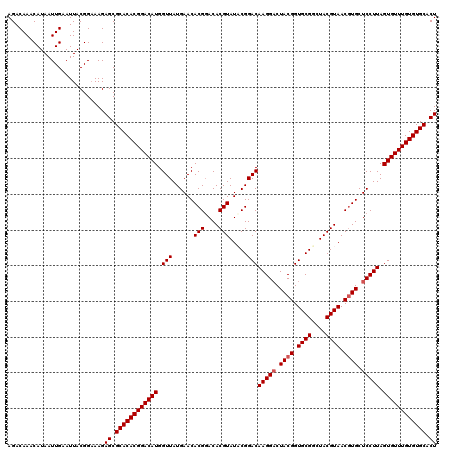
| Location | 17,259,377 – 17,259,497 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.83 |
| Mean single sequence MFE | -34.88 |
| Consensus MFE | -33.50 |
| Energy contribution | -33.58 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.30 |
| SVM RNA-class probability | 0.991964 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 17259377 120 - 23771897 AGUGCACACAAACACUAAGGAGCACGUUACGUAGACGCACCGUAGUCCUUGUCCGUAUACGUGUACGUGUUCAUAACCAUGUCCGUGUGCGCUCUUGCCGUAAUUCAAUUAUGUUUGUCU (((((((((..(((.....((((((((((((((.(((.((..........)).))).)))))).)))))))).......)))..))))))))).....((((((...))))))....... ( -36.80) >DroSec_CAF1 143037 120 - 1 AGUGCACACAAACACUAAGGAGCACGUUACGUAGCCGCACCGUAGACCUUGUCCGUAUACGUGUCCGUGUUCAUAACCAUGUCCGUGUGCGCUCUUUCCGUAAUUCAAUUAUGUUUGUCU (((((((((..(((.....(((((((.((((((..((.((..........)).))..))))))..))))))).......)))..))))))))).....((((((...))))))....... ( -33.40) >DroSim_CAF1 137903 120 - 1 AGUGCACACAAACACUAAGGAGCACGUUACGUAGCCGCACCGUAGUCCUUGUCCGUAUACGUGUCCGUGUUCAUAACCAUGUCCGUGUGCGCUCUUUCCGUAAUUCAAUUAUGUUUGUCU (((((((((..(((.....(((((((.((((((..((.((..........)).))..))))))..))))))).......)))..))))))))).....((((((...))))))....... ( -33.40) >DroEre_CAF1 146113 120 - 1 AGUGCACACAAACACUAAGGAGCACGUUACGUAGCCGCACCGUAGUCCUUGUCCGGAUACGUGUCCGUGUUCAUAACCAUGUCCGUGUGCGCUCUUUCCGUAAUUCAAUUAUGUUUGUCU (((((((((..(((.....(((((((.((((((.(((.((..........)).))).))))))..))))))).......)))..))))))))).....((((((...))))))....... ( -36.30) >DroYak_CAF1 150319 120 - 1 AGUGCACACAAACACUAAGGAGCAUGUUACGUAGCCGCACCGUAGUCCUUGUCCGGAUACGUGUCCGUGUUCAUAACCAUGUCCGUGUGCGCUCUUUCCGUAAUUCAAUUAUGUUUGUCU (((((((((..(((.....(((((((.((((((.(((.((..........)).))).))))))..))))))).......)))..))))))))).....((((((...))))))....... ( -34.50) >consensus AGUGCACACAAACACUAAGGAGCACGUUACGUAGCCGCACCGUAGUCCUUGUCCGUAUACGUGUCCGUGUUCAUAACCAUGUCCGUGUGCGCUCUUUCCGUAAUUCAAUUAUGUUUGUCU (((((((((..(((.....(((((((.((((((.(((.((..........)).))).))))))..))))))).......)))..))))))))).....((((((...))))))....... (-33.50 = -33.58 + 0.08)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:21:13 2006