
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
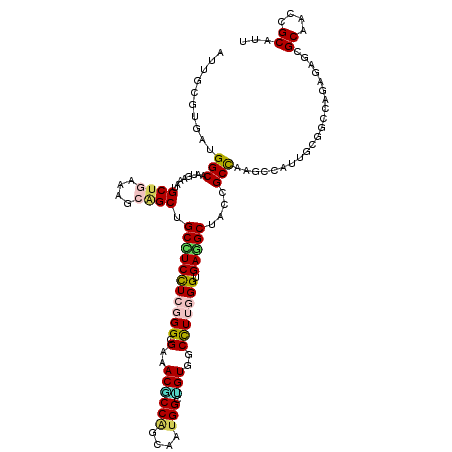
| Location | 16,894,051 – 16,894,171 |
| Length | 120 |
| Max. P | 0.935778 |

| Location | 16,894,051 – 16,894,171 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.50 |
| Mean single sequence MFE | -52.65 |
| Consensus MFE | -25.99 |
| Energy contribution | -26.38 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.49 |
| SVM decision value | 1.25 |
| SVM RNA-class probability | 0.935778 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16894051 120 + 23771897 AUUGCGAGUGGGCAAUGAAAUGCUGAAGGCAGCUGCCUCCUCGGGAGAAACUCCAGCAAUGGCUGUGGCCUUGGGCGAAGCUACGGCCAAUCCAUUCCGGCCGGAGAGAGCCACAGCGUU ((((((((..((((.......((((....))))))))..))).((((...)))).))))).(((((((((((.(((...))).(((((..........)))))..))).))))))))... ( -52.61) >DroVir_CAF1 7597 120 + 1 GUUGCGUGAUGGCAGCGUUAUGCUAAAUGCCGCUGCUUCCUCGGGCGUUACGCCGGCAAUGGCUGUUGCCUUGGGUGAGGCCACCGCCAGGCCGUUGCGUCCAGAGAGUGCGACCGCAUU ((((((.((.(((((((.(((.....))).)))))))))(((((((((.((((((((..(((((.(..((...))..))))))..))).)).))).)))))).)))..))))))...... ( -53.40) >DroGri_CAF1 6946 120 + 1 AUUGCGUGAUGGCAGUGUUAUGCUAAACGCAGCAGCUUCUUCUGGCGACACGCCAGCGAUGGCCGUUGCCUUAGGUGAAGCCACCGCCAAUCCAUUGCGUCCAGAGAGCGCAAUCGCAUU ..((((...((((.(((...((((......))))(((((.(((((((((..((((....)))).))))))..))).)))))))).))))....(((((((.......))))))))))).. ( -46.30) >DroWil_CAF1 6739 120 + 1 AUUGCGGGUGGGCAAUGAGAUGCUGAAAGCGGCGGCUUCCUCCGGAGAGACACCAGCAAUGGCUGUGGCCUUGGGUGAGGCAACUGCCAGACCAUUGCGCCCAGAAAGGGCAACUGCAUU ..(((((....((((((.(...(((...((((..(((((.((((..(..((((((....))).)))..)..)))).)))))..))))))).)))))))((((.....))))..))))).. ( -50.30) >DroMoj_CAF1 7822 120 + 1 GUUGCGUGAUGGCAGCGUAAUGCUGAACGCAGCCGCCUCCUCGGGCGUAACGCCCGCAAUGGCCGUGGCUUUGGGUGAGGCUACAGCCAGGCCAUUGCGGCCCGAGAGCGCAACGGCAUU (((((((...(((.((((........)))).))).....((((((((.....).((((((((((.(((((.(((......))).)))))))))))))))))))))).)))))))...... ( -64.60) >DroAna_CAF1 6088 120 + 1 AUUCCUGGAAGGCAAGGAAAUGCUGAAAGCAGCUGCCUCCUCGGGUGAGACUCCGGCAAUGGCAGUGGCUUUUGGAGAGGCUACGGCUAAGCCAUUACGGCCGGAAAGGGCCACGGCAUU .(((((........)))))(((((((((((.((((((...(((((......)))))....)))))).)))))(((...(((....)))...)))....((((......)))).)))))). ( -48.70) >consensus AUUGCGUGAUGGCAAUGAAAUGCUGAAAGCAGCUGCCUCCUCGGGCGAAACGCCAGCAAUGGCUGUGGCCUUGGGUGAGGCUACCGCCAAGCCAUUGCGGCCAGAGAGCGCAACCGCAUU (((((......)))))....(((.....)))((((((((.((.((((...)))).((((((((.((.(((((....))))).)).)))).....)))).....))))).))...)))... (-25.99 = -26.38 + 0.39)


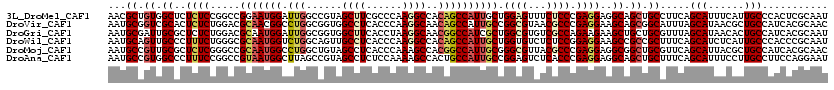
| Location | 16,894,051 – 16,894,171 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.50 |
| Mean single sequence MFE | -48.23 |
| Consensus MFE | -24.39 |
| Energy contribution | -25.25 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.39 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.19 |
| SVM RNA-class probability | 0.928951 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16894051 120 - 23771897 AACGCUGUGGCUCUCUCCGGCCGGAAUGGAUUGGCCGUAGCUUCGCCCAAGGCCACAGCCAUUGCUGGAGUUUCUCCCGAGGAGGCAGCUGCCUUCAGCAUUUCAUUGCCCACUCGCAAU ...((((((((.((((.(((((((......))))))).)).........)))))))))).(((((.((((...)))).(((..((((((((....)))).......))))..)))))))) ( -46.21) >DroVir_CAF1 7597 120 - 1 AAUGCGGUCGCACUCUCUGGACGCAACGGCCUGGCGGUGGCCUCACCCAAGGCAACAGCCAUUGCCGGCGUAACGCCCGAGGAAGCAGCGGCAUUUAGCAUAACGCUGCCAUCACGCAAC ..((((((.((..((((.((((((..((((.((((.((.((((......)))).)).))))..))))))))....)).))))..)).))((((....((.....))))))....)))).. ( -45.50) >DroGri_CAF1 6946 120 - 1 AAUGCGAUUGCGCUCUCUGGACGCAAUGGAUUGGCGGUGGCUUCACCUAAGGCAACGGCCAUCGCUGGCGUGUCGCCAGAAGAAGCUGCUGCGUUUAGCAUAACACUGCCAUCACGCAAU ..((((((((((.((....)))))))).(((.((((((((((((......(((....)))....((((((...))))))..)))))(((((....)))))...)))))))))).)))).. ( -52.20) >DroWil_CAF1 6739 120 - 1 AAUGCAGUUGCCCUUUCUGGGCGCAAUGGUCUGGCAGUUGCCUCACCCAAGGCCACAGCCAUUGCUGGUGUCUCUCCGGAGGAAGCCGCCGCUUUCAGCAUCUCAUUGCCCACCCGCAAU ..(((....((((.....))))(((((((...(((.(((.((((.....((((..((((....))))..)))).....)))).))).)))((.....))...)))))))......))).. ( -45.10) >DroMoj_CAF1 7822 120 - 1 AAUGCCGUUGCGCUCUCGGGCCGCAAUGGCCUGGCUGUAGCCUCACCCAAAGCCACGGCCAUUGCGGGCGUUACGCCCGAGGAGGCGGCUGCGUUCAGCAUUACGCUGCCAUCACGCAAC ......((((((..((((((((((((((((((((((..............))))).))))))))))(......))))))))(((((((((((.....)))....)))))).)).)))))) ( -62.44) >DroAna_CAF1 6088 120 - 1 AAUGCCGUGGCCCUUUCCGGCCGUAAUGGCUUAGCCGUAGCCUCUCCAAAAGCCACUGCCAUUGCCGGAGUCUCACCCGAGGAGGCAGCUGCUUUCAGCAUUUCCUUGCCUUCCAGGAAU ......((((....(((((((.(((.((((((.................)))))).)))....)))))))..))))((..(((((((((((....)))).......)))))))..))... ( -37.94) >consensus AAUGCCGUUGCCCUCUCUGGCCGCAAUGGCCUGGCCGUAGCCUCACCCAAGGCCACAGCCAUUGCUGGCGUCUCGCCCGAGGAAGCAGCUGCGUUCAGCAUUACACUGCCAUCACGCAAU ...((.((.((..((((.....(((((((.(((......((((......))))..)))))))))).((((...)))).))))..)).)).)).....(((......)))........... (-24.39 = -25.25 + 0.86)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:15:39 2006