
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 16,775,845 – 16,776,023 |
| Length | 178 |
| Max. P | 0.981727 |

| Location | 16,775,845 – 16,775,964 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.11 |
| Mean single sequence MFE | -48.88 |
| Consensus MFE | -30.94 |
| Energy contribution | -30.37 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.833093 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16775845 119 - 23771897 AAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCCGCUGUUCUGGCGGCUGCCAAGAGGGGAUCGAGGAGCUCCCAGGGCUCCAGCGACAGUAACAACAGCACGCGGGUGAGU-AA .....(((..((((((..((.((((..((....(((((((((.....)))))))))..............((((((.....)))))).))..)))).))..))))))....)))...-.. ( -49.40) >DroVir_CAF1 24008 119 - 1 AAUGCGACCAGCGCGGCGGUUACCGCAGCUGCUGCGGCGGCUGUGCUGGCGGCUGCCAAGCGUGGAUCACGCUCCUCGCAGGGCUCCAGCGACAGCAACAACAGCACACGGGUGAGU-AA ..(((.((((((((((......)))).)))((((..(..((((((((((.....(((..(((.(((......))).)))..))).))))).)))))..)..)))).....)))..))-). ( -54.00) >DroGri_CAF1 24716 120 - 1 AAUGCCACAAGUGCAGCAGUUACAGCUGCUGCGGCGGCAGCAGUGCUGGCUGCUGCCAAGCGAGGAUCACGCUCCUCCCAGGGCUCCAGCGAUAGCAACAAUAGCACAAGGGUAGGUCAA ..((((....((((((((((((..((((((((....))))))))..))))))))(((..(.(((((......))))).)..)))....((....)).......))))...))))...... ( -50.00) >DroWil_CAF1 62886 119 - 1 AAUGCCACAAGUGCAGCUGUGACAGCAGCAGCAGCCGCUGCUGUACUUGCAGCAGCCAAACGUGGUUCACGCUCUUCCCAAGGAUCCAGUGAUAGCAAUAAUAGCACACGGGUAAGU-UA ..((((....((((.((((..(..((((((((....))))))))..)..))))........((...((((.....(((...)))....))))..)).......))))...))))...-.. ( -42.20) >DroMoj_CAF1 24522 119 - 1 AAUGGAACUAGCGCAGCUGUCACAGCGGCUGCUGCGGCGGCUGUCUUGGCGGCUGCCAAGCGCGGAUCACGCUCGUCACAGGGCUCCAGCGACAGCAACAACAGCACACGGGUGAGU-AG ......(((.((((((((((((..((((((((....))))))))..))))))))))...(((((((....((((......))))))).))).).))........(((....))))))-.. ( -48.30) >DroAna_CAF1 24691 118 - 1 -AUGCGACAAGUGCUGCUGUGACAGCGGCAGCGGCGGCGGCAGUUCUGGCGGCUGCCAAAAGGGGAUCACGGAGCUCUCAGGGCUCCAGCGAUAGCAACAACAGCACACGGGUGAGU-AA -.(((.((..(((.((((((....((....((((((((.((.......)).)))))).............((((((.....)))))).))....))....)))))))))..))..))-). ( -49.40) >consensus AAUGCCACAAGUGCAGCUGUGACAGCGGCAGCAGCGGCGGCUGUGCUGGCGGCUGCCAAGCGGGGAUCACGCUCCUCCCAGGGCUCCAGCGACAGCAACAACAGCACACGGGUGAGU_AA ..(((.......((((((((....)))))))).(((((((((((....)))))))))......(((((..(.......)..)).))).))....)))....((.(.....).))...... (-30.94 = -30.37 + -0.58)

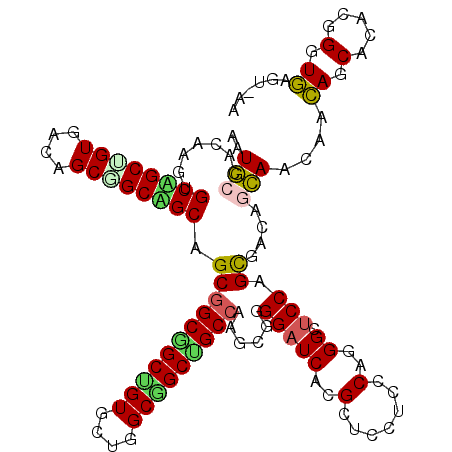

| Location | 16,775,884 – 16,775,993 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 72.82 |
| Mean single sequence MFE | -40.27 |
| Consensus MFE | -26.93 |
| Energy contribution | -26.77 |
| Covariance contribution | -0.17 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.81 |
| SVM RNA-class probability | 0.978310 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16775884 109 - 23771897 CCAGUAACACUAGCAGCAUUCACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCCGCUGUUCUGGCGGCUGCCAAGAGGGGAUCGAGGAGCUCCCA .((((...(((.((((((((..................))))))))))).)))).((.(((.(((((((((.....)))))))))...((.....)).....))).)). ( -38.17) >DroVir_CAF1 24047 104 - 1 -----AACGUUUCGGAUGCGACCAAUUCGAAUGCGACCAGCGCGGCGGUUACCGCAGCUGCUGCGGCGGCUGUGCUGGCGGCUGCCAAGCGUGGAUCACGCUCCUCGCA -----.....(((((((.......)))))))(((((...(.(((((.((((.(((((((((....))))))))).)))).)))))).((((((...))))))..))))) ( -48.00) >DroSec_CAF1 24704 109 - 1 CCAGUAACAGUGGCAGCAUUAACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCGGCUGUUCUGGCGGCAGCCAAGAGGGGAUCGAGGAGCUCCCA ((((.((((((.((((((((..................)))))))).((..((((....))))..)).)))))))))).((.((((..((.....))...).))).)). ( -37.27) >DroEre_CAF1 25123 109 - 1 CCAGUAACAGUAGCAACAUUAACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCGGCUGUUCUGGCUGCUGCCAAGAGGGGAUCGAGGAGUUCCCA .((((.((.((((((..((((..........)))).....)))))).)).)))).(((((((((..(((.....))))))))))))....((((((......)))))). ( -37.70) >DroMoj_CAF1 24561 101 - 1 -----AUCCACUCC---ACAACCAAUUCGAAUGGAACUAGCGCAGCUGUCACAGCGGCUGCUGCGGCGGCUGUCUUGGCGGCUGCCAAGCGCGGAUCACGCUCGUCACA -----.((((.((.---...........)).))))....((((((((((....)))))))).))(((((((((....)))))))))..(.(((.....))).)...... ( -41.00) >DroAna_CAF1 24730 106 - 1 CCAGCAACAUCGCCAGCAGCAACAAUA---AUGCGACAAGUGCUGCUGUGACAGCGGCAGCGGCGGCGGCAGUUCUGGCGGCUGCCAAAAGGGGAUCACGGAGCUCUCA ..(((..((((.((.((.(((......---.)))......((((((((...))))))))))((((((.((.......)).))))))....)).)))...)..))).... ( -39.50) >consensus CCAGUAACAGUACCAGCAUUAACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCGGCUGUUCUGGCGGCUGCCAAGAGGGGAUCAAGGAGCUCCCA .......................................(((((((.((....)).))))).))(((((((((....)))))))))....((((..........)))). (-26.93 = -26.77 + -0.17)



| Location | 16,775,924 – 16,776,023 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 77.51 |
| Mean single sequence MFE | -37.63 |
| Consensus MFE | -20.20 |
| Energy contribution | -20.82 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.824374 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16775924 99 + 23771897 CGGCCGCUGCUGCCGCAGUUACUGCAGCACUUGUAUUAUUGUUAUUGUGAAUGCUGCUAGUGUUACUGGGCGAACUGCGUUGGUUUAGGGUAGGUGGAU ...(((((((((((.((((((((((((((.(..((.((....)).))..).)))))).))))..)))))))(((((.....)))))..)))).)))).. ( -35.10) >DroSec_CAF1 24744 99 + 1 CCGCCGCUGCUGCCGCAGUUACUGCAGCACUUGUAUUAUUGUUAUUGUUAAUGCUGCCACUGUUACUGGGCGAACUGCGCUGGUUUAGGGUAGGCGGAU (((((..((((((.(......).))))))...((((((..........))))))((((.(((..((..(((.....)).)..)).)))))))))))).. ( -33.40) >DroEre_CAF1 25163 99 + 1 CCGCCGCUGCUGCCGCAGUUACUGCAGCACUUGUAUUAUUGUUAUUGUUAAUGUUGCUACUGUUACUGGGCGAACUGCGCUGGUUCAGUGUGGGCGGAU (((((((.((((((.((((.((.((((((.(..((.((....)).))..).))))))....)).)))))))(((((.....)))))))))).))))).. ( -33.50) >DroYak_CAF1 24928 99 + 1 CCGCCGCUGCUGCCGCAGUUACAGCAGCACUUGUAUUGUUGUUAUUGUUAAUGCUGCUACUGUUACUGGGCGAACUGCGCUGGUUCAGGGUAGGCGGAU (((((..(((((((.((((.((((.((((...((((((..........)))))))))).)))).)))))))(((((.....)))))..))))))))).. ( -39.30) >DroMoj_CAF1 24601 87 + 1 CCGCCGCAGCAGCCGCUGUGACAGCUGCGCUAGUUCCAUUCGAAUUGGUUGU---GGAGUGGAU---------GCUGCGCUGUGCAAGCGUUGGCGGAU (((((((((((.(((((.(.(((.....((((((((.....)))))))))))---.)))))).)---------)))))(((.....)))...))))).. ( -40.60) >DroAna_CAF1 24770 96 + 1 CCGCCGCCGCUGCCGCUGUCACAGCAGCACUUGUCGCAU---UAUUGUUGCUGCUGGCGAUGUUGCUGGGAGAGCUGCGCUGGUUGAGGGUGGGCGGAU (((((((((.(((.(((.((.((((((((.((((((((.---((....)).))).))))))))))))).)).))).))).))))........))))).. ( -43.90) >consensus CCGCCGCUGCUGCCGCAGUUACAGCAGCACUUGUAUUAUUGUUAUUGUUAAUGCUGCUACUGUUACUGGGCGAACUGCGCUGGUUCAGGGUAGGCGGAU ((((((((((....)))))(((.((((((......................))))))..............(((((.....)))))...)))))))).. (-20.20 = -20.82 + 0.62)

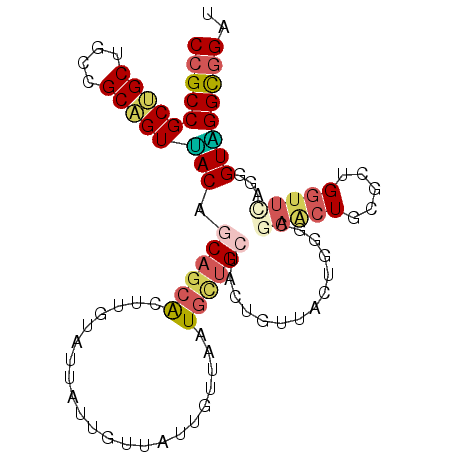

| Location | 16,775,924 – 16,776,023 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 77.51 |
| Mean single sequence MFE | -31.89 |
| Consensus MFE | -16.23 |
| Energy contribution | -16.65 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.90 |
| SVM RNA-class probability | 0.981727 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16775924 99 - 23771897 AUCCACCUACCCUAAACCAACGCAGUUCGCCCAGUAACACUAGCAGCAUUCACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCCG ....................(((.(((.(((((((...(((.((((((((..................))))))))))).)))).)))))).))).... ( -27.17) >DroSec_CAF1 24744 99 - 1 AUCCGCCUACCCUAAACCAGCGCAGUUCGCCCAGUAACAGUGGCAGCAUUAACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCGG ..(((((....((.....)).((.(((.(((((((.((....((((((((..................)))))))).)).)))).)))))).))))))) ( -31.97) >DroEre_CAF1 25163 99 - 1 AUCCGCCCACACUGAACCAGCGCAGUUCGCCCAGUAACAGUAGCAACAUUAACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCGG ..(((((....(((...))).((.(((.(((((((.((.((((((..((((..........)))).....)))))).)).)))).)))))).))))))) ( -30.30) >DroYak_CAF1 24928 99 - 1 AUCCGCCUACCCUGAACCAGCGCAGUUCGCCCAGUAACAGUAGCAGCAUUAACAAUAACAACAAUACAAGUGCUGCUGUAACUGCGGCAGCAGCGGCGG ..(((((....(((...))).((.(((.(((((((.(((((((((.........................))))))))).)))).)))))).))))))) ( -35.41) >DroMoj_CAF1 24601 87 - 1 AUCCGCCAACGCUUGCACAGCGCAGC---------AUCCACUCC---ACAACCAAUUCGAAUGGAACUAGCGCAGCUGUCACAGCGGCUGCUGCGGCGG ..(((((...(((.((.....)))))---------.((((.((.---...........)).))))....((((((((((....)))))))).))))))) ( -33.40) >DroAna_CAF1 24770 96 - 1 AUCCGCCCACCCUCAACCAGCGCAGCUCUCCCAGCAACAUCGCCAGCAGCAACAAUA---AUGCGACAAGUGCUGCUGUGACAGCGGCAGCGGCGGCGG ..(((((.................(((.....)))......(((.(..(((......---.)))..)....((((((((....)))))))))))))))) ( -33.10) >consensus AUCCGCCUACCCUAAACCAGCGCAGUUCGCCCAGUAACAGUAGCAGCAUUAACAAUAACAAUAAUACAAGUGCUGCAGUAACUGCGGCAGCAGCGGCGG ..(((((..............((.....)).......................................(((((((.((....)).))))).))))))) (-16.23 = -16.65 + 0.42)

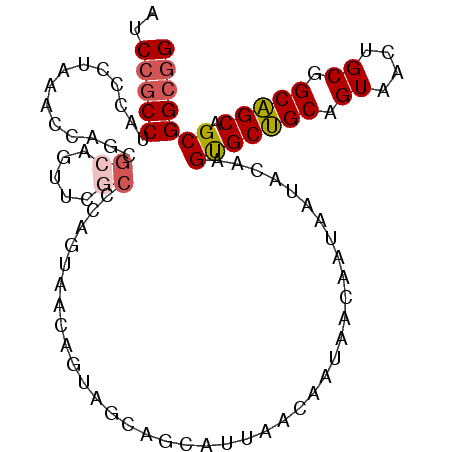

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:15:00 2006