
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 16,681,893 – 16,682,016 |
| Length | 123 |
| Max. P | 0.790487 |

| Location | 16,681,893 – 16,681,989 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 88.08 |
| Mean single sequence MFE | -41.62 |
| Consensus MFE | -34.36 |
| Energy contribution | -34.58 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.790487 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
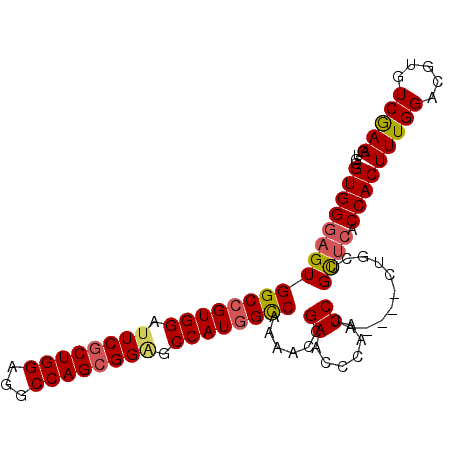
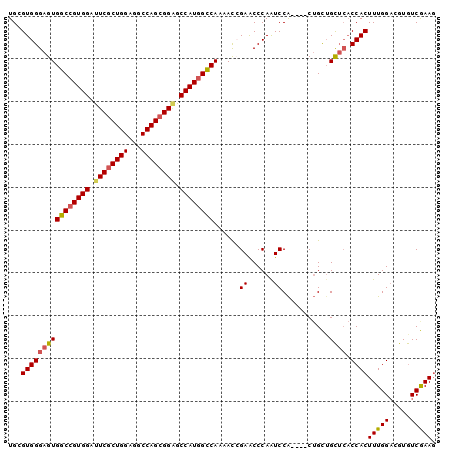
>3L_DroMel_CAF1 16681893 96 - 23771897 UGGGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA----CUGCUGCUCACCACUUUGGACGUGUCGAAG (((((.((..(((((((((.((((((((...)))))))).)))))))))...))..))))).((((----..(.((.....)))..)))).......... ( -43.40) >DroSec_CAF1 3599 96 - 1 UGCGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA----CUGCUGCUCACCACUUUGGACGUGUCGAAG ...((((((((((((((((.((((((((...)))))))).))))))))......((......))..----.....)))).)))).((.((....)).)). ( -39.70) >DroSim_CAF1 3394 96 - 1 UGCGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA----CUGCUGCUCACCACUUUGGACGUGUCGAAG ...((((((((((((((((.((((((((...)))))))).))))))))......((......))..----.....)))).)))).((.((....)).)). ( -39.70) >DroEre_CAF1 3870 96 - 1 UGGGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGGGCCAUGGCCAAAACCGAACCCAAUCCA----CUGUUGCUCACCACUUUGGAAGUGUCAAAU (((((.((..(((((((((.((((((((...)))))))).)))))))))...))..))))).((((----..((........))..)))).......... ( -44.00) >DroYak_CAF1 3163 96 - 1 UGGGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACGGAACCCAAUCCA----CUGUGGCUCACCACUUUGGAAGUGUCAAAG (((((.(...(((((((((.((((((((...)))))))).)))))))))...)...))))).((((----..((((....))))..)))).......... ( -48.60) >DroAna_CAF1 1134 99 - 1 UGCGUGGGCGUGGCCGUGGACUCGCUGGAAGCCAGAGGUGCCAUCGUCAUAAUAGAGCCCAGUCCGGCAGCAGCUGU-GCCCACUUUGGUGGUAUCGAAG ...(((((((..((((((((((..((((...)))).))).))).))..........(((......)))....))..)-))))))((((((...)))))). ( -34.30) >consensus UGCGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA____CUGCUGCUCACCACUUUGGACGUGUCGAAG ...((((((((((((((((.((((((((...)))))))).))))))))......((......))...........)))).))))(((((.....))))). (-34.36 = -34.58 + 0.22)

| Location | 16,681,916 – 16,682,016 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 87.24 |
| Mean single sequence MFE | -45.45 |
| Consensus MFE | -34.45 |
| Energy contribution | -35.32 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.17 |
| SVM RNA-class probability | 0.614886 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
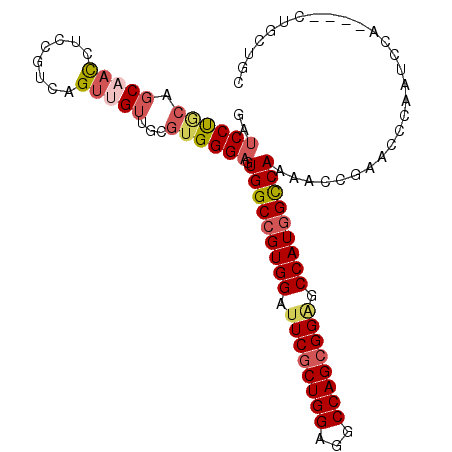
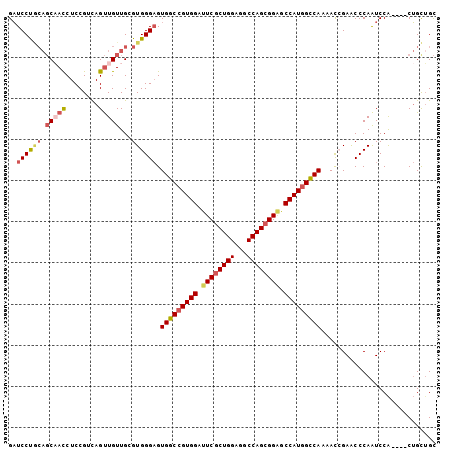
>3L_DroMel_CAF1 16681916 100 - 23771897 GAUCCUGCAGCAACCUCCGUCAGUUGUUGGGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA----CUGCUGC ......((((((((........)))(((((((.((..(((((((((.((((((((...)))))))).)))))))))...))..)))))))...----..))))) ( -49.50) >DroSec_CAF1 3622 100 - 1 GAUCCUGCAGCAAUCUCCGUCAGUUGUUGCGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA----CUGCUGC ......((((((.(((((((........))).)))).(((((((((.((((((((...)))))))).))))))))).................----.)))))) ( -44.40) >DroSim_CAF1 3417 100 - 1 GAUCCUGCAGCAAUCUCCGUCAGUUGUUGCGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA----CUGCUGC ......((((((.(((((((........))).)))).(((((((((.((((((((...)))))))).))))))))).................----.)))))) ( -44.40) >DroEre_CAF1 3893 100 - 1 GAUCCUACAGCAACCUCCGACAGUUGUUGGGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGGGCCAUGGCCAAAACCGAACCCAAUCCA----CUGUUGC .................(((((((.(((((((.((..(((((((((.((((((((...)))))))).)))))))))...))..)))))))..)----)))))). ( -50.10) >DroYak_CAF1 3186 100 - 1 GAUCCUACAGCUACCUCCGUCAGUUGUUGGGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACGGAACCCAAUCCA----CUGUGGC ................(((.((((.(((((((.(...(((((((((.((((((((...)))))))).)))))))))...)...)))))))..)----)))))). ( -46.60) >DroAna_CAF1 1156 101 - 1 GUUCCCC---CCAUCUCCGUUAGCUGUUGCGUGGGCGUGGCCGUGGACUCGCUGGAAGCCAGAGGUGCCAUCGUCAUAAUAGAGCCCAGUCCGGCAGCAGCUGU .......---..........((((((((((.((((((((((.(((((((..((((...)))).))).)))).)))))....(....).))))))))))))))). ( -37.70) >consensus GAUCCUGCAGCAACCUCCGUCAGUUGUUGCGUGGGAGUGGCCGUGGAUUCGCUGGAGGCCAGCGGAGCCAUGGCCAAAACCGAACCCAAUCCA____CUGCUGC ..((((((.(((((........)))))...)))))).(((((((((.((((((((...)))))))).)))))))))............................ (-34.45 = -35.32 + 0.86)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:14:11 2006