
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 16,615,729 – 16,615,846 |
| Length | 117 |
| Max. P | 0.992496 |

| Location | 16,615,729 – 16,615,846 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.95 |
| Mean single sequence MFE | -37.78 |
| Consensus MFE | -30.82 |
| Energy contribution | -30.90 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.647124 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
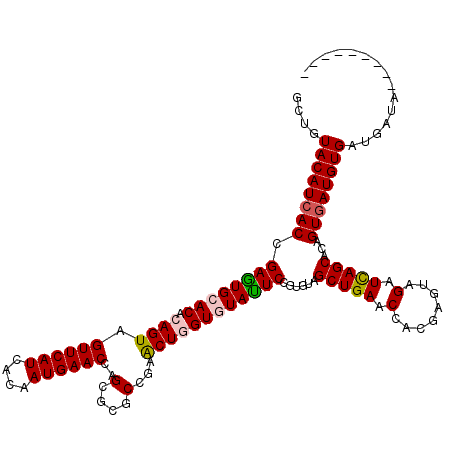
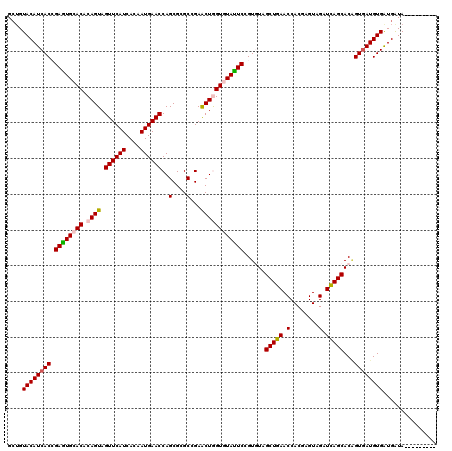
>3L_DroMel_CAF1 16615729 117 + 23771897 GCUGUACAUCACCGAGUGCACGCAGUAGUUCAUCACGAUGAACCAGCGGGCAGAGCUGGUAUAUUCCGUAUAGCUGAACCACGAGUAGAUCAGCACGGUGAUGUGGUGAUACCUGU---G ....((((((((((..(((.(((....((((((....))))))..))).)))..((((((.(((((.((...........))))))).)))))).))))))))))...........---. ( -39.90) >DroVir_CAF1 407 111 + 1 GCUGUACAUCACCGAAUGCACACAGUAGUUCAUCACAAUGAACCAGCGUGCGGAACUAGUGUAUUCCGUAUAGCUGAACCACGAGUAGAUUAGCACCGUAAUGUGAUGAUA--------- (((((.(((......)))...)))))...((((((((......((((((((((((........)))))))).))))....(((.((........)))))..))))))))..--------- ( -30.90) >DroGri_CAF1 391 111 + 1 GCUGUACAUCACCGAGUGCACACAGUAGUUCAUCACAAUGAACCAGCGCGCCGAACUGGUGUAUUCGGUGUAGCUGAACCACGAGUAGAUCAGCACAGUGAUGUGAUGAUA--------- (((((....((((((((((((.((((.((((((....))))))...((...)).))))))))))))))))..(((((.(........).))))))))))............--------- ( -38.20) >DroWil_CAF1 56776 111 + 1 GGAGUACAUCACGGAAUGCACACAGUAGUUCAUCACAAUGAACCAGCGCGCCGAGCUGGUGUAUUCGGUGUAGCUGAACCACGAGUAGAUCAGCACGGUGAUGUGAUGAUA--------- ......(((((((...........((.((((((....))))))..)).(((((.((((((.((((((..((......))..)))))).)))))).))))).)))))))...--------- ( -40.10) >DroMoj_CAF1 404 111 + 1 GCUGUACAUCACAGAGUGCACACAGUAGUUCAUCACAAUGAACCAGCGGGCGGAACUAGUGUAUUCGGUGUAGCUGAACCACGAGUAGAUCAGCACAGUGAUGUGAUGAUA--------- ....((((((((.((((((((...((.((((((....))))))..))((......)).))))))))..(((.(((((.(........).))))))))))))))))......--------- ( -34.10) >DroAna_CAF1 407 120 + 1 GCUGUACAUCACCGAGUGGACGCAGUAGUUCAUCACGAUGAACCAGCGUGCCGAGCUGGUGUACUCCGUGUAGCUGAACCACGAGUAGAUCAGCACAGUGAUGUGAUGGUACCUGUUUCG ((((((((((((.(..(((((((....((((((....))))))..)))).))).((((((.(((((.(((.........)))))))).)))))).).))))))).))))).......... ( -43.50) >consensus GCUGUACAUCACCGAGUGCACACAGUAGUUCAUCACAAUGAACCAGCGCGCCGAACUGGUGUAUUCCGUGUAGCUGAACCACGAGUAGAUCAGCACAGUGAUGUGAUGAUA_________ ....((((((((.((((((((.((((.((((((....))))))..(....)...))))))))))))......(((((.(........).)))))...))))))))............... (-30.82 = -30.90 + 0.08)

| Location | 16,615,729 – 16,615,846 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.95 |
| Mean single sequence MFE | -37.87 |
| Consensus MFE | -34.14 |
| Energy contribution | -34.37 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.33 |
| SVM RNA-class probability | 0.992496 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
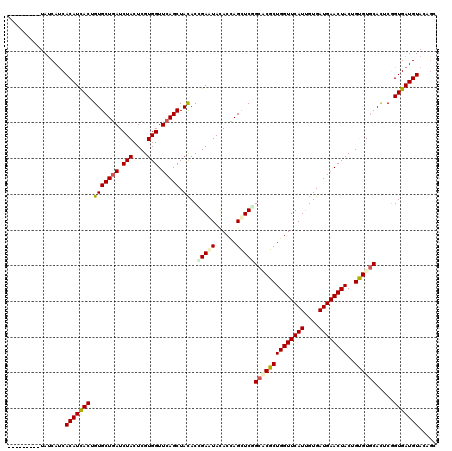
>3L_DroMel_CAF1 16615729 117 - 23771897 C---ACAGGUAUCACCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUAUACGGAAUAUACCAGCUCUGCCCGCUGGUUCAUCGUGAUGAACUACUGCGUGCACUCGGUGAUGUACAGC .---...((.....))(((((((((.(((((.(((....))).))))).....((......)).....(((.(((((((((((....))))))))..))).)))..)))))))))..... ( -40.70) >DroVir_CAF1 407 111 - 1 ---------UAUCAUCACAUUACGGUGCUAAUCUACUCGUGGUUCAGCUAUACGGAAUACACUAGUUCCGCACGCUGGUUCAUUGUGAUGAACUACUGUGUGCAUUCGGUGAUGUACAGC ---------...(((((((((((((((......))).))))))..........(((((......)))))((((((((((((((....))))))))..)))))).....))))))...... ( -34.30) >DroGri_CAF1 391 111 - 1 ---------UAUCAUCACAUCACUGUGCUGAUCUACUCGUGGUUCAGCUACACCGAAUACACCAGUUCGGCGCGCUGGUUCAUUGUGAUGAACUACUGUGUGCACUCGGUGAUGUACAGC ---------.......(((((((((.(((((.(((....))).))))).....(((((......)))))((((((((((((((....))))))))..))))))...)))))))))..... ( -41.10) >DroWil_CAF1 56776 111 - 1 ---------UAUCAUCACAUCACCGUGCUGAUCUACUCGUGGUUCAGCUACACCGAAUACACCAGCUCGGCGCGCUGGUUCAUUGUGAUGAACUACUGUGUGCAUUCCGUGAUGUACUCC ---------.......(((((((((.((((...((.(((..((......))..))).))...)))).))((((((((((((((....))))))))..)))))).....)))))))..... ( -36.40) >DroMoj_CAF1 404 111 - 1 ---------UAUCAUCACAUCACUGUGCUGAUCUACUCGUGGUUCAGCUACACCGAAUACACUAGUUCCGCCCGCUGGUUCAUUGUGAUGAACUACUGUGUGCACUCUGUGAUGUACAGC ---------.......(((((((((((((((.(((....))).))))).)))..((((......)))).((.(((((((((((....))))))))..))).)).....)))))))..... ( -32.60) >DroAna_CAF1 407 120 - 1 CGAAACAGGUACCAUCACAUCACUGUGCUGAUCUACUCGUGGUUCAGCUACACGGAGUACACCAGCUCGGCACGCUGGUUCAUCGUGAUGAACUACUGCGUCCACUCGGUGAUGUACAGC ........((((.(((((.....((((((((.(((....))).))))).)))((((((......))))((.((((((((((((....))))))))..))))))...)))))))))))... ( -42.10) >consensus _________UAUCAUCACAUCACUGUGCUGAUCUACUCGUGGUUCAGCUACACCGAAUACACCAGCUCGGCACGCUGGUUCAUUGUGAUGAACUACUGUGUGCACUCGGUGAUGUACAGC ................(((((((.(((((((.(((....))).))))).))..(((((......)))))((((((((((((((....))))))))..)))))).....)))))))..... (-34.14 = -34.37 + 0.23)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:13:42 2006