
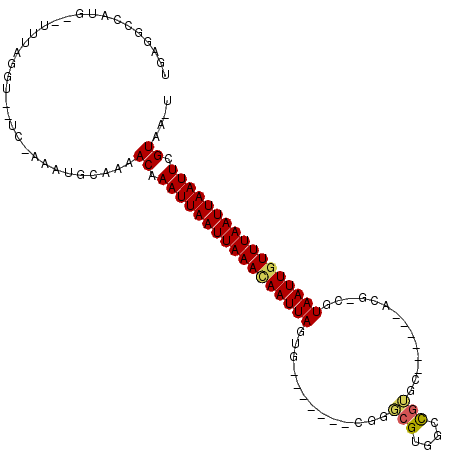
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 16,388,259 – 16,388,354 |
| Length | 95 |
| Max. P | 0.995470 |

| Location | 16,388,259 – 16,388,354 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 76.51 |
| Mean single sequence MFE | -26.33 |
| Consensus MFE | -13.62 |
| Energy contribution | -13.65 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.67 |
| Structure conservation index | 0.52 |
| SVM decision value | 1.98 |
| SVM RNA-class probability | 0.984544 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16388259 95 + 23771897 UGAGGCCUUG--UUUACGC--UU-AAAUGCAAAACAAAUUAAUUAAACAAUUAGUG-------CGAUCGUGCCCGGGC------CCG-UGUAAUUGUUUAAUUAAUUCGUAAAU .........(--(((((((--..-....))......(((((((((((((((((.((-------((.(((....)))..------.))-))))))))))))))))))).)))))) ( -23.60) >DroVir_CAF1 3676 103 + 1 UGAGGCCAUGGGCUUGGGU--UCAAUAUGCAAAACAAAUUAAUUAAACAAUUAGUG-------UGGGCGUGGGCGUGGGCGUUGGCG-CGUAAUUGUUUAAUUAAUUCGUAA-U ((((.(((......))).)--))).........((.((((((((((((((((((((-------(.(((((........))))).)))-).))))))))))))))))).))..-. ( -35.50) >DroPse_CAF1 4690 102 + 1 UGAGGCCUUG--UUUACGC--UU-AAAUGCAAAACAAAUUAAUUAAAAAAUUAGUGGGCGACGCGGGCGGACCCGGAC------ACG-UGUAAUUGUUUAAUUAAUUCGUAAAU .........(--(((((((--..-....))......(((((((((((.(((((((((....).((((....))))..)------)).-..))))).))))))))))).)))))) ( -22.20) >DroGri_CAF1 3444 94 + 1 UGAGGCCAUG--CUAGGGU--UCGACAUGCAAAACAAAUUAAUUAAACAAUUAGUG-------UGAGCG-GGGCGUGU------GCG-CGUAAUUGUUUAAUUAAUUCGUAA-U .(((.((...--...)).)--))..........((.((((((((((((((((((((-------((.(((-...))).)------)))-).))))))))))))))))).))..-. ( -27.30) >DroWil_CAF1 3277 94 + 1 UGAGGCCUUU--AUUAAG----A-AAUUGCAAAACAAAUUAAUUAAAUAAUUAAUG-------UGGGCGUGUCUCUCA------ACGCCUUAAUUGUUUAAUUAAUUCGUAAAU ..........--......----.-.........((.(((((((((((((((((...-------.((((((........------))))))))))))))))))))))).)).... ( -23.00) >DroMoj_CAF1 3476 98 + 1 UGAUGCCAUAU-UUUGGGUUUUCGAUAUGCAAAACAAAUUAAUUAAACAAUUAGUU-------CGGGCGUGGGCGUGG------ACG-CAUAAUUGUUUAAUUAAUUCGUAA-U .((.(((....-....)))..))..........((.((((((((((((((((((((-------((.((....)).)))------)).-..))))))))))))))))).))..-. ( -26.40) >consensus UGAGGCCAUG__UUUAGGU__UC_AAAUGCAAAACAAAUUAAUUAAACAAUUAGUG_______CGGGCGUGGCCGUGC______ACG_CGUAAUUGUUUAAUUAAUUCGUAA_U .................................((.(((((((((((((((((.............(((....)))..............))))))))))))))))).)).... (-13.62 = -13.65 + 0.03)


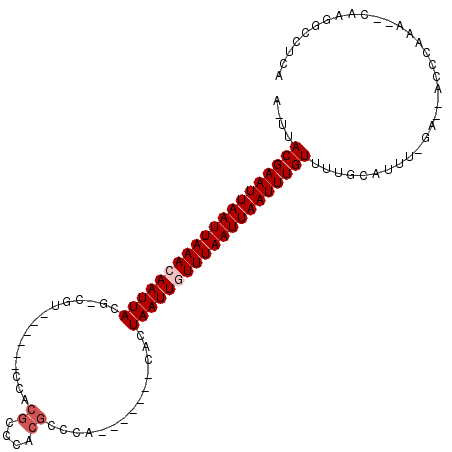
| Location | 16,388,259 – 16,388,354 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 76.51 |
| Mean single sequence MFE | -22.58 |
| Consensus MFE | -14.07 |
| Energy contribution | -14.73 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.35 |
| Structure conservation index | 0.62 |
| SVM decision value | 2.58 |
| SVM RNA-class probability | 0.995470 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16388259 95 - 23771897 AUUUACGAAUUAAUUAAACAAUUACA-CGG------GCCCGGGCACGAUCG-------CACUAAUUGUUUAAUUAAUUUGUUUUGCAUUU-AA--GCGUAAA--CAAGGCCUCA ....((((((((((((((((((((..-((.------...)).((......)-------)..))))))))))))))))))))(((((....-..--..)))))--.......... ( -23.00) >DroVir_CAF1 3676 103 - 1 A-UUACGAAUUAAUUAAACAAUUACG-CGCCAACGCCCACGCCCACGCCCA-------CACUAAUUGUUUAAUUAAUUUGUUUUGCAUAUUGA--ACCCAAGCCCAUGGCCUCA .-..((((((((((((((((((((.(-((....)))....((....))...-------...)))))))))))))))))))).........(((--..(((......)))..))) ( -23.90) >DroPse_CAF1 4690 102 - 1 AUUUACGAAUUAAUUAAACAAUUACA-CGU------GUCCGGGUCCGCCCGCGUCGCCCACUAAUUUUUUAAUUAAUUUGUUUUGCAUUU-AA--GCGUAAA--CAAGGCCUCA ....((((((((((((((.(((((..-.((------(.(((((....)))).).)))....))))).))))))))))))))(((((....-..--..)))))--.......... ( -25.10) >DroGri_CAF1 3444 94 - 1 A-UUACGAAUUAAUUAAACAAUUACG-CGC------ACACGCCC-CGCUCA-------CACUAAUUGUUUAAUUAAUUUGUUUUGCAUGUCGA--ACCCUAG--CAUGGCCUCA .-..((((((((((((((((((((.(-((.------...)))..-.(....-------)..))))))))))))))))))))....(((((...--......)--))))...... ( -21.90) >DroWil_CAF1 3277 94 - 1 AUUUACGAAUUAAUUAAACAAUUAAGGCGU------UGAGAGACACGCCCA-------CAUUAAUUAUUUAAUUAAUUUGUUUUGCAAUU-U----CUUAAU--AAAGGCCUCA ....((((((((((((((.(((((((((((------........)))))..-------..)))))).)))))))))))))).........-.----......--.......... ( -20.00) >DroMoj_CAF1 3476 98 - 1 A-UUACGAAUUAAUUAAACAAUUAUG-CGU------CCACGCCCACGCCCG-------AACUAAUUGUUUAAUUAAUUUGUUUUGCAUAUCGAAAACCCAAA-AUAUGGCAUCA .-..((((((((((((((((((((.(-(((------........))))...-------...))))))))))))))))))))..(((((((............-)))).)))... ( -21.60) >consensus A_UUACGAAUUAAUUAAACAAUUACG_CGU______CCACGCCCACGCCCA_______CACUAAUUGUUUAAUUAAUUUGUUUUGCAUUU_GA__ACCCAAA__CAAGGCCUCA ....((((((((((((((((((((...............((....))..............))))))))))))))))))))................................. (-14.07 = -14.73 + 0.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:12:19 2006