
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 16,211,111 – 16,211,271 |
| Length | 160 |
| Max. P | 0.905140 |

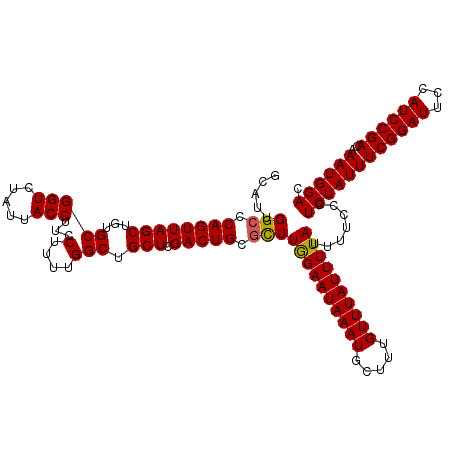
| Location | 16,211,111 – 16,211,231 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.17 |
| Mean single sequence MFE | -29.12 |
| Consensus MFE | -26.32 |
| Energy contribution | -26.12 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.04 |
| SVM RNA-class probability | 0.905140 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16211111 120 + 23771897 GCAUUGGCCCAGUUAGCUGUGCGGUAUAUUACCUUCCUUUUGGCUGCUCGACUGCGCUUAGAAUAAAUGCUUUGUUUAUUUUAUUUUCGCUCUAUUUCGGAUUCCAUCCGACAAAUGGAC ((...(((.((((((((...((((((...))))...(....))).))).))))).)))((((((((((.....)))))))))).....))((((((((((((...))))))..)))))). ( -30.80) >DroSec_CAF1 29881 120 + 1 GCAUUGGCCCAGUUAGCUGUGCGGUCUAUUACCUUCCUUUUGGCUGCUCGACUGCGCUUGGAAUAAAUGCUUUGUUUAUUUUAUUUUCCCUCUAUUUCGGAUUCCAUCCGACAAAUGGAC (((((..((.((((((.((.((((((...............)))))).)).))).))).))....)))))....................((((((((((((...))))))..)))))). ( -29.56) >DroSim_CAF1 31753 120 + 1 GCAUUGGCCCAGUUAGCUGUGCGGUCUAUUACCUUCCUUUUGGCUGCUCGACUGCGCUUGGAAUAAAUGCUUUGUUUAUUUUAUUUUCCCUCUAUUUCGGAUUCCAUCCGACAAAUGGAC (((((..((.((((((.((.((((((...............)))))).)).))).))).))....)))))....................((((((((((((...))))))..)))))). ( -29.56) >DroEre_CAF1 29367 120 + 1 GCAUUGGCCCAGUUAGCAAUGCGGUCUAUUACCUUCCUUUUGGCUGCUCGACUGCGUUUGGAAUAAAUGUUUUGUUUAUUUUAUUUUUCCUCUAUUUCGGAUUCCAUCCGACAAAUGGAC .....(((.(((((((((....(((.....)))..((....)).)))).))))).)))((((((((((.....)))))))))).......((((((((((((...))))))..)))))). ( -26.10) >DroYak_CAF1 66475 119 + 1 GCAUUGGGCCAGUUAGCUGUGCGGUCUAUUACCUUCCU-UUGGCUGCUCGACUGCGUUUGGAAUAAAUGUUUUGUUUAUUUUAUUUUUCCUCUAUUUCGGAUUCCAUCCGACAAAUGGAC (((((((((.((((((..(...(((.....)))..)..-)))))))))))).)))...((((((((((.....)))))))))).......((((((((((((...))))))..)))))). ( -29.60) >consensus GCAUUGGCCCAGUUAGCUGUGCGGUCUAUUACCUUCCUUUUGGCUGCUCGACUGCGCUUGGAAUAAAUGCUUUGUUUAUUUUAUUUUCCCUCUAUUUCGGAUUCCAUCCGACAAAUGGAC .....(((.((((((((...(((((.....)))...(....))).))).))))).)))((((((((((.....)))))))))).......((((((((((((...))))))..)))))). (-26.32 = -26.12 + -0.20)

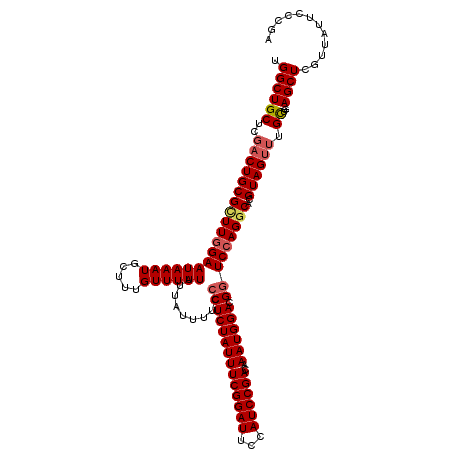
| Location | 16,211,151 – 16,211,271 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.45 |
| Mean single sequence MFE | -32.32 |
| Consensus MFE | -26.04 |
| Energy contribution | -26.72 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.855040 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16211151 120 + 23771897 UGGCUGCUCGACUGCGCUUAGAAUAAAUGCUUUGUUUAUUUUAUUUUCGCUCUAUUUCGGAUUCCAUCCGACAAAUGGACGGUCCAGGCACGUAGUUUGUUCGUAGCUCGUUAUUCCCGA .((((((..(((((((..((((((((((.....))))))))))....)))((((((((((((...))))))..)))))).))))(((((.....)))))...))))))............ ( -32.00) >DroSec_CAF1 29921 118 + 1 UGGCUGCUCGACUGCGCUUGGAAUAAAUGCUUUGUUUAUUUUAUUUUCCCUCUAUUUCGGAUUCCAUCCGACAAAUGGACGGUCCAGUCACGUAGUUUGCUC--AGCUCGUUAUUCCCGA .(((((...(((((((.(((((((((((.....))))))........((.((((((((((((...))))))..)))))).)))))))...)))))))....)--))))............ ( -31.10) >DroSim_CAF1 31793 118 + 1 UGGCUGCUCGACUGCGCUUGGAAUAAAUGCUUUGUUUAUUUUAUUUUCCCUCUAUUUCGGAUUCCAUCCGACAAAUGGACGGUCCAGGCACGUAGUUUGCUC--AGCUCGUUAUUCCCGA .(((((...(((((((((((((((((((.....))))))........((.((((((((((((...))))))..)))))).)))))))))..))))))....)--))))............ ( -34.80) >DroEre_CAF1 29407 117 + 1 UGGCUGCUCGACUGCGUUUGGAAUAAAUGUUUUGUUUAUUUUAUUUUUCCUCUAUUUCGGAUUCCAUCCGACAAAUGGACGGUCCAGGCACGUAGC-UGCUC--AGCUCGUUAUUCCCAA .(((((..((.(((((((((((((((((.....)))))).........((((((((((((((...))))))..)))))).)))))))..)))))).-))..)--))))............ ( -31.40) >DroYak_CAF1 66514 118 + 1 UGGCUGCUCGACUGCGUUUGGAAUAAAUGUUUUGUUUAUUUUAUUUUUCCUCUAUUUCGGAUUCCAUCCGACAAAUGGACGGUCCAGGCACGUAGUUUGCUC--AGCUCGUUAUUCCCGA .(((((...(((((((((((((((((((.....)))))).........((((((((((((((...))))))..)))))).)))))))..))))))))....)--))))............ ( -32.30) >consensus UGGCUGCUCGACUGCGCUUGGAAUAAAUGCUUUGUUUAUUUUAUUUUCCCUCUAUUUCGGAUUCCAUCCGACAAAUGGACGGUCCAGGCACGUAGUUUGCUC__AGCUCGUUAUUCCCGA .((((((..(((((((((((((((((((.....)))))).........((((((((((((((...))))))..)))))).)))))))))..)))))).))....))))............ (-26.04 = -26.72 + 0.68)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:10:23 2006