
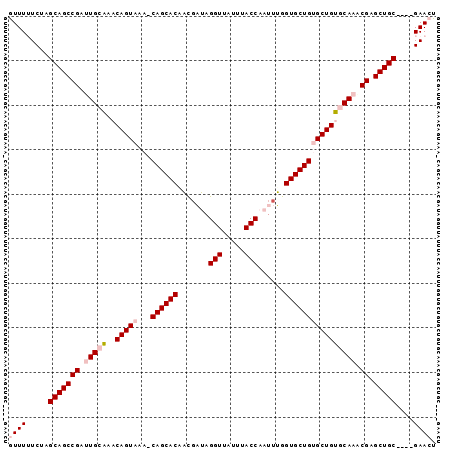
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 245,398 – 245,489 |
| Length | 91 |
| Max. P | 0.978532 |

| Location | 245,398 – 245,489 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | forward |
| Mean pairwise identity | 85.51 |
| Mean single sequence MFE | -36.67 |
| Consensus MFE | -28.86 |
| Energy contribution | -29.75 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.03 |
| Mean z-score | -4.45 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.82 |
| SVM RNA-class probability | 0.978532 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
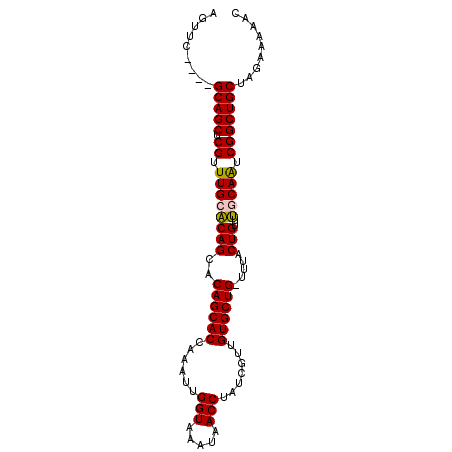
>3L_DroMel_CAF1 245398 91 + 23771897 GUUUUUCUGGCAGCCGAUUGCAAACAGUAAA-CAGCACGACGAUAGGUUAUUUACCAAUUUGGUGCUGUGCUGUGCAAGCGAGCUGC----GAACU .........(((((((.((((..(((((..(-((((((.(.(((.(((.....))).)))).)))))))))))))))).)).)))))----..... ( -35.60) >DroPse_CAF1 13818 94 + 1 AUUUAUCUAGCAGCCGCCUGCU--CAGUAAAGCAGCACAUCGCAAGGUUAUGUACCAAUUGGGUGCUGCGCUGAGCAAACGAGCUGCCUAUGAACU .(((((...(((((((..((((--((((...((((((((((....)))......((....)))))))))))))))))..)).)))))..))))).. ( -41.90) >DroSec_CAF1 13379 91 + 1 GUUUUUCUAGCAGCCGAUUGGAAACAGUAGA-CAGCACAACGAUAGGUUAUUUACCAAUUUGGUGCUGUGCUGUGCAAACGAGCUGC----GAACU .........(((((((.(((...(((((..(-((((((...(((.(((.....))).)))..)))))))))))).))).)).)))))----..... ( -33.70) >DroSim_CAF1 13796 91 + 1 GUUUUUCUAGCAGCCGAUUGGAAACAGUAGA-CAGCACAACGAUAGGUUAUUUACCAAUUUGGUGCUGUGCUGUGCAAACGAGCUGC----GAACU .........(((((((.(((...(((((..(-((((((...(((.(((.....))).)))..)))))))))))).))).)).)))))----..... ( -33.70) >DroEre_CAF1 13256 91 + 1 GUUUUUCUGGCAGCCGAUUGCAAACAGUAAA-CAGCACAAUCAUAGGUUAUUUACCAAUUUAGUGCUGUGCUGUGCAAGCGAGCUGC----GAACU .........(((((((.((((..(((((..(-((((((.......(((.....)))......)))))))))))))))).)).)))))----..... ( -33.22) >DroPer_CAF1 14188 94 + 1 AUUUAUCUAGCAGCCGCCUGCU--CAGUAAAGCAGCACAUCGCAAGGUUAUGUACCAAUUGGGUGCUGCGCUGAGCAAACGAGCUGCCUAUGAACU .(((((...(((((((..((((--((((...((((((((((....)))......((....)))))))))))))))))..)).)))))..))))).. ( -41.90) >consensus GUUUUUCUAGCAGCCGAUUGCAAACAGUAAA_CAGCACAACGAUAGGUUAUUUACCAAUUUGGUGCUGUGCUGUGCAAACGAGCUGC____GAACU ((((.....(((((((.(((((..(((((...((((((.......(((.....)))......)))))))))))))))).)).)))))....)))). (-28.86 = -29.75 + 0.89)


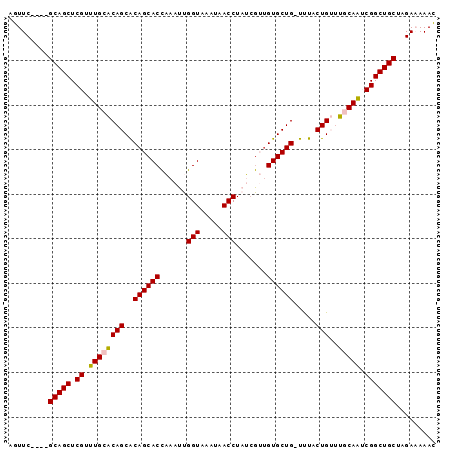
| Location | 245,398 – 245,489 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | reverse |
| Mean pairwise identity | 85.51 |
| Mean single sequence MFE | -34.15 |
| Consensus MFE | -26.83 |
| Energy contribution | -26.50 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.08 |
| Mean z-score | -4.25 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972992 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 245398 91 - 23771897 AGUUC----GCAGCUCGCUUGCACAGCACAGCACCAAAUUGGUAAAUAACCUAUCGUCGUGCUG-UUUACUGUUUGCAAUCGGCUGCCAGAAAAAC .....----(((((.((.((((((((.(((((((......(((.....))).......))))))-)...))))..)))).)))))))......... ( -30.92) >DroPse_CAF1 13818 94 - 1 AGUUCAUAGGCAGCUCGUUUGCUCAGCGCAGCACCCAAUUGGUACAUAACCUUGCGAUGUGCUGCUUUACUG--AGCAGGCGGCUGCUAGAUAAAU ........((((((.(((((((((((.(((((((......(((.....))).......)))))))....)))--))))))))))))))........ ( -43.12) >DroSec_CAF1 13379 91 - 1 AGUUC----GCAGCUCGUUUGCACAGCACAGCACCAAAUUGGUAAAUAACCUAUCGUUGUGCUG-UCUACUGUUUCCAAUCGGCUGCUAGAAAAAC .....----(((((.((.(((.((((.(((((((......(((.....))).......))))))-)...))))...))).)))))))......... ( -27.92) >DroSim_CAF1 13796 91 - 1 AGUUC----GCAGCUCGUUUGCACAGCACAGCACCAAAUUGGUAAAUAACCUAUCGUUGUGCUG-UCUACUGUUUCCAAUCGGCUGCUAGAAAAAC .....----(((((.((.(((.((((.(((((((......(((.....))).......))))))-)...))))...))).)))))))......... ( -27.92) >DroEre_CAF1 13256 91 - 1 AGUUC----GCAGCUCGCUUGCACAGCACAGCACUAAAUUGGUAAAUAACCUAUGAUUGUGCUG-UUUACUGUUUGCAAUCGGCUGCCAGAAAAAC .....----(((((.((.((((((((.(((((((...((.(((.....))).))....))))))-)...))))..)))).)))))))......... ( -31.90) >DroPer_CAF1 14188 94 - 1 AGUUCAUAGGCAGCUCGUUUGCUCAGCGCAGCACCCAAUUGGUACAUAACCUUGCGAUGUGCUGCUUUACUG--AGCAGGCGGCUGCUAGAUAAAU ........((((((.(((((((((((.(((((((......(((.....))).......)))))))....)))--))))))))))))))........ ( -43.12) >consensus AGUUC____GCAGCUCGUUUGCACAGCACAGCACCAAAUUGGUAAAUAACCUAUCGUUGUGCUG_UUUACUGUUUGCAAUCGGCUGCUAGAAAAAC .........(((((.((.((((((((..((((((......(((.....))).......)))))).....)))..))))).)))))))......... (-26.83 = -26.50 + -0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:31:21 2006