
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 16,105,597 – 16,105,832 |
| Length | 235 |
| Max. P | 0.998198 |

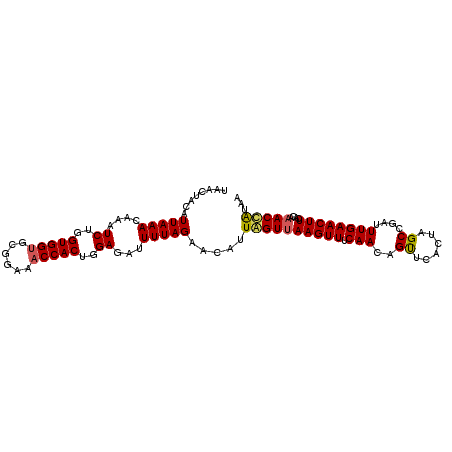
| Location | 16,105,597 – 16,105,701 |
|---|---|
| Length | 104 |
| Sequences | 5 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 94.62 |
| Mean single sequence MFE | -21.04 |
| Consensus MFE | -18.02 |
| Energy contribution | -17.90 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.29 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.603350 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16105597 104 - 23771897 UAACUACAUUAAACAAAUCUGGUGGUGCGGAAACCACUGGAGAUUUUAGAACAUUAGUUAAGUUUCAACAGUUCACUAGCCGAUUUGAACUUCUCAAACCGUAA ((((((..(((((....((..(((((......)))))..))...))))).....)))))).((((....((((((..........))))))....))))..... ( -20.80) >DroSec_CAF1 7809 104 - 1 UAACUACAUUAAACAAAUCUGGUGGUGCGGAAACCACUGGAGAUUUUAGAACAUUGGUUAAGUUUCAACAGUUCACUAGCCGAUUUGAACUUCACAAACCAUAA ((((((..(((((....((..(((((......)))))..))...))))).....)))))).((((....((((((..........))))))....))))..... ( -20.50) >DroSim_CAF1 7815 104 - 1 UAACUACAUUAAACAAAUCUGGUGGUGCGGAAACCACUGGAGAUUUUAGAACAUUGGUUAAGUUUCAACAGUUCACUAGCCGAUUUGAACUUCACAAACCAUAA ((((((..(((((....((..(((((......)))))..))...))))).....)))))).((((....((((((..........))))))....))))..... ( -20.50) >DroEre_CAF1 7867 104 - 1 UAACUACAUUAAACAAAUCUGGUGGUGCGGAAACCACUGGAGAUUUUAGAACAUUAGUUAAGUUACAAGUGCUCACUAGCUGAUUUGAACUUCACACACUAUAA ........(((((....((..(((((......)))))..))...))))).......((.(((((.((((((((....)))..)))))))))).))......... ( -22.40) >DroYak_CAF1 12508 104 - 1 UAACUACAUUAAACAAAUCUGGUGGAGCGGAAACCACUGGAGAUUUUAGAACAUUAGUUAAGUUUCAAGUGCUCACUAGCUGAUUUGAACUUCACAAACUAUAA .............((((((.(((.(((((....).(((.(((((((...(((....)))))))))).)))))))....)))))))))................. ( -21.00) >consensus UAACUACAUUAAACAAAUCUGGUGGUGCGGAAACCACUGGAGAUUUUAGAACAUUAGUUAAGUUUCAACAGUUCACUAGCCGAUUUGAACUUCACAAACCAUAA ........(((((....((..(((((......)))))..))...))))).....((((((((((.(((..((......))....))))))))....)))))... (-18.02 = -17.90 + -0.12)

| Location | 16,105,701 – 16,105,804 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 90.19 |
| Mean single sequence MFE | -38.52 |
| Consensus MFE | -35.51 |
| Energy contribution | -36.12 |
| Covariance contribution | 0.61 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.92 |
| SVM decision value | 3.03 |
| SVM RNA-class probability | 0.998198 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16105701 103 + 23771897 --------------AACUUAACACGAGCUCAGUUAUGAGUUGGCCGCACAGUGGCUGCAAUUUCGGCCAUUGCAGCUGCAGUGGUAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGG --------------.........(.((((((((.((((((((.((((...(..((((((((.......))))))))..).))))))))))))...))).))))).)........... ( -38.40) >DroPse_CAF1 8365 117 + 1 UGUAACUAAGAGUUAACUUAACACGAGCUCAGUUAUGAGUUGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAAUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGG .((((.((((......))))...(.((((((((.((((((((.(((....(..((((((((.......))))))))..)..)))))))))))...))).))))).).....)))).. ( -39.80) >DroEre_CAF1 7971 103 + 1 --------------AACUUAACACGAGCUCAGUUAUGAGUUGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAGUGGUAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGG --------------.........(.((((((((.((((((((.((((...(..((((((((.......))))))))..).))))))))))))...))).))))).)........... ( -39.10) >DroYak_CAF1 12612 103 + 1 --------------AACUUAACACGAGCUCAGUUAUGAGUUGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAGUGGUAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGG --------------.........(.((((((((.((((((((.((((...(..((((((((.......))))))))..).))))))))))))...))).))))).)........... ( -39.10) >DroAna_CAF1 7799 103 + 1 --------------AACUUAACACGAGCUCAGUUAUGAGGCGGCUCCACAGUGACUGCAAUUCCGGCUAUUGCAGCUGCAGUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGG --------------.........((..((((....)))).))...((.((((((.(.((((((..((...((.((((((....))))))))....))..)))))).).)))))).)) ( -34.90) >DroPer_CAF1 8336 117 + 1 UGUAACUAAGAGUUAACUUAACACGAGCUCAGUUAUGAGUUGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAAUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGG .((((.((((......))))...(.((((((((.((((((((.(((....(..((((((((.......))))))))..)..)))))))))))...))).))))).).....)))).. ( -39.80) >consensus ______________AACUUAACACGAGCUCAGUUAUGAGUUGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAGUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGG .......................(.((((((((.((((((((.((((...(((((((((((.......))))))))))).))))))))))))...))).))))).)........... (-35.51 = -36.12 + 0.61)



| Location | 16,105,701 – 16,105,804 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 90.19 |
| Mean single sequence MFE | -31.12 |
| Consensus MFE | -28.10 |
| Energy contribution | -27.47 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948583 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16105701 103 - 23771897 CCGCAAUAAUUCCAACUCUAGCAUGAUGAGCUACCACUGCAGCUGCAAUGGCCGAAAUUGCAGCCACUGUGCGGCCAACUCAUAACUGAGCUCGUGUUAAGUU-------------- .............((((.((((((((((.(((..(((.(..((((((((.......))))))))..).))).))))).((((....)))).))))))))))))-------------- ( -29.80) >DroPse_CAF1 8365 117 - 1 CCGCAAUAAUUCCAACUCUAGCAUGAUGAGCUGCCAUUGCAGCUGCAAUAGCCGGAAUUGCAGCCACUGUGCGGCCAACUCAUAACUGAGCUCGUGUUAAGUUAACUCUUAGUUACA .............((((.((((((((((.(((((((.((..((((((((.......)))))))))).)).))))))).((((....)))).)))))))))))).............. ( -33.70) >DroEre_CAF1 7971 103 - 1 CCGCAAUAAUUCCAACUCUAGCAUGAUGAGCUACCACUGCAGCUGCAAUAGCCGGAAUUGCAGCCACUGUGCGGCCAACUCAUAACUGAGCUCGUGUUAAGUU-------------- .............((((.((((((((((.(((..(((.(..((((((((.......))))))))..).))).))))).((((....)))).))))))))))))-------------- ( -28.90) >DroYak_CAF1 12612 103 - 1 CCGCAAUAAUUCCAACUCUAGCAUGAUGAGCUACCACUGCAGCUGCAAUAGCCGGAAUUGCAGCCACUGUGCGGCCAACUCAUAACUGAGCUCGUGUUAAGUU-------------- .............((((.((((((((((.(((..(((.(..((((((((.......))))))))..).))).))))).((((....)))).))))))))))))-------------- ( -28.90) >DroAna_CAF1 7799 103 - 1 CCGCAAUAAUUCCAACUCUAGCAUGAUGAGCUGCCACUGCAGCUGCAAUAGCCGGAAUUGCAGUCACUGUGGAGCCGCCUCAUAACUGAGCUCGUGUUAAGUU-------------- .............((((.((((((((.(.(((.((((.(..((((((((.......))))))))..).))))))))((.(((....)))))))))))))))))-------------- ( -31.70) >DroPer_CAF1 8336 117 - 1 CCGCAAUAAUUCCAACUCUAGCAUGAUGAGCUGCCAUUGCAGCUGCAAUAGCCGGAAUUGCAGCCACUGUGCGGCCAACUCAUAACUGAGCUCGUGUUAAGUUAACUCUUAGUUACA .............((((.((((((((((.(((((((.((..((((((((.......)))))))))).)).))))))).((((....)))).)))))))))))).............. ( -33.70) >consensus CCGCAAUAAUUCCAACUCUAGCAUGAUGAGCUACCACUGCAGCUGCAAUAGCCGGAAUUGCAGCCACUGUGCGGCCAACUCAUAACUGAGCUCGUGUUAAGUU______________ .............((((.((((((((((.(((..(((.(..((((((((.......))))))))..).))).))))).((((....)))).)))))))))))).............. (-28.10 = -27.47 + -0.64)


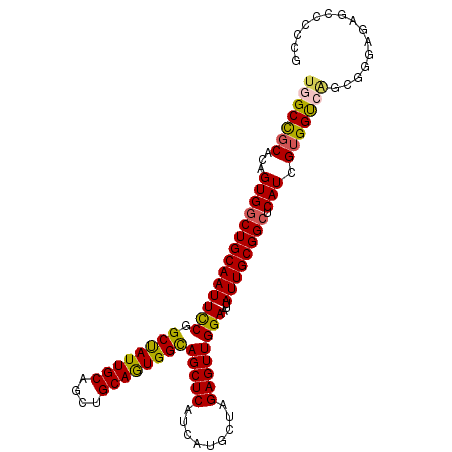
| Location | 16,105,727 – 16,105,832 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 88.38 |
| Mean single sequence MFE | -44.82 |
| Consensus MFE | -37.24 |
| Energy contribution | -36.80 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.89 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948660 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 16105727 105 + 23771897 UGGCCGCACAGUGGCUGCAAUUUCGGCCAUUGCAGCUGCAGUGGUAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGGCUCAUCGUGGUCAGCGGGAGAUCCCCCG (((((((.(((((((((......))))))))).(((((((((((((((((.........)))))...))))))))))))....))))))).((((......)))) ( -46.50) >DroPse_CAF1 8405 105 + 1 UGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAAUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGGCUCAUUGUGGCAGGGAGCAGGGACUCUG ..((((((..((((((((((((((((((...((((((((....)))))......)))...)))))))...)))))))).)))))))))..((((......)))). ( -45.40) >DroEre_CAF1 7997 105 + 1 UGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAGUGGUAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGGCUCAUCGUGGUCAGCGGGAGAUCCCCCG (((((((...((((((((((((((((((...((((((((....)))))......)))...)))))))...)))))))).))).))))))).((((......)))) ( -45.40) >DroYak_CAF1 12638 105 + 1 UGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAGUGGUAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGGCUCAUCGUGGUCAGCGGGAAAUCACCCG (((((((...((((((((((((((((((...((((((((....)))))......)))...)))))))...)))))))).))).))))))).((((......)))) ( -45.00) >DroAna_CAF1 7825 100 + 1 CGGCUCCACAGUGACUGCAAUUCCGGCUAUUGCAGCUGCAGUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGGCUCAUCGUGGUCAGCGGGGCAGC----- (.(((((((.((((((((((((((((((...((((((((....)))))......)))...)))))))...))))))).)))).))))..))).)......----- ( -41.20) >DroPer_CAF1 8376 105 + 1 UGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAAUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGGCUCAUUGUGGCAGGGAGCAGGGACUCUG ..((((((..((((((((((((((((((...((((((((....)))))......)))...)))))))...)))))))).)))))))))..((((......)))). ( -45.40) >consensus UGGCCGCACAGUGGCUGCAAUUCCGGCUAUUGCAGCUGCAGUGGCAGCUCAUCAUGCUAGAGUUGGAAUUAUUGCGGCUCAUCGUGGUCAGCGGGAGAGCCCCCG (((((((...((((((((((((((.((((((((....))))))))(((((.........))))))))...)))))))).))).)))))))............... (-37.24 = -36.80 + -0.44)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:07:39 2006