| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,832,557 – 15,832,677 |
| Length | 120 |
| Max. P | 0.999986 |

| Location | 15,832,557 – 15,832,677 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 99.44 |
| Mean single sequence MFE | -35.37 |
| Consensus MFE | -35.51 |
| Energy contribution | -35.37 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.12 |
| Structure conservation index | 1.00 |
| SVM decision value | 5.40 |
| SVM RNA-class probability | 0.999986 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
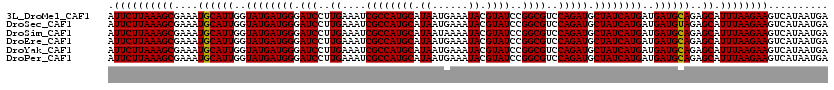
>3L_DroMel_CAF1 15832557 120 + 23771897 AUUCUUAAAGCGAAAUGCAUUGGUAUGAUGGGAUCCUUGAAAUCGCCAUGCAUAAUGAAAUACGUAUCCGGCGUCCAGAUGCUAUCAUGAUGAUGCAGAGCAUUUAAGAAGUCAUAAUGA .((((((((((....((((((..((((((((.(((..((....((((((((.((......)).))))..))))..))))).))))))))..))))))..)).)))))))).......... ( -35.70) >DroSec_CAF1 39620 120 + 1 AUUCUUAAAGCGAAAUGCAUUGGUAUGAUGGGAUCCUUGAAAUCGCCAUGCAUAAUGAAAUACGUAUCCGGCGUCCAGAUGCUAUCAUGAUGAUGUAGAGCAUUUAAGAAGUCAUAAUGA .((((((((((....((((((..((((((((.(((..((....((((((((.((......)).))))..))))..))))).))))))))..))))))..)).)))))))).......... ( -33.70) >DroSim_CAF1 40838 120 + 1 AUUCUUAAAGCGAAAUGCAUUGGUAUGAUGGGAUCCUUGAAAUCGCCAUGCAUAAUAAAAUACGUAUCCGGCGUCCAGAUGCUAUCAUGAUGAUGCAGAGCAUUUAAGAAGUCAUAAUGA .((((((((((....((((((..((((((((.(((..((....((((((((.((......)).))))..))))..))))).))))))))..))))))..)).)))))))).......... ( -35.70) >DroEre_CAF1 38548 120 + 1 AUUCUUAAAGCGAAAUGCAUUGGUAUGAUGGGAUCCUUGAAAUCGCCAUGCAUAAUGAAAUACGUAUCCGGCGUCCAGAUGCUAUCAUGAUGAUGCAGAGCAUUUAAGAAGUCAUAAUGA .((((((((((....((((((..((((((((.(((..((....((((((((.((......)).))))..))))..))))).))))))))..))))))..)).)))))))).......... ( -35.70) >DroYak_CAF1 80832 120 + 1 AUUCUUAAAGCGAAAUGCAUUGGUAUGAUGGGAUCCUUGAAAUCGCCAUGCAUAAUGAAAUACGUAUCCGGCGUCCAGAUGCUAUCAUGAUGAUGCAGAGCAUUUAAGAAGUCAUAAUGA .((((((((((....((((((..((((((((.(((..((....((((((((.((......)).))))..))))..))))).))))))))..))))))..)).)))))))).......... ( -35.70) >DroPer_CAF1 41282 120 + 1 AUUCUUAAAGCGAAAUGCAUUGGUAUGAUGGGAUCCUUGAAAUCGCCAUGCAUAAUGAAAUACGUAUCCGGCGUCCAGAUGCUAUCAUGAUGAUGCAGAGCAUUUAAGAAGUCAUAAUGA .((((((((((....((((((..((((((((.(((..((....((((((((.((......)).))))..))))..))))).))))))))..))))))..)).)))))))).......... ( -35.70) >consensus AUUCUUAAAGCGAAAUGCAUUGGUAUGAUGGGAUCCUUGAAAUCGCCAUGCAUAAUGAAAUACGUAUCCGGCGUCCAGAUGCUAUCAUGAUGAUGCAGAGCAUUUAAGAAGUCAUAAUGA .((((((((((....((((((..((((((((.(((..((....((((((((.((......)).))))..))))..))))).))))))))..))))))..)).)))))))).......... (-35.51 = -35.37 + -0.14)
| Location | 15,832,557 – 15,832,677 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 99.44 |
| Mean single sequence MFE | -27.65 |
| Consensus MFE | -26.90 |
| Energy contribution | -27.07 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.97 |
| SVM decision value | 3.38 |
| SVM RNA-class probability | 0.999121 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
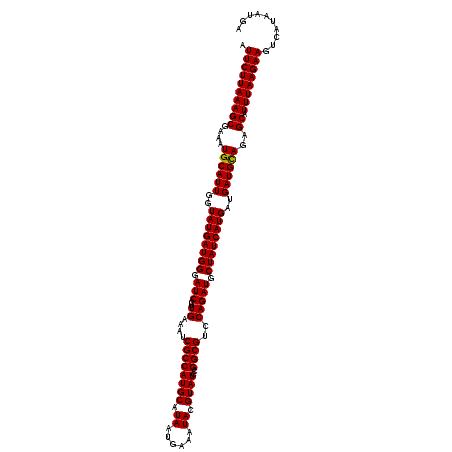
>3L_DroMel_CAF1 15832557 120 - 23771897 UCAUUAUGACUUCUUAAAUGCUCUGCAUCAUCAUGAUAGCAUCUGGACGCCGGAUACGUAUUUCAUUAUGCAUGGCGAUUUCAAGGAUCCCAUCAUACCAAUGCAUUUCGCUUUAAGAAU ..........((((((((.((...(((((.....))).))((((((...))))))..(((((....((((.((((.((((.....))))))))))))..))))).....)))))))))). ( -28.00) >DroSec_CAF1 39620 120 - 1 UCAUUAUGACUUCUUAAAUGCUCUACAUCAUCAUGAUAGCAUCUGGACGCCGGAUACGUAUUUCAUUAUGCAUGGCGAUUUCAAGGAUCCCAUCAUACCAAUGCAUUUCGCUUUAAGAAU ..........((((((((.((.....(((.....))).((((.((((((.......))).......((((.((((.((((.....))))))))))))))))))).....)))))))))). ( -25.90) >DroSim_CAF1 40838 120 - 1 UCAUUAUGACUUCUUAAAUGCUCUGCAUCAUCAUGAUAGCAUCUGGACGCCGGAUACGUAUUUUAUUAUGCAUGGCGAUUUCAAGGAUCCCAUCAUACCAAUGCAUUUCGCUUUAAGAAU ..........((((((((.((...(((((.....))).))((((((...))))))..(((((....((((.((((.((((.....))))))))))))..))))).....)))))))))). ( -28.00) >DroEre_CAF1 38548 120 - 1 UCAUUAUGACUUCUUAAAUGCUCUGCAUCAUCAUGAUAGCAUCUGGACGCCGGAUACGUAUUUCAUUAUGCAUGGCGAUUUCAAGGAUCCCAUCAUACCAAUGCAUUUCGCUUUAAGAAU ..........((((((((.((...(((((.....))).))((((((...))))))..(((((....((((.((((.((((.....))))))))))))..))))).....)))))))))). ( -28.00) >DroYak_CAF1 80832 120 - 1 UCAUUAUGACUUCUUAAAUGCUCUGCAUCAUCAUGAUAGCAUCUGGACGCCGGAUACGUAUUUCAUUAUGCAUGGCGAUUUCAAGGAUCCCAUCAUACCAAUGCAUUUCGCUUUAAGAAU ..........((((((((.((...(((((.....))).))((((((...))))))..(((((....((((.((((.((((.....))))))))))))..))))).....)))))))))). ( -28.00) >DroPer_CAF1 41282 120 - 1 UCAUUAUGACUUCUUAAAUGCUCUGCAUCAUCAUGAUAGCAUCUGGACGCCGGAUACGUAUUUCAUUAUGCAUGGCGAUUUCAAGGAUCCCAUCAUACCAAUGCAUUUCGCUUUAAGAAU ..........((((((((.((...(((((.....))).))((((((...))))))..(((((....((((.((((.((((.....))))))))))))..))))).....)))))))))). ( -28.00) >consensus UCAUUAUGACUUCUUAAAUGCUCUGCAUCAUCAUGAUAGCAUCUGGACGCCGGAUACGUAUUUCAUUAUGCAUGGCGAUUUCAAGGAUCCCAUCAUACCAAUGCAUUUCGCUUUAAGAAU ..........((((((((.((...(((((.....))).))((((((...))))))..(((((....((((.((((.((((.....))))))))))))..))))).....)))))))))). (-26.90 = -27.07 + 0.17)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:02:58 2006