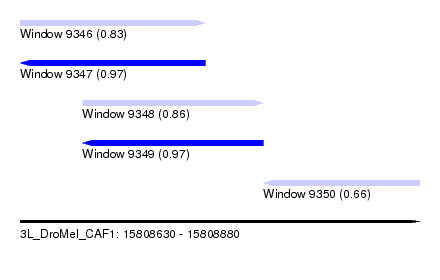
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,808,630 – 15,808,880 |
| Length | 250 |
| Max. P | 0.973592 |

| Location | 15,808,630 – 15,808,746 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 80.18 |
| Mean single sequence MFE | -26.83 |
| Consensus MFE | -18.94 |
| Energy contribution | -18.81 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.825587 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
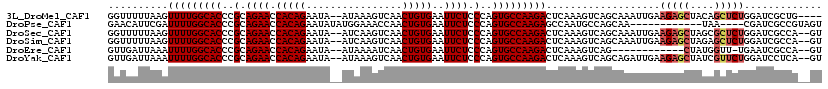
>3L_DroMel_CAF1 15808630 116 + 23771897 GUUCAAAUUGUAAUGGCUUAGAAAUCGUUGUACAAAGCCGGUUUUUAAGUUUUGGCACCCGCAGAACCACAGAAUA--AUAAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAA .................((((((((((((......)).))))))))))((((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))))..... ( -28.94) >DroPse_CAF1 13902 110 + 1 -----GGCUGAAACUUUUC---AGCUGCUGUUUCGAGCCGAACAUUCGAUUUUGGCACCCGCAGAACCACAGAAUAUAUGGAAACCAACUGUGAAUUCUCCCAGUGCCAAGAGCCAAU -----(((((((....)))---))))((....(((((.......)))))(((((((((..(.((((.(((((.......(....)...)))))..)))).)..))))))))))).... ( -31.30) >DroSec_CAF1 16221 116 + 1 GUUUAAAUUGUAAUUGUUUAGAAAUCGUUGUAUAAAGCCGGUUUUUAAGUUUUGGCACCCGCAGAACCACAGAAUA--AUCAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAA .................((((((((((((......)).))))))))))((((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))))..... ( -29.04) >DroSim_CAF1 16547 116 + 1 GCUUAAAUUGUAAUUGUUAAGAAUUUGUUGUAUAAAGCCGGUUUUUAAGUUUUGGCACCCGCAGAACCACAGAAUA--AUCAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAA ................((((((((..(((......)))..))))))))((((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))))..... ( -28.64) >DroEre_CAF1 15421 105 + 1 GUUUAAUUUUU-----------UAUCGUUGUAUGGAGCUGUUGAUUAAAUUUUGGCACCCGCAGAACCACAGAAUA--AUAAAAUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAA .(((((((..(-----------..((........))...)..)))))))(((((((((..(.((((.(((((....--..........)))))..)))).)..)))))))))...... ( -21.14) >DroYak_CAF1 54403 115 + 1 GCCUAAAUUUUAGUUC-UUAGAAAUCGUUGUAUAGAGCCGUUGAUUAAAUUUUGGCACCCGCAGAACCACAGAAUA--AUAAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAA ............((((-(((.((....)).)).)))))..((((.....(((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))).)))). ( -21.94) >consensus GUUUAAAUUGUAAUUGUUUAGAAAUCGUUGUAUAAAGCCGGUUAUUAAAUUUUGGCACCCGCAGAACCACAGAAUA__AUAAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAA ..........................(((......)))...........(((((((((..(.((((.(((((................)))))..)))).)..)))))))))...... (-18.94 = -18.81 + -0.14)


| Location | 15,808,630 – 15,808,746 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 80.18 |
| Mean single sequence MFE | -34.51 |
| Consensus MFE | -30.12 |
| Energy contribution | -30.17 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.24 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.71 |
| SVM RNA-class probability | 0.973592 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15808630 116 - 23771897 UUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUUAU--UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACCGGCUUUGUACAACGAUUUCUAAGCCAUUACAAUUUGAAC (((((((.((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).)))))))).)))))))....(((((...............))))).............. ( -38.46) >DroPse_CAF1 13902 110 - 1 AUUGGCUCUUGGCACUGGGAGAAUUCACAGUUGGUUUCCAUAUAUUCUGUGGUUCUGCGGGUGCCAAAAUCGAAUGUUCGGCUCGAAACAGCAGCU---GAAAAGUUUCAGCC----- ...((((.((((((((.(.(((((.(((((...((......))...)))))))))).).))))))))....(((..((((((((......).))))---)))....)))))))----- ( -41.90) >DroSec_CAF1 16221 116 - 1 UUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUGAU--UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACCGGCUUUAUACAACGAUUUCUAAACAAUUACAAUUUAAAC (((((((.((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).)))))))).)))))))........................................... ( -32.90) >DroSim_CAF1 16547 116 - 1 UUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUGAU--UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACCGGCUUUAUACAACAAAUUCUUAACAAUUACAAUUUAAGC (((((((.((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).)))))))).)))))))........................................... ( -32.90) >DroEre_CAF1 15421 105 - 1 UUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGAUUUUAU--UAUUCUGUGGUUCUGCGGGUGCCAAAAUUUAAUCAACAGCUCCAUACAACGAUA-----------AAAAAUUAAAC .((((((.((((((((.(.(((((.(((((.((((...))--))..)))))))))).).)))))))).))))))......................-----------........... ( -30.10) >DroYak_CAF1 54403 115 - 1 UUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUUAU--UAUUCUGUGGUUCUGCGGGUGCCAAAAUUUAAUCAACGGCUCUAUACAACGAUUUCUAA-GAACUAAAAUUUAGGC ...(((((((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).))))))))............)))))............(((((-(........)))))). ( -30.80) >consensus UUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUUAU__UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACCGGCUCUAUACAACGAUUUCUAAACAAUUACAAUUUAAAC (((((((.((((((((.(.(((((.(((((................)))))))))).).)))))))).)))))))........................................... (-30.12 = -30.17 + 0.06)

| Location | 15,808,669 – 15,808,782 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 80.75 |
| Mean single sequence MFE | -30.50 |
| Consensus MFE | -19.97 |
| Energy contribution | -20.76 |
| Covariance contribution | 0.79 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.864976 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15808669 113 + 23771897 GGUUUUUAAGUUUUGGCACCCGCAGAACCACAGAAUA--AUAAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAAGUCAGCAAAUUGAAGAGCUACAGCUCUGGAUCGCUG---- ...((((.(((((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))))).)))).((((..(((..(((((....))))).))).))))---- ( -34.54) >DroPse_CAF1 13933 103 + 1 GAACAUUCGAUUUUGGCACCCGCAGAACCACAGAAUAUAUGGAAACCAACUGUGAAUUCUCCCAGUGCCAAGAGCCAAUGCCAGCAA------------UAA----CGAUCGCCGUAGU ...((((.(.(((((((((..(.((((.(((((.......(....)...)))))..)))).)..))))))))).).)))).......------------..(----((.....)))... ( -23.30) >DroSec_CAF1 16260 115 + 1 GGUUUUUAAGUUUUGGCACCCGCAGAACCACAGAAUA--AUCAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAAGUCAGCAAAUUGAAGAGCUAGCGCUCUGGAUCGCCA--GU (((((((.(((((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))))).))))............(((((....))))).....))).--.. ( -32.84) >DroSim_CAF1 16586 115 + 1 GGUUUUUAAGUUUUGGCACCCGCAGAACCACAGAAUA--AUCAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAAGUCAGCAAAUUGAAGAGCUAGAGCUCUGGAUCGCCA--GU (((((((.(((((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))))).))))............(((((....))))).....))).--.. ( -33.14) >DroEre_CAF1 15449 102 + 1 GUUGAUUAAAUUUUGGCACCCGCAGAACCACAGAAUA--AUAAAAUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAAGUCAG------------CUAUGGUU-UGAAUCGCCA--GU (((((((...(((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))).....))))))------------)..((((.-......))))--.. ( -27.24) >DroYak_CAF1 54441 115 + 1 GUUGAUUAAAUUUUGGCACCCGCAGAACCACAGAAUA--AUAAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAAGUCAGCAGAUUGAAGAGCUAUCGUUCUGGAUCCUCA--GU (((((((...(((((((((..(.((((.(((((....--..........)))))..)))).)..))))))))).....))))))).((((..(((((....))))).))))....--.. ( -31.94) >consensus GGUUAUUAAAUUUUGGCACCCGCAGAACCACAGAAUA__AUAAAGUCAACUGUGAAUUCUCCCAGUGCCAAGACUCAAAGUCAGCAAAUUGAAGAGCUAUAGCUCUGGAUCGCCA__GU ..........(((((((((..(.((((.(((((................)))))..)))).)..)))))))))...................(((((....)))))............. (-19.97 = -20.76 + 0.79)

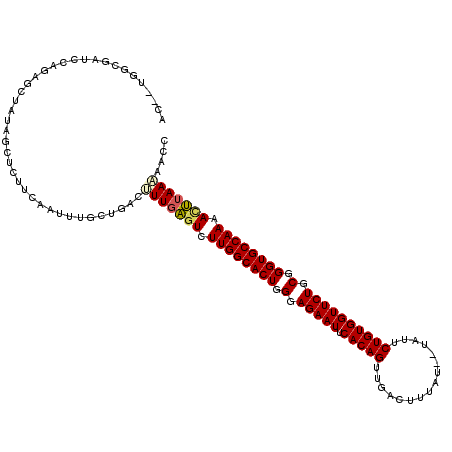
| Location | 15,808,669 – 15,808,782 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 80.75 |
| Mean single sequence MFE | -40.05 |
| Consensus MFE | -30.22 |
| Energy contribution | -30.27 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.82 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966627 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15808669 113 - 23771897 ----CAGCGAUCCAGAGCUGUAGCUCUUCAAUUUGCUGACUUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUUAU--UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACC ----((((((...(((((....))))).....))))))..(((((((.((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).)))))))).))))))).... ( -46.50) >DroPse_CAF1 13933 103 - 1 ACUACGGCGAUCG----UUA------------UUGCUGGCAUUGGCUCUUGGCACUGGGAGAAUUCACAGUUGGUUUCCAUAUAUUCUGUGGUUCUGCGGGUGCCAAAAUCGAAUGUUC ....(((((((..----..)------------))))))(((((.(...((((((((.(.(((((.(((((...((......))...)))))))))).).))))))))...).))))).. ( -38.80) >DroSec_CAF1 16260 115 - 1 AC--UGGCGAUCCAGAGCGCUAGCUCUUCAAUUUGCUGACUUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUGAU--UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACC ..--.(((((...(((((....))))).....)))))...(((((((.((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).)))))))).))))))).... ( -44.20) >DroSim_CAF1 16586 115 - 1 AC--UGGCGAUCCAGAGCUCUAGCUCUUCAAUUUGCUGACUUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUGAU--UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACC ..--.(((((...(((((....))))).....)))))...(((((((.((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).)))))))).))))))).... ( -43.70) >DroEre_CAF1 15449 102 - 1 AC--UGGCGAUUCA-AACCAUAG------------CUGACUUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGAUUUUAU--UAUUCUGUGGUUCUGCGGGUGCCAAAAUUUAAUCAAC ..--....((((((-((.((...------------.))..))))))))((((((((.(.(((((.(((((.((((...))--))..)))))))))).).))))))))............ ( -32.70) >DroYak_CAF1 54441 115 - 1 AC--UGAGGAUCCAGAACGAUAGCUCUUCAAUCUGCUGACUUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUUAU--UAUUCUGUGGUUCUGCGGGUGCCAAAAUUUAAUCAAC .(--((......)))...((((((..........))))...((((((.((((((((.(.(((((.(((((.(((.....)--))..)))))))))).).)))))))).))))))))... ( -34.40) >consensus AC__UGGCGAUCCAGAGCUAUAGCUCUUCAAUUUGCUGACUUUGAGUCUUGGCACUGGGAGAAUUCACAGUUGACUUUAU__UAUUCUGUGGUUCUGCGGGUGCCAAAACUUAAAAACC ........................................(((((((.((((((((.(.(((((.(((((................)))))))))).).)))))))).))))))).... (-30.22 = -30.27 + 0.06)

| Location | 15,808,782 – 15,808,880 |
|---|---|
| Length | 98 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 85.38 |
| Mean single sequence MFE | -25.56 |
| Consensus MFE | -18.37 |
| Energy contribution | -19.53 |
| Covariance contribution | 1.16 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.659825 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15808782 98 - 23771897 AAG-AAACACGGCCAAAAGCCGAAAAGGAUCAACCUGAAAAGUGCU-GACAACUGCCAGAUUUGGAGAAACUAUCUAAAGGUAAGCGAUCUCAGGUGAUC----------- ...-.....((((.....)))).....(((((.(((((....((((-......((((...((((((.......))))))))))))))...))))))))))----------- ( -26.50) >DroSec_CAF1 16375 109 - 1 AAA-AAACACGGUCAGAAACCGAAAAGGAGCAACCUGAAAAGUGCU-GACAACUGCCAGAUUUGGAGAAACUACCUAAAGGUAAGCGAUCCUAGGUGAUCUUCGCUUUUAA ...-.....((((.....))))...((((((...(((...(((...-....)))..)))....(((((...(((((..(((........)))))))).))))))))))).. ( -24.40) >DroSim_CAF1 16701 110 - 1 AAAAAAACACGGUCAGAAACCGAAAAGGAUCAACCUGAAAAGUGCU-GACAACUGCCAGAUUUGGAGAAACUAUCUAAAGGUAAGCGAUCCUAGGUGAUCUUCGCUUUUAA .........((((.....))))...((((((...((....)).(((-......((((...((((((.......)))))))))))))))))))(((((.....))))).... ( -23.90) >DroEre_CAF1 15551 110 - 1 AUG-AAACGCGACCAGAAACCGGAAAGGAUCAACCUGAAAAGUGCUUUACAACUGUCAGAUUUGGAGAAACUAUCCAAAGGUAAGCGAUCUCAGGUGAUCUCCGCUUAGCC ...-....(((.((.......))...((((((.(((((....(((((.((..........((((((.......)))))).)))))))...))))))))))).)))...... ( -28.10) >DroYak_CAF1 54556 110 - 1 AAG-CAACACGGCCAGAAACCGAAAAGGAUCAACCUUAAAAGUGCUCAACAACUCUCAGAUUUGGAGAAACUAUCUAAAGGUAAGCGAUCUCAGGUGAUCCCCGCUUAGAA (((-(....(((.......)))....((((((.(((......((((.......((((.......))))...((((....)))))))).....)))))))))..)))).... ( -24.90) >consensus AAG_AAACACGGCCAGAAACCGAAAAGGAUCAACCUGAAAAGUGCU_GACAACUGCCAGAUUUGGAGAAACUAUCUAAAGGUAAGCGAUCUCAGGUGAUCUCCGCUUAGAA .........((((.....))))....((((((.(((((....((((.......((((...((((((.......))))))))))))))...))))))))))).......... (-18.37 = -19.53 + 1.16)

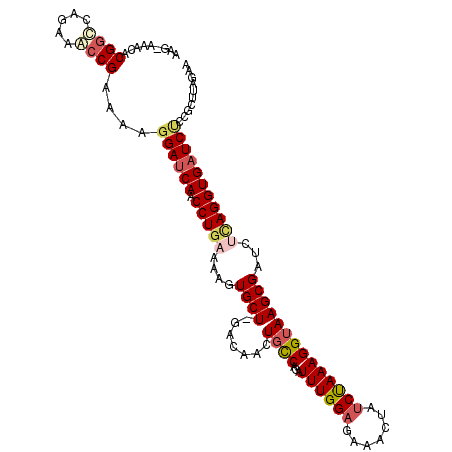
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:02:24 2006