| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,745,887 – 15,745,981 |
| Length | 94 |
| Max. P | 0.783895 |

| Location | 15,745,887 – 15,745,981 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 79.70 |
| Mean single sequence MFE | -31.83 |
| Consensus MFE | -20.53 |
| Energy contribution | -21.03 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.783895 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
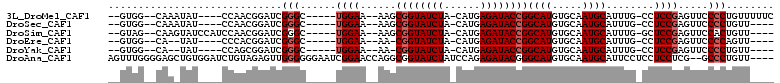
>3L_DroMel_CAF1 15745887 94 + 23771897 --GUGG--CAAAUAU----CCAACGGAUCGGGC-----UGGAA--AAGCGGUAUCUA-CAUGAGAUACCGGCAUGUGCAAUGCAUUUG-CCUCCGAGUUCCCCUGUUUUUC --.(((--.......----)))..(((((((((-----(....--.)))(((((((.-....)))))))((((.((((...)))).))-)).))))..))).......... ( -30.20) >DroSec_CAF1 61110 90 + 1 --GUGG--CAAAUAU----CCAACGGAUCGGGC-----UGGAA--AAGCGGUAUCUA-CAUGAGAUACCGGCAUGUGCAAUGCAUUUG-CCUCCGAGUUCCCCUGUU---- --.(((--.......----)))..(((((((((-----(....--.)))(((((((.-....)))))))((((.((((...)))).))-)).))))..)))......---- ( -30.20) >DroSim_CAF1 63292 94 + 1 --GUAG--CAAGUAUCCAUCCAACGGAUCGGGC-----UGGAA--AAGCGGUAUCUA-CAUGAGAUACCGGCAUGUGCAAUGCAUUUG-GCUCCGAGUUCCACUGUU---- --....--................(((((((((-----..((.--...((((((((.-....))))))))((((.....)))).))..-)).))))..)))......---- ( -31.10) >DroEre_CAF1 52360 87 + 1 --GUGG--CA--UAU----CCCACGGAUCGGGC-----UGGAA--AA-CGGUAUCUA-CAUGAGAUACCGGCAUGUGCAAUGCAUUUG-CCUCCGAGUUCCCCAGUU---- --((((--..--...----.))))......(((-----(((..--((-((((((((.-....)))))))((((.((((...)))).))-)).....)))..))))))---- ( -33.20) >DroYak_CAF1 161483 87 + 1 --GUGG--CA--UAU----CCAGCGGAUCGGGC-----UGGAA--AA-CGGUAUCUA-CAUGAGAUACCGGCAUGUGCAAUGCAUUUG-CCUCCGAGUUCCCCUGUU---- --....--..--...----.(((.((((((((.-----.....--..-.(((((((.-....)))))))((((.((((...)))).))-)))))))..))).)))..---- ( -30.80) >DroAna_CAF1 63463 105 + 1 AGUUUGGGGAGCUGUGGAUCUGUAGAGUUGGGGGGAAUCGGAACCAGGCGGUAUCUAUCCAGAGAUACGGGCAUGUGCAAUGCAUUCCUCCUCCUCG--GCCCUGUU---- ......(((.((((.(((((....))....((((((((..........(.((((((......)))))).)((((.....))))))))))))))).))--)))))...---- ( -35.50) >consensus __GUGG__CAA_UAU____CCAACGGAUCGGGC_____UGGAA__AAGCGGUAUCUA_CAUGAGAUACCGGCAUGUGCAAUGCAUUUG_CCUCCGAGUUCCCCUGUU____ .............................(((......((((......((((((((......))))))))((((.....))))........)))).....)))........ (-20.53 = -21.03 + 0.50)
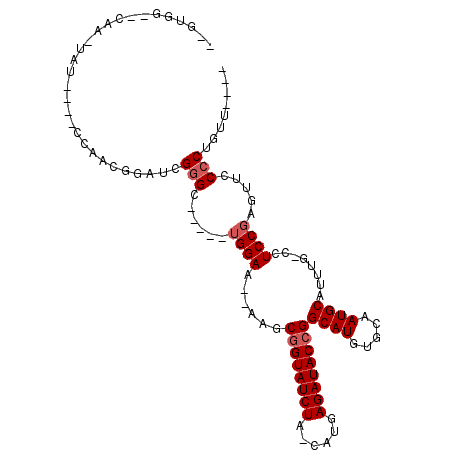
| Location | 15,745,887 – 15,745,981 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 79.70 |
| Mean single sequence MFE | -28.80 |
| Consensus MFE | -19.15 |
| Energy contribution | -20.15 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.756490 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15745887 94 - 23771897 GAAAAACAGGGGAACUCGGAGG-CAAAUGCAUUGCACAUGCCGGUAUCUCAUG-UAGAUACCGCUU--UUCCA-----GCCCGAUCCGUUGG----AUAUUUG--CCAC-- ......(((.(((..((((.((-((..(((...)))..))))(((((((....-.)))))))(((.--....)-----))))))))).))).----.......--....-- ( -29.80) >DroSec_CAF1 61110 90 - 1 ----AACAGGGGAACUCGGAGG-CAAAUGCAUUGCACAUGCCGGUAUCUCAUG-UAGAUACCGCUU--UUCCA-----GCCCGAUCCGUUGG----AUAUUUG--CCAC-- ----..(((.(((..((((.((-((..(((...)))..))))(((((((....-.)))))))(((.--....)-----))))))))).))).----.......--....-- ( -29.80) >DroSim_CAF1 63292 94 - 1 ----AACAGUGGAACUCGGAGC-CAAAUGCAUUGCACAUGCCGGUAUCUCAUG-UAGAUACCGCUU--UUCCA-----GCCCGAUCCGUUGGAUGGAUACUUG--CUAC-- ----..(((((((..((((.((-.....((((.....))))((((((((....-.))))))))...--.....-----)))))))))))))............--....-- ( -25.70) >DroEre_CAF1 52360 87 - 1 ----AACUGGGGAACUCGGAGG-CAAAUGCAUUGCACAUGCCGGUAUCUCAUG-UAGAUACCG-UU--UUCCA-----GCCCGAUCCGUGGG----AUA--UG--CCAC-- ----..((((..(((.....((-((..(((...)))..))))(((((((....-.))))))))-))--..)))-----)((((.....))))----...--..--....-- ( -32.70) >DroYak_CAF1 161483 87 - 1 ----AACAGGGGAACUCGGAGG-CAAAUGCAUUGCACAUGCCGGUAUCUCAUG-UAGAUACCG-UU--UUCCA-----GCCCGAUCCGCUGG----AUA--UG--CCAC-- ----..(((.(((..((((.((-((..(((...)))..))))(((((((....-.))))))).-..--.....-----..))))))).))).----...--..--....-- ( -31.30) >DroAna_CAF1 63463 105 - 1 ----AACAGGGC--CGAGGAGGAGGAAUGCAUUGCACAUGCCCGUAUCUCUGGAUAGAUACCGCCUGGUUCCGAUUCCCCCCAACUCUACAGAUCCACAGCUCCCCAAACU ----....((((--.(.(((((.(((((.(...((.((.((..((((((......)))))).)).))))...)))))).))....((....))))).).))))........ ( -23.50) >consensus ____AACAGGGGAACUCGGAGG_CAAAUGCAUUGCACAUGCCGGUAUCUCAUG_UAGAUACCGCUU__UUCCA_____GCCCGAUCCGUUGG____AUA_UUG__CCAC__ ......(((.(((..((((.........((((.....))))((((((((......)))))))).................))))))).))).................... (-19.15 = -20.15 + 1.00)

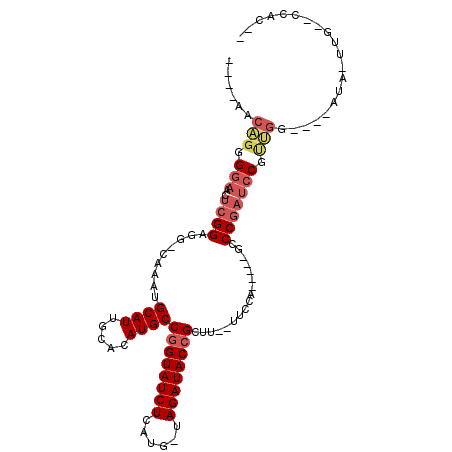
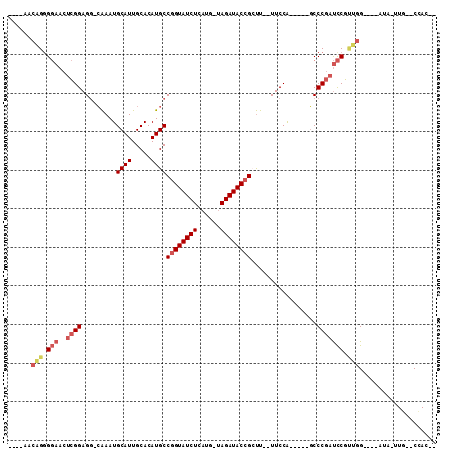
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:00:39 2006