
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,598,698 – 15,598,898 |
| Length | 200 |
| Max. P | 0.998060 |

| Location | 15,598,698 – 15,598,818 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.10 |
| Mean single sequence MFE | -31.68 |
| Consensus MFE | -23.74 |
| Energy contribution | -24.42 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.735320 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15598698 120 - 23771897 AAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGGUUAGGUUACGCCUAUCUAGUUGUCAGUUGAUCGAAUUUAAAUAAAUCAAAUAGGCAUUAUUAUUGAUUG ......(((((((((((..((((((...(((((......))))))))))).......)))))))))))...((((..(((((.............)))))...))))............. ( -26.52) >DroSec_CAF1 21928 116 - 1 AAAAUGAGAUGGGUGUGUGCUU-AAGACUCGGAAAAGCCUCUGAUUGUGGGUUACGUUACGCCCAUCUAGUUGUCAGUUGAUCGAAUUUGAAUAAAUCAAAUAGGCAUU---AUUGAUUG ..(((.(((((((((((.((((-(....(((((......)))))...))))).....))))))))))).)))((((((((.((..((((((.....)))))).))))..---)))))).. ( -31.50) >DroSim_CAF1 21663 117 - 1 AAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGCUUAAGUUACGCCUAUCUAGUUGUCAGUUGAUCGAAUUUGAAUAAAUCAAAUAGGCAUU---AUUGAUUG ..(((.(((((((((((.((((............(((((((.....)))))))))))))))))))))).)))((((((((.((..((((((.....)))))).))))..---)))))).. ( -33.40) >DroEre_CAF1 21882 115 - 1 AAAGUGAGAUGGGU--GUGCUUUAAGACUCGGAAAACCCUCUGAUUGAGGGUUAAGUUACGCCUAUCUAGUUGUCAGUGGAUCGAAUAUGAUUAAAUCGAAUGGGCAUU---ACUGACUG ......((((((((--((((((............(((((((.....)))))))))).)))))))))))....((((((((.....((.((((...)))).)).....))---)))))).. ( -34.90) >DroYak_CAF1 23316 117 - 1 AAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGGUUGAGUUACGCCUAUCUAGUUGUCAGUGGGUCGAAUAUGAUUAUGGCGAAUAGGCAUU---AAUGAUUG ..(((((((((((((((.(((.(((..(((((...((...))..)))))..)))))))))))))))))..((((((...((((......)))).)))))).....))))---........ ( -32.10) >consensus AAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGGUUAAGUUACGCCUAUCUAGUUGUCAGUUGAUCGAAUUUGAAUAAAUCAAAUAGGCAUU___AUUGAUUG ..(((.(((((((((((..((((((...(((((......))))))))))).......))))))))))).)))((((((.((((..((((((.....))))))..).)))...)))))).. (-23.74 = -24.42 + 0.68)

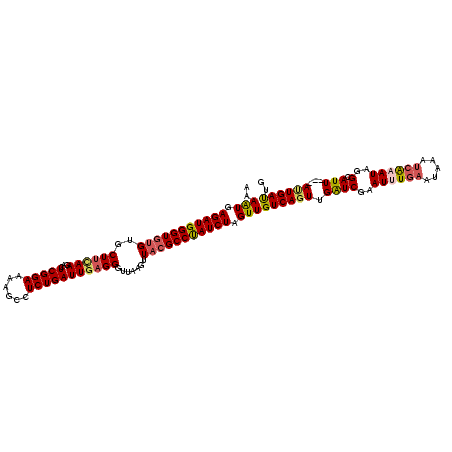
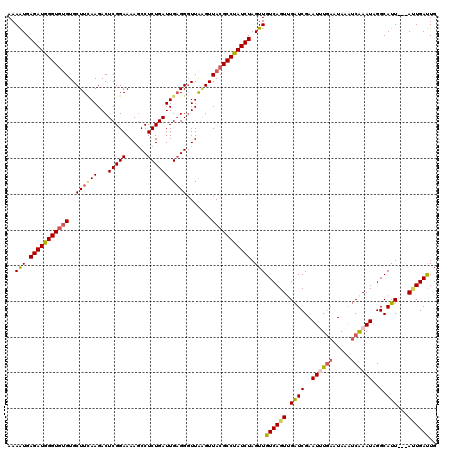
| Location | 15,598,738 – 15,598,858 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.67 |
| Mean single sequence MFE | -28.36 |
| Consensus MFE | -21.66 |
| Energy contribution | -22.42 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.48 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.822343 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15598738 120 + 23771897 CAACUGACAACUAGAUAGGCGUAACCUAACCCUCAAUCAGAGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAA .....(((.......((((.....))))..((((.....)))).........)))(((((((......((((........))))........(((((((....))))))))))))))... ( -28.10) >DroSec_CAF1 21965 119 + 1 CAACUGACAACUAGAUGGGCGUAACGUAACCCACAAUCAGAGGCUUUUCCGAGUCUU-AAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAA ...(((.((...(((((((.((...((.....)).....(((((((....)))))))-......)).))))))).......)).)))..(.((((((((....)))))))))........ ( -28.80) >DroSim_CAF1 21700 120 + 1 CAACUGACAACUAGAUAGGCGUAACUUAAGCCUCAAUCAGAGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAA .................(((.......(((((((.....)))))))......)))(((((((......((((........))))........(((((((....))))))))))))))... ( -31.12) >DroEre_CAF1 21919 118 + 1 CCACUGACAACUAGAUAGGCGUAACUUAACCCUCAAUCAGAGGGUUUUCCGAGUCUUAAAGCAC--ACCCAUCUCACUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAA (((.(((.....((((.((.((.....(((((((.....)))))))......((......))))--.)).))))....))))))..((.(.((((((((....))))))))).))..... ( -27.90) >DroYak_CAF1 23353 120 + 1 CCACUGACAACUAGAUAGGCGUAACUCAACCCUCAAUCAGAGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAA .............((((((.((......)))))..))).(((((((....)))))))(((((......((((........))))........(((((((....))))))))))))..... ( -25.90) >consensus CAACUGACAACUAGAUAGGCGUAACUUAACCCUCAAUCAGAGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAA .....(((.......((((.....))))..((((.....)))).........)))(((((((......((((........))))........(((((((....))))))))))))))... (-21.66 = -22.42 + 0.76)

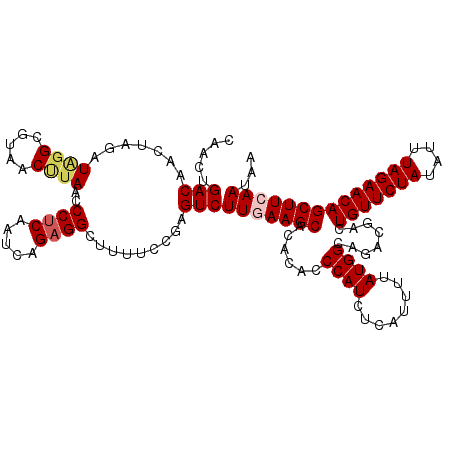
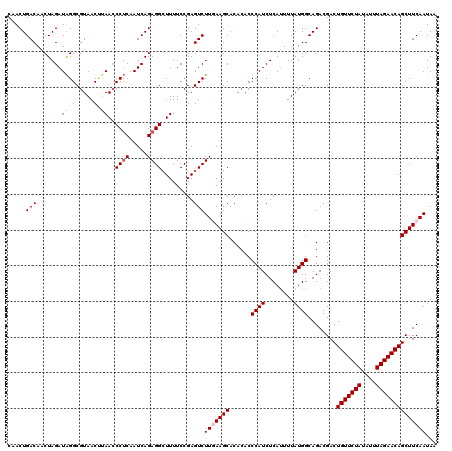
| Location | 15,598,738 – 15,598,858 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.67 |
| Mean single sequence MFE | -37.94 |
| Consensus MFE | -32.36 |
| Energy contribution | -32.72 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.85 |
| SVM decision value | 3.00 |
| SVM RNA-class probability | 0.998060 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15598738 120 - 23771897 UUAUUGAAGCUGUUCUAAAUAUAGAACAGUCGUCUGCCAUAAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGGUUAGGUUACGCCUAUCUAGUUGUCAGUUG ..(((((.(((((((((....)))))))))............(((.(((((((((((..((((((...(((((......))))))))))).......))))))))))).))).))))).. ( -35.00) >DroSec_CAF1 21965 119 - 1 UUAUUGAAGCUGUUCUAAAUAUAGAACAGUCGUCUGCCAUAAAAUGAGAUGGGUGUGUGCUU-AAGACUCGGAAAAGCCUCUGAUUGUGGGUUACGUUACGCCCAUCUAGUUGUCAGUUG ..(((((.(((((((((....)))))))))............(((.(((((((((((.((((-(....(((((......)))))...))))).....))))))))))).))).))))).. ( -37.40) >DroSim_CAF1 21700 120 - 1 UUAUUGAAGCUGUUCUAAAUAUAGAACAGUCGUCUGCCAUAAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGCUUAAGUUACGCCUAUCUAGUUGUCAGUUG ..(((((.(((((((((....)))))))))............(((.(((((((((((.((((............(((((((.....)))))))))))))))))))))).))).))))).. ( -39.30) >DroEre_CAF1 21919 118 - 1 UUAUUGAAGCUGUUCUAAAUAUAGAACAGUCGUCUGCCAUAAAGUGAGAUGGGU--GUGCUUUAAGACUCGGAAAACCCUCUGAUUGAGGGUUAAGUUACGCCUAUCUAGUUGUCAGUGG .((((((.(((((((((....)))))))))................((((((((--((((((............(((((((.....)))))))))).))))))))))).....)))))). ( -40.00) >DroYak_CAF1 23353 120 - 1 UUAUUGAAGCUGUUCUAAAUAUAGAACAGUCGUCUGCCAUAAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGGUUGAGUUACGCCUAUCUAGUUGUCAGUGG .((((((.(((((((((....)))))))))............(((.(((((((((((.(((.(((..(((((...((...))..)))))..))))))))))))))))).))).)))))). ( -38.00) >consensus UUAUUGAAGCUGUUCUAAAUAUAGAACAGUCGUCUGCCAUAAAAUGAGAUGGGUGUGUGCUUCAAGACUCGGAAAAGCCUCUGAUUGAGGGUUAAGUUACGCCUAUCUAGUUGUCAGUUG ..(((((.(((((((((....)))))))))............(((.(((((((((((..((((((...(((((......))))))))))).......))))))))))).))).))))).. (-32.36 = -32.72 + 0.36)

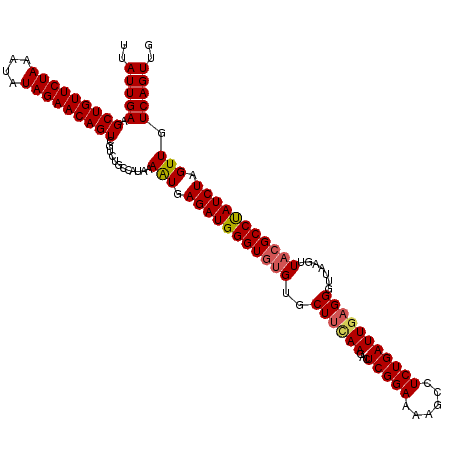
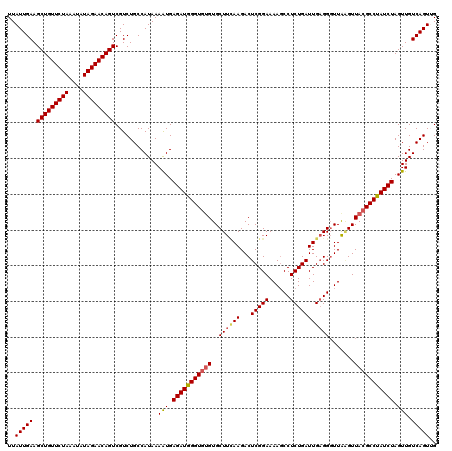
| Location | 15,598,778 – 15,598,898 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.75 |
| Mean single sequence MFE | -24.94 |
| Consensus MFE | -23.04 |
| Energy contribution | -23.64 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.921245 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15598778 120 + 23771897 AGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAACAGUCAAUUAGGCCGCUUGUUUUCAGCAUCUAUAAACAUU .(((((....((.(.(((((((......((((........))))........(((((((....))))))))))))))....).))....)))))(((.......)))............. ( -26.40) >DroSec_CAF1 22005 119 + 1 AGGCUUUUCCGAGUCUU-AAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAACAGUCAAUUAGGCCGCUUGUUUUCAGCAUCUAUAAACAUU .(((((....((.(.((-(.........((((........))))..((.(.((((((((....))))))))).))..))).).))....)))))(((.......)))............. ( -23.00) >DroSim_CAF1 21740 120 + 1 AGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAACAGUCAAUUAGGCCGCUUGUUUUCAGCAUCUAUAAACAUU .(((((....((.(.(((((((......((((........))))........(((((((....))))))))))))))....).))....)))))(((.......)))............. ( -26.40) >DroEre_CAF1 21959 118 + 1 AGGGUUUUCCGAGUCUUAAAGCAC--ACCCAUCUCACUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAACAGUCAAUUAGGCCGCUUGUUUUUAGCAUCUAUAAACAUU ..(((((...(((((.(((((...--..........))))).))).((.(.((((((((....))))))))).))........))....)))))...(((((.(((...))).))))).. ( -22.52) >DroYak_CAF1 23393 120 + 1 AGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAACAGUCAAUUAGGCCGCUUGUUUUCAGCAUCUAUAAACAUU .(((((....((.(.(((((((......((((........))))........(((((((....))))))))))))))....).))....)))))(((.......)))............. ( -26.40) >consensus AGGCUUUUCCGAGUCUUGAAGCACACACCCAUCUCAUUUUAUGGCAGACGACUGUUCUAUAUUUAGAACAGCUUCAAUAACAGUCAAUUAGGCCGCUUGUUUUCAGCAUCUAUAAACAUU .(((((....((.(.(((((((......((((........))))........(((((((....))))))))))))))....).))....)))))(((.......)))............. (-23.04 = -23.64 + 0.60)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:57:41 2006