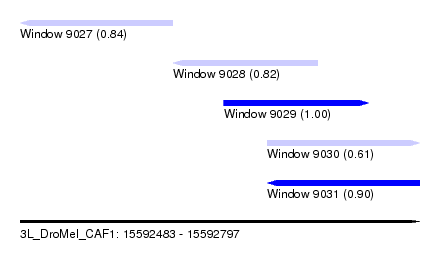
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,592,483 – 15,592,797 |
| Length | 314 |
| Max. P | 0.998640 |

| Location | 15,592,483 – 15,592,603 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 99.67 |
| Mean single sequence MFE | -26.67 |
| Consensus MFE | -26.59 |
| Energy contribution | -26.43 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.44 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.843600 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15592483 120 - 23771897 CAUAAUUGGGCAGCAUAUUUCAAUAAUUGAAGCGCGCUAUAUUUUUAAUUAGUUAAAUGCGCGUUUAAUUAGCUUUAUAAAUUAUGCACAUUCAGCGGUUAGUUGCAGUCCAUCUCGCUC ......(((((.(((((......((((((((.(((((......(((((....))))).)))))))))))))...........))))).....((((.....))))..)))))........ ( -26.33) >DroSec_CAF1 15880 120 - 1 CAUAAUUGGGCAGCAUAUUUCAAUAAUUGAAGCGCGCUAUAUUUUUAAUUAGUUAAAUGCGCGUUUAAUUAGCUUUAUAAAUUAUGCACAUUCAGCGGUUAGUUGCAGUCCAUCUCGCUC ......(((((.(((((......((((((((.(((((......(((((....))))).)))))))))))))...........))))).....((((.....))))..)))))........ ( -26.33) >DroSim_CAF1 15829 120 - 1 CAUAAUUGGGCAGCAUAUUUCAAUAAUUGAAGCGCGCUAUAUUUUUAAUUAGUUAAAUGCGCGUUUAAUUAGCUUUAUAAAUUAUGCACAUUCAGCGGUUAGUUGCAGUCCAUCUCGCUC ......(((((.(((((......((((((((.(((((......(((((....))))).)))))))))))))...........))))).....((((.....))))..)))))........ ( -26.33) >DroEre_CAF1 15891 120 - 1 CAUAAUUGGGCAGCAUAUUUCAAUAAUUGAAGCGCGCUAUAUUUUUAAUUAGUUAAAUGCGCGUUUAAUUAGCUUUAUAAAUUAUGCACAUUCAGCGGUUAGUUGCAGUCCAUCUCGCUC ......(((((.(((((......((((((((.(((((......(((((....))))).)))))))))))))...........))))).....((((.....))))..)))))........ ( -26.33) >DroYak_CAF1 17095 120 - 1 CAUAAUUGGGCAGCAUAUUUCAAUAAUUGAAGCGCGCUAUAUUUUUAAUUAGUUAAAUGCGCGUUUAAUUAGCUUUAUAAAUUAUGCACAUUCAGCGGUUAGUCGCAGUCCAUCUCGCUC ......(((((.(((((......((((((((.(((((......(((((....))))).)))))))))))))...........))))).......((((....)))).)))))........ ( -28.03) >consensus CAUAAUUGGGCAGCAUAUUUCAAUAAUUGAAGCGCGCUAUAUUUUUAAUUAGUUAAAUGCGCGUUUAAUUAGCUUUAUAAAUUAUGCACAUUCAGCGGUUAGUUGCAGUCCAUCUCGCUC ......(((((.(((((......((((((((.(((((......(((((....))))).)))))))))))))...........))))).......((((....)))).)))))........ (-26.59 = -26.43 + -0.16)

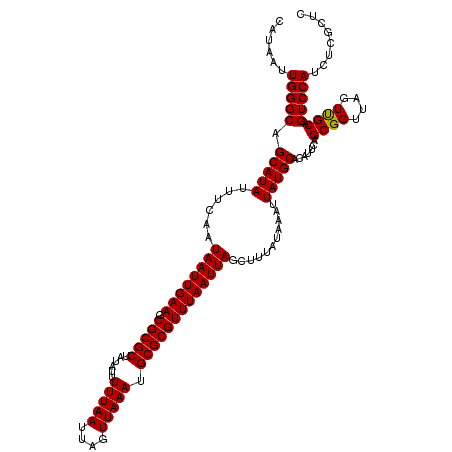
| Location | 15,592,603 – 15,592,717 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.44 |
| Mean single sequence MFE | -35.30 |
| Consensus MFE | -28.82 |
| Energy contribution | -28.42 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.67 |
| SVM RNA-class probability | 0.816932 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15592603 114 - 23771897 GUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGCUAGGGAAUCGAAUCUGUUCAUGU--AU----GUGUGUGUGCACUGCAUAUAUUUUAAUUGCAAAUUUCCUCCGACUGCAU (((((((((..((((......))))..))))))..((((..(((((((.(((....)))((((--((----(((((....))).))))))))...........))))))))))).))).. ( -32.10) >DroSec_CAF1 16000 118 - 1 GUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGCUAGGGAAUCGAAUCUGUGCAUAU--AUGUAGGUGUGUGUGCACUGCAUGUAUUUUAAUUGCAAAUUUCCUCGGACUGCAU ...((((((..((((......))))..))))))...((((..(((((........((((((((--((......))))))))))((((...........))))...)))))..).)))... ( -35.70) >DroSim_CAF1 15949 118 - 1 GUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGCUAGGGAAUCGAAUCUGUGCAUAU--AUGUAGGUGUGUGUGCACUGCAUGUAUUUUAAUUGCAAAUUUCCUCCGACUGCAU (((((((((..((((......))))..))))))..((((..(((((((.......((((((((--((......))))))))))((((...........)))).))))))))))).))).. ( -37.90) >DroEre_CAF1 16011 118 - 1 GUAUGCGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGCUAGGGAAUCGAACCUGUGUAUGUAGGUGUAG--GUGUGUGCACUGCAUAUAUUUUAAUUGCGAAUUUCCUCCGACUGCAU (((((((((..((((......))))..))))))..((((..(((((((((.....((((((((((.((((.--.....))))))))))))))........)).))))))))))).))).. ( -38.12) >DroYak_CAF1 17215 118 - 1 GUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGCUGGGGAAACGAAUCUGUGUACUCGUAUGUAG--GUGUGUGCACUGCAUAUAUUUUAAUUGCAAAUUUCCUCCGACUGCAU (((((((((..((((......))))..))))))..((((..(((((((((.(((((..((....))..)))--)).)))....((((...........))))..)))))))))).))).. ( -32.70) >consensus GUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGCUAGGGAAUCGAAUCUGUGCAUAU__AUGUAGGUGUGUGUGCACUGCAUAUAUUUUAAUUGCAAAUUUCCUCCGACUGCAU (((((((((..((((......))))..))))))..((((..(((((((.......((((((((............))))))))((((...........)))).))))))))))).))).. (-28.82 = -28.42 + -0.40)

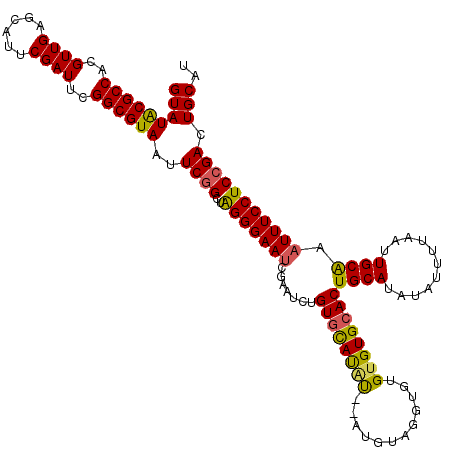
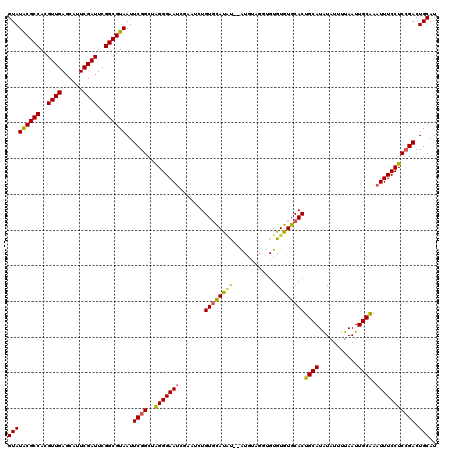
| Location | 15,592,643 – 15,592,757 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.95 |
| Mean single sequence MFE | -28.66 |
| Consensus MFE | -25.40 |
| Energy contribution | -25.48 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.89 |
| SVM decision value | 3.17 |
| SVM RNA-class probability | 0.998640 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15592643 114 + 23771897 CACACACAC----AU--ACAUGAACAGAUUCGAUUCCCUAGCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGUUU .........----..--.((((...(((((((((((....((........)).))))))))).))...)))).(((((((((((((((......)))))....))))))))))....... ( -30.10) >DroSec_CAF1 16040 118 + 1 CACACACACCUACAU--AUAUGCACAGAUUCGAUUCCCUAGCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUU .........((((..--....(((....((((((((....((........)).))))))))))).....))))(((((((((((((((......)))))....))))))))))....... ( -28.80) >DroSim_CAF1 15989 118 + 1 CACACACACCUACAU--AUAUGCACAGAUUCGAUUCCCUAGCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUU .........((((..--....(((....((((((((....((........)).))))))))))).....))))(((((((((((((((......)))))....))))))))))....... ( -28.80) >DroEre_CAF1 16051 118 + 1 CACACAC--CUACACCUACAUACACAGGUUCGAUUCCCUAGCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGCAUACGUAACGCUAAUCAAAGUGUAUGUUACGUAUACGACAGCUU .......--.(((....((((((((..(((((((((....((........)).)))))))))((...(((((.....)))))...))........))))))))...)))........... ( -26.40) >DroYak_CAF1 17255 118 + 1 CACACAC--CUACAUACGAGUACACAGAUUCGUUUCCCCAGCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUU .......--........(((((....((((((........((........))))))))...)))))...((..(((((((((((((((......)))))....))))))))))...)).. ( -29.20) >consensus CACACACACCUACAU__ACAUGCACAGAUUCGAUUCCCUAGCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUU ...........................(((((((((....((........)).))))))))).......((..(((((((((((((((......)))))....))))))))))...)).. (-25.40 = -25.48 + 0.08)

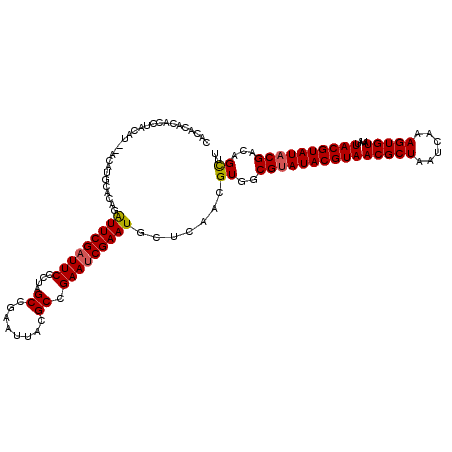
| Location | 15,592,677 – 15,592,797 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.58 |
| Mean single sequence MFE | -33.12 |
| Consensus MFE | -30.96 |
| Energy contribution | -31.16 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.21 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.614024 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15592677 120 + 23771897 GCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGUUUGACAUGUGUGAGGGUACAAGCUAAUUCUGGUAGCUGUUAU (((((((((.((((...)).((.(((((((((.(((((((((((((((......)))))....)))))))))).........))))).)))).))....)))))))).)))......... ( -32.90) >DroSec_CAF1 16078 120 + 1 GCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUUGACAUGUGUGAUGGUACAAGCUAAUUCUGGUAGCUGUUAU (((((((((.((...((((.(((.((((.((..(((((((((((((((......)))))....))))))))))...)))))))))...)))).......)))))))).)))......... ( -33.90) >DroSim_CAF1 16027 120 + 1 GCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUUGACAUGUGUGAUGGUACAAGCUAAUUCUGGUAGCUGUUAU (((((((((.((...((((.(((.((((.((..(((((((((((((((......)))))....))))))))))...)))))))))...)))).......)))))))).)))......... ( -33.90) >DroEre_CAF1 16089 120 + 1 GCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGCAUACGUAACGCUAAUCAAAGUGUAUGUUACGUAUACGACAGCUUGACAUGUGUGAGGGUACAUGCUAAUUCUGGUAGCUGUUAU .........(((((....((....))....))))).((((((((((((......)))....)))))))))..(((((((.((((((((......)))))).....))....))))))).. ( -29.00) >DroYak_CAF1 17293 120 + 1 GCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUUGACAUGUGUGAGGGUACACGCUAAUUCUGGUAGCUGGUAU (((((((((.((........(((.((((.((..(((((((((((((((......)))))....))))))))))...)))))))))(((((....))))))))))))).)))......... ( -35.90) >consensus GCCGAAUUACGCCGAAUCGAAUGCUCAACGUGGCGUAUACGUAACGCUAAUCAAAGUGUAUAUUACGUAUACGACAGCUUGACAUGUGUGAGGGUACAAGCUAAUUCUGGUAGCUGUUAU (((((((((((((...(((.(((.((((..((.(((((((((((((((......)))))....)))))))))).))..)))))))...))).)))....).)))))).)))......... (-30.96 = -31.16 + 0.20)



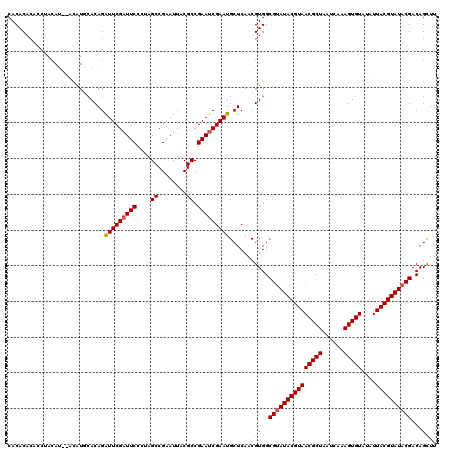
| Location | 15,592,677 – 15,592,797 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.58 |
| Mean single sequence MFE | -30.31 |
| Consensus MFE | -27.81 |
| Energy contribution | -28.09 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.900082 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15592677 120 - 23771897 AUAACAGCUACCAGAAUUAGCUUGUACCCUCACACAUGUCAAACUGUCGUAUACGUAAUAUACACUUUGAUUAGCGUUACGUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGC ..........((.((((((((((((......))).((((..(((((.((((((((((((................)))))))))))).)).)))..)))).......))).)))))))). ( -27.29) >DroSec_CAF1 16078 120 - 1 AUAACAGCUACCAGAAUUAGCUUGUACCAUCACACAUGUCAAGCUGUCGUAUACGUAAUAUACACUUUGAUUAGCGUUACGUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGC ..........((.(((((((((((...(((.....))).))))))(.((((((((((((................)))))))))))).)(((((((((....).))))))))))))))). ( -30.59) >DroSim_CAF1 16027 120 - 1 AUAACAGCUACCAGAAUUAGCUUGUACCAUCACACAUGUCAAGCUGUCGUAUACGUAAUAUACACUUUGAUUAGCGUUACGUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGC ..........((.(((((((((((...(((.....))).))))))(.((((((((((((................)))))))))))).)(((((((((....).))))))))))))))). ( -30.59) >DroEre_CAF1 16089 120 - 1 AUAACAGCUACCAGAAUUAGCAUGUACCCUCACACAUGUCAAGCUGUCGUAUACGUAACAUACACUUUGAUUAGCGUUACGUAUGCGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGC ...((((((..........((((((........))))))..)))))).(((((((((((................)))))))))))((((((((((((....).)))))))).....))) ( -32.09) >DroYak_CAF1 17293 120 - 1 AUACCAGCUACCAGAAUUAGCGUGUACCCUCACACAUGUCAAGCUGUCGUAUACGUAAUAUACACUUUGAUUAGCGUUACGUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGC ..........((.((((((((((((......))))((((..(((((.((((((((((((................)))))))))))).)).)))..))))........)).)))))))). ( -30.99) >consensus AUAACAGCUACCAGAAUUAGCUUGUACCCUCACACAUGUCAAGCUGUCGUAUACGUAAUAUACACUUUGAUUAGCGUUACGUAUACGCCACGUUGAGCAUUCGAUUCGGCGUAAUUCGGC ..........((.(((((((((((.((..........))))))))(.((((((((((((................)))))))))))).)(((((((((....).))))))))))))))). (-27.81 = -28.09 + 0.28)

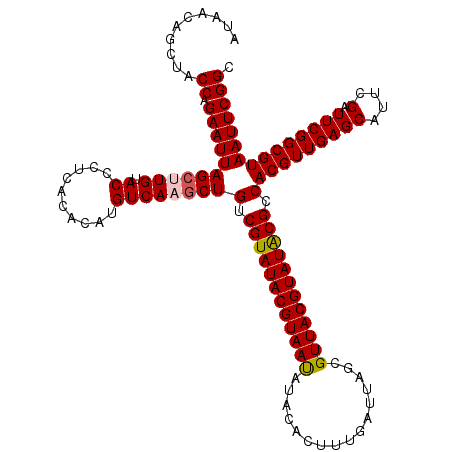
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:57:15 2006