
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,590,097 – 15,590,254 |
| Length | 157 |
| Max. P | 0.999733 |

| Location | 15,590,097 – 15,590,215 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.40 |
| Mean single sequence MFE | -35.28 |
| Consensus MFE | -33.84 |
| Energy contribution | -34.16 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.96 |
| SVM decision value | 3.97 |
| SVM RNA-class probability | 0.999733 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15590097 118 - 23771897 CUGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCAAGCGAA--GCAAAAGAAAUUGGCUUUUGUUUAGACCGGAA (((((((.((((((....)))))))))..(((((((((..(((.((((((((((((.(((.....))).)))).......)))..)--))))((....))))).))))))))).)))).. ( -34.21) >DroSec_CAF1 13354 120 - 1 CCGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCAUGCGAAGAGCAAAAGAAAUUGGCUUUUGUUUAGACCGGAA (((((((.((((((....)))))))))..(((((((((((((((((((....))..))))))))......((((((.((.(((.....)))...))..))))))))))))))).)))).. ( -38.00) >DroSim_CAF1 13314 120 - 1 CCGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCAUGCGAAGAGCAAAAGAAAUUGGCUUUUGUUUAGACCGGAA (((((((.((((((....)))))))))..(((((((((((((((((((....))..))))))))......((((((.((.(((.....)))...))..))))))))))))))).)))).. ( -38.00) >DroEre_CAF1 13320 118 - 1 CGGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCGAAUCAUGCGAA--GCAAACGAAAUUGGCUUUUGUUUAGACCGGAA (((.(((.((((((....)))))))))..(((((((((((((((((((....))..))))))))......((((((.((.(((...--)))...))..))))))))))))))).)))... ( -33.20) >DroYak_CAF1 14642 118 - 1 CCGGCUGCGCUUUUGUUUGAGAGCCAAAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCAUGCAAA--GCAAAAGAAAUUGGCUUUUGUUUAGUCCGGAA ((((.((.((((((....))))))))..((((((((((..(((.(((((..(((...(((((..((.....))...)))))))).)--))))((....))))).)))))))))))))).. ( -33.00) >consensus CCGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCAUGCGAA__GCAAAAGAAAUUGGCUUUUGUUUAGACCGGAA (((((((.((((((....)))))))))..(((((((((((((((((((....))..))))))))......((((((.((.(((.....)))...))..))))))))))))))).)))).. (-33.84 = -34.16 + 0.32)


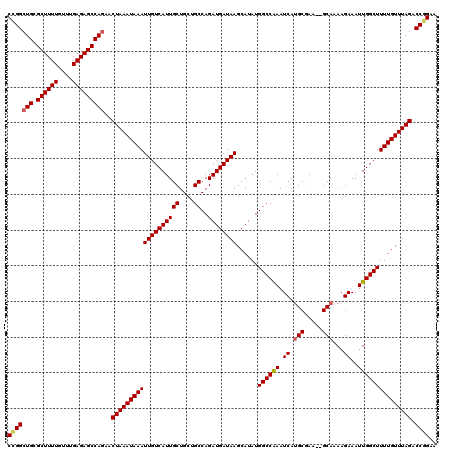
| Location | 15,590,135 – 15,590,254 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.92 |
| Mean single sequence MFE | -30.00 |
| Consensus MFE | -27.62 |
| Energy contribution | -27.50 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.09 |
| SVM RNA-class probability | 0.987584 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15590135 119 + 23771897 UGAUUUGGCCAUAUGCUUAUCAUCUGGCAGCAGCAAUGACAAUUUAUUUAGUUCUGGCUCUCAAACAAAAGCGCAGCCAGGGAA-AAAUGCUAAAAUCAGCGAAUAACAGAGGCCAAAUA ..((((((((...((((........))))...((..(((.....(((((..(((((((((.(........).).))))))))..-)))))......)))))..........)))))))). ( -30.20) >DroSec_CAF1 13394 120 + 1 UGAUUUGGCCAUAUGCUUAUCAUCUGGCAGCAGCAAUGACAAUUUAUUUAGUUCUGGCUCUCAAACAAAAGCGCAGCCGGGGAAAAAAUGCUAAAAUCAGCGAAUAACAGAGGCCAAAUA ..((((((((...((((........))))(((....)).)...((((((..(((((((((.(........).).)))))))).......(((......)))))))))....)))))))). ( -29.50) >DroSim_CAF1 13354 120 + 1 UGAUUUGGCCAUAUGCUUAUCAUCUGGCAGCAGCAAUGACAAUUUAUUUAGUUCUGGCUCUCAAACAAAAGCGCAGCCGGGGAAAAAAUGCUAAAAUCAGCGAAUAACAGAGGCCAAAUA ..((((((((...((((........))))(((....)).)...((((((..(((((((((.(........).).)))))))).......(((......)))))))))....)))))))). ( -29.50) >DroEre_CAF1 13358 120 + 1 UGAUUCGGCCAUAUGCUUAUCAUCUGGCAGCAGCAAUGACAAUUUAUUUAGUUCUGGCUCUCAAACAAAAGCGCAGCCCGGGAAAAAAUGCUAAAAUCAGCGAAUAGCAGAGGCCAAAUA ......((((...((((..((.((((((((.((((((((....)))))..))))))((.((........)).))...))))).......(((......)))))..))))..))))..... ( -26.50) >DroYak_CAF1 14680 120 + 1 UGAUUUGGCCAUAUGCUUAUCAUCUGGCAGCAGCAAUGACAAUUUAUUUAGUUUUGGCUCUCAAACAAAAGCGCAGCCGGGGAAAAAAUGCUAAAAUCAGCGAAUAGCAGAGGCCAAAUA ..((((((((...((((..((.((((((.((.(((((((....)))))...(((((.........))))))))).))))))........(((......)))))..))))..)))))))). ( -34.30) >consensus UGAUUUGGCCAUAUGCUUAUCAUCUGGCAGCAGCAAUGACAAUUUAUUUAGUUCUGGCUCUCAAACAAAAGCGCAGCCGGGGAAAAAAUGCUAAAAUCAGCGAAUAACAGAGGCCAAAUA ..((((((((...((((........))))..............((((((..(((((((((.(........).).)))))))).......(((......)))))))))....)))))))). (-27.62 = -27.50 + -0.12)

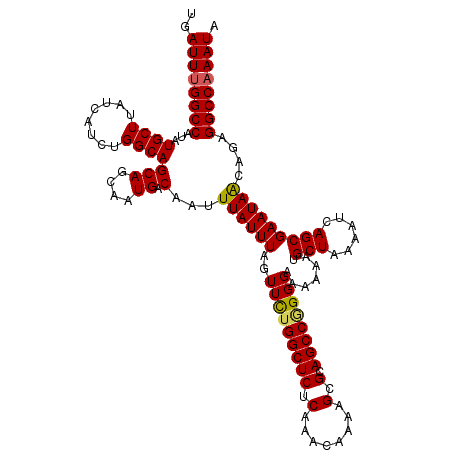
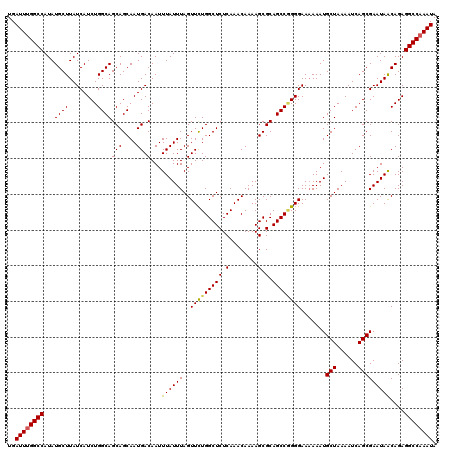
| Location | 15,590,135 – 15,590,254 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.92 |
| Mean single sequence MFE | -32.76 |
| Consensus MFE | -31.82 |
| Energy contribution | -31.62 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.97 |
| SVM decision value | 3.33 |
| SVM RNA-class probability | 0.999022 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15590135 119 - 23771897 UAUUUGGCCUCUGUUAUUCGCUGAUUUUAGCAUUU-UUCCCUGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCA .((((((((..((((..((((((....))))....-....(((((.((((((((....))))))..((.(..........)))......)).)))))..))..))))...)))))))).. ( -33.20) >DroSec_CAF1 13394 120 - 1 UAUUUGGCCUCUGUUAUUCGCUGAUUUUAGCAUUUUUUCCCCGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCA .((((((((..((((....((((....)))).............(((.((((((....)))))))))............(((((((((....))..)))))))))))...)))))))).. ( -31.50) >DroSim_CAF1 13354 120 - 1 UAUUUGGCCUCUGUUAUUCGCUGAUUUUAGCAUUUUUUCCCCGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCA .((((((((..((((....((((....)))).............(((.((((((....)))))))))............(((((((((....))..)))))))))))...)))))))).. ( -31.50) >DroEre_CAF1 13358 120 - 1 UAUUUGGCCUCUGCUAUUCGCUGAUUUUAGCAUUUUUUCCCGGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCGAAUCA .((((((((..((((....((((....))))..........(..(((.((((((....)))))))))..).........(((((((((....))..)))))))))))...)))))))).. ( -35.70) >DroYak_CAF1 14680 120 - 1 UAUUUGGCCUCUGCUAUUCGCUGAUUUUAGCAUUUUUUCCCCGGCUGCGCUUUUGUUUGAGAGCCAAAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCA .((((((((..((((..((((((....))))...........(((.((((((((....))))))................((....)).)).)))....))..))))...)))))))).. ( -31.90) >consensus UAUUUGGCCUCUGUUAUUCGCUGAUUUUAGCAUUUUUUCCCCGGCUGCGCUUUUGUUUGAGAGCCAGAACUAAAUAAAUUGUCAUUGCUGCUGCCAGAUGAUAAGCAUAUGGCCAAAUCA .((((((((..((((....((((....)))).............(((.((((((....)))))))))............(((((((((....))..)))))))))))...)))))))).. (-31.82 = -31.62 + -0.20)

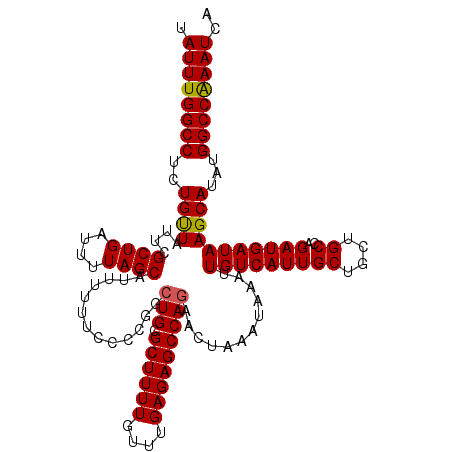
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:57:00 2006