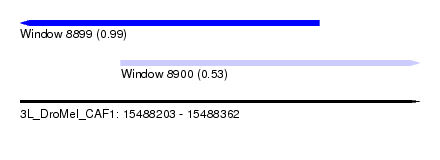
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,488,203 – 15,488,362 |
| Length | 159 |
| Max. P | 0.987360 |

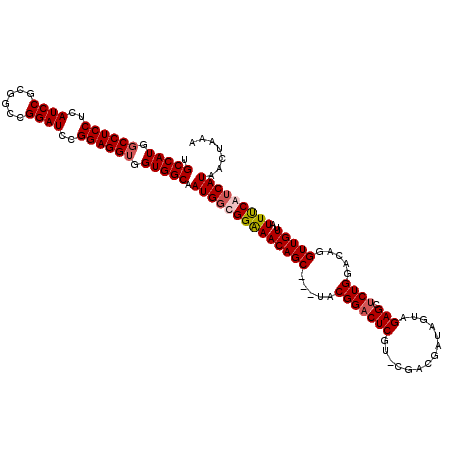
| Location | 15,488,203 – 15,488,322 |
|---|---|
| Length | 119 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.32 |
| Mean single sequence MFE | -46.14 |
| Consensus MFE | -40.43 |
| Energy contribution | -41.24 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.08 |
| SVM RNA-class probability | 0.987360 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15488203 119 - 23771897 UGCCAUGGCCUCCUCAUCCCCAUCCGGAUCCGGAGGUGGUGGCAAUGGCGGGAACAGCUCCUACGGACUCGU-CGACGAUAGUAGAGCUCUGGACAGGUUGUUAUUCCAUCAUAACUAAA ((((((.((((((..((((......))))..)))))).))))))(((..((((((((((....(((((((..-..((....)).))).))))....))))))..))))..)))....... ( -46.80) >DroSec_CAF1 29326 116 - 1 UGCCAUGGCCUCCUCAUCCGCGGCUGGAUCCGGAGGUGGUGGCAAUGGUGGAAACAGC---UACGGACUCGU-CGACGAUAGUAGAGCUCUGGACAGGUUGUUAUUUCAUCAUAACUAAA ((((((.((((((..(((((....)))))..)))))).))))))((((((((((((((---(.(((((((..-..((....)).))).))))....))))))..)))))))))....... ( -48.30) >DroSim_CAF1 31351 117 - 1 UGCCAUGGCCUCCUCAUCCGCGGCUGGAUCCGGAGGUGGUGGCAAUGGUGGAAACAGC---UACGGACUCGUUUGAGGAUAGUAGAGCUCUGGACAGGUUGUUAUUUCAUCAUAACUAAA ((((((.((((((..(((((....)))))..)))))).))))))((((((((((((((---(.(((((((..............))).))))....))))))..)))))))))....... ( -46.64) >DroYak_CAF1 111696 119 - 1 CGCCAUGGCCUCCUCAUCCACGGCCGGAUCCGGAGGAGGUGGCAAUGCCGGGAACAGCUCCUACGGACUCGU-CGACGACAGUAGAGCCCUGGACAGGUUGUUAUUUCAUCAUAACUAAA .(((((..(((((..((((......))))..)))))..)))))..(((((((....((((.(((....(((.-...)))..)))))))))))).))((((((.........))))))... ( -42.80) >consensus UGCCAUGGCCUCCUCAUCCGCGGCCGGAUCCGGAGGUGGUGGCAAUGGCGGAAACAGC___UACGGACUCGU_CGACGAUAGUAGAGCUCUGGACAGGUUGUUAUUUCAUCAUAACUAAA .(((((.((((((..((((......))))..)))))).))))).((((((((((((((.....(((((((..............))).)))).....)))))..)))))))))....... (-40.43 = -41.24 + 0.81)

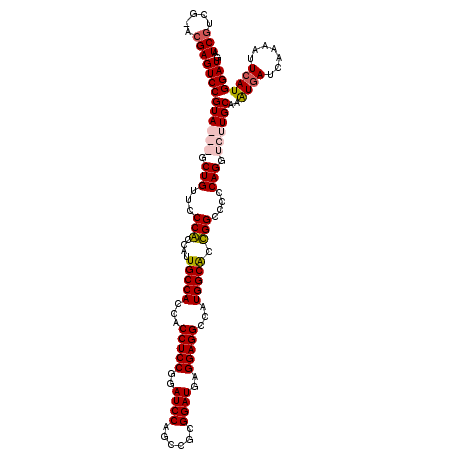
| Location | 15,488,243 – 15,488,362 |
|---|---|
| Length | 119 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.36 |
| Mean single sequence MFE | -42.81 |
| Consensus MFE | -37.20 |
| Energy contribution | -38.32 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.87 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.527828 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15488243 119 + 23771897 AUCGUCG-ACGAGUCCGUAGGAGCUGUUCCCGCCAUUGCCACCACCUCCGGAUCCGGAUGGGGAUGAGGAGGCCAUGGCAUCGGCCCUCAGGUCUUGCAAAUGAUCAAAAUUCAUGGAUG .(((...-.)))((((((((((.(((.....(((..(((((...(((((..((((......))))..)))))...)))))..)))...))).))))))..((((.......)))))))). ( -48.20) >DroSec_CAF1 29366 116 + 1 AUCGUCG-ACGAGUCCGUA---GCUGUUUCCACCAUUGCCACCACCUCCGGAUCCAGCCGCGGAUGAGGAGGCCAUGGCACUGGCCCCCAGGCCUUGCAAAUGAUCAAAAUUCAUGGAUG ((((((.-.(..(((((..---((((..(((..................)))..))))..)))))..)..)))(((((((..((((....)))).))).(((.......)))))))))). ( -38.47) >DroSim_CAF1 31391 117 + 1 AUCCUCAAACGAGUCCGUA---GCUGUUUCCACCAUUGCCACCACCUCCGGAUCCAGCCGCGGAUGAGGAGGCCAUGGCACUGGCCCCCAGGUCUUGCAAGUGAUCAAAAUUCAUGGAUG ((((........(((((..---((((..(((..................)))..))))..)))))((((.(((((......))))).)).((((........)))).....))..)))). ( -38.87) >DroYak_CAF1 111736 119 + 1 GUCGUCG-ACGAGUCCGUAGGAGCUGUUCCCGGCAUUGCCACCUCCUCCGGAUCCGGCCGUGGAUGAGGAGGCCAUGGCGCCGGCCCUCAGGUCUUGCAAAUAAUCAAAAUUCAUGGAUG .(((...-.)))((((((((((.(((...(((((...(((((((((((...(((((....))))))))))))...)))))))))....))).)))))).(((.......)))...)))). ( -45.70) >consensus AUCGUCG_ACGAGUCCGUA___GCUGUUCCCACCAUUGCCACCACCUCCGGAUCCAGCCGCGGAUGAGGAGGCCAUGGCACCGGCCCCCAGGUCUUGCAAAUGAUCAAAAUUCAUGGAUG .(((.....)))((((((((((.(((...(((....(((((...(((((..((((......))))..)))))...))))).)))....))).))))))..((((.......)))))))). (-37.20 = -38.32 + 1.12)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:55:07 2006