
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,248,099 – 15,248,249 |
| Length | 150 |
| Max. P | 0.999607 |

| Location | 15,248,099 – 15,248,211 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 91.11 |
| Mean single sequence MFE | -29.38 |
| Consensus MFE | -24.45 |
| Energy contribution | -25.25 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.66 |
| Structure conservation index | 0.83 |
| SVM decision value | 3.78 |
| SVM RNA-class probability | 0.999607 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
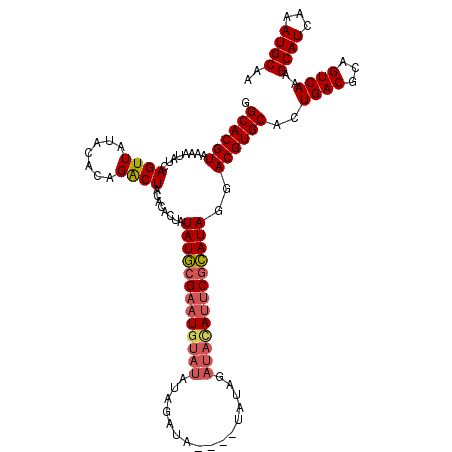
>3L_DroMel_CAF1 15248099 112 + 23771897 GGCACGUAAAAUAUCAGUUAUACACAGACUACACACUAUAUGCGAAUGUCUAUAGAUA----UAUAGAUACAUUCGCAUAGGACGUGCACUGACGCAGUCAAAGCAUCAAAUGCAA .((((((........((((.......))))........((((((((((((((((....----))))))..))))))))))..))))))..((((...))))..((((...)))).. ( -32.20) >DroSec_CAF1 137299 110 + 1 GGCACGUAAAAUAUCAGUUAUACACAGACUACACACUAUAUGCGAAUGUA--UAGACA----UAUAGAUACAUUCGCAUAGGACGUGCACUGACGCAGUCAAAGCAUCAAAUGCAA .((((((........((((.......))))........((((((((((((--(.....----.....)))))))))))))..))))))..((((...))))..((((...)))).. ( -30.50) >DroSim_CAF1 145062 112 + 1 GGCACGUAAAAUAUCAGUUAUACACAGACUACACACUAUAUGCGAAUGUAUAUAGAUA----UAUAGAUACAUUCGCAUAGGACGUGCACUGACGCAGUCAAAGCAUCAAAUGCAA .((((((........((((.......))))........(((((((((((((.((....----..)).)))))))))))))..))))))..((((...))))..((((...)))).. ( -31.70) >DroEre_CAF1 137245 116 + 1 GGCACGUAAAAUAUCAGUUAUACACAGACUACACACUAUAUGCGAAUGUAUUUAGAUACAUACAUAGAUAUAUUCGCAUAGGACGUGCACUGACGCAGUCAAAGCAUCAAAUGCAG .((((((........((((.......))))........((((((((((((((((..........))))))))))))))))..))))))..((((...))))..((((...)))).. ( -32.30) >DroAna_CAF1 129780 106 + 1 GGCACGUAAAAUAUCAGUUAUACACAGGCUACACACUAUAUAAG--UUCAUAUAGAUA----U----AUAUAUUCGUAUAGGACGUGCAGUGACGCAGUCAAAGCAUCAAAUGCAA .((((((........((((.......)))).....(((((..((--(..(((((....----)----)))))))..))))).))))))..((((...))))..((((...)))).. ( -20.20) >consensus GGCACGUAAAAUAUCAGUUAUACACAGACUACACACUAUAUGCGAAUGUAUAUAGAUA____UAUAGAUACAUUCGCAUAGGACGUGCACUGACGCAGUCAAAGCAUCAAAUGCAA .((((((........((((.......))))........(((((((((((((................)))))))))))))..))))))..((((...))))..((((...)))).. (-24.45 = -25.25 + 0.80)

| Location | 15,248,099 – 15,248,211 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 91.11 |
| Mean single sequence MFE | -32.28 |
| Consensus MFE | -26.87 |
| Energy contribution | -27.99 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.26 |
| Structure conservation index | 0.83 |
| SVM decision value | 3.56 |
| SVM RNA-class probability | 0.999384 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15248099 112 - 23771897 UUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA----UAUCUAUAGACAUUCGCAUAUAGUGUGUAGUCUGUGUAUAACUGAUAUUUUACGUGCC .((((((.(((((.......))))))))))).....((((((((((..((((((----....))))))))))))))))..(..(((((((.((.....)).)))....))))..). ( -35.60) >DroSec_CAF1 137299 110 - 1 UUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA----UGUCUA--UACAUUCGCAUAUAGUGUGUAGUCUGUGUAUAACUGAUAUUUUACGUGCC .((((((.(((((.......))))))))))).....(((((((((((((.....----.....)--))))))))))))..(..(((((((.((.....)).)))....))))..). ( -34.10) >DroSim_CAF1 145062 112 - 1 UUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA----UAUCUAUAUACAUUCGCAUAUAGUGUGUAGUCUGUGUAUAACUGAUAUUUUACGUGCC .((((((.(((((.......))))))))))).....(((((((((((((.((..----....)).)))))))))))))..(..(((((((.((.....)).)))....))))..). ( -35.10) >DroEre_CAF1 137245 116 - 1 CUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUAUAUCUAUGUAUGUAUCUAAAUACAUUCGCAUAUAGUGUGUAGUCUGUGUAUAACUGAUAUUUUACGUGCC ..((((.((((((.......))))))(((((((..(((((((..(((((..((((((........))))))....))))).)))))))..)))))))..............)))). ( -30.70) >DroAna_CAF1 129780 106 - 1 UUGCAUUUGAUGCUUUGACUGCGUCACUGCACGUCCUAUACGAAUAUAU----A----UAUCUAUAUGAA--CUUAUAUAGUGUGUAGCCUGUGUAUAACUGAUAUUUUACGUGCC ..((((.((((((.......)))))).((((((..(((((((..(((((----(----..((.....)).--..)))))).)))))))..))))))...............)))). ( -25.90) >consensus UUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA____UAUCUAUAUACAUUCGCAUAUAGUGUGUAGUCUGUGUAUAACUGAUAUUUUACGUGCC .((((((.(((((.......))))))))))).....(((((((((((((................)))))))))))))..(..(((((...((((.......)))).)))))..). (-26.87 = -27.99 + 1.12)



| Location | 15,248,139 – 15,248,249 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 86.74 |
| Mean single sequence MFE | -26.12 |
| Consensus MFE | -18.61 |
| Energy contribution | -19.77 |
| Covariance contribution | 1.16 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.894931 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15248139 110 - 23771897 CAGAACAGAAC--ACAUCUCUUUCUUAGUCAUUGUUCAAGUUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA----UAUCUAUAGACAUUCGCA .((.((.((((--(.((..((.....))..)))))))..((.(((((.(((((.......)))))))))))))).)).((((((((..((((((----....)))))))))))))) ( -31.70) >DroSec_CAF1 137339 108 - 1 CAGAACAGAAC--CCACCUCUUUCUUAGUCAUUGUUCAAGUUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA----UGUCUA--UACAUUCGCA .((.((.((((--......((.....)).....))))..((.(((((.(((((.......)))))))))))))).)).(((((((((((.....----.....)--)))))))))) ( -28.60) >DroSim_CAF1 145102 110 - 1 CAGAACAGAAC--CCAUCUCUUUCUUAGUCAUUGUUCAAGUUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA----UAUCUAUAUACAUUCGCA .((.((.((((--......((.....)).....))))..((.(((((.(((((.......)))))))))))))).)).(((((((((((.((..----....)).))))))))))) ( -29.60) >DroEre_CAF1 137285 114 - 1 CAGAACUGAAC--CCAUCUCUUUCUUAGUCAUUGUUCAAGCUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUAUAUCUAUGUAUGUAUCUAAAUACAUUCGCA .((.(((((((--......((.....)).....))))).(.((((((.(((((.......)))))))))))))).)).(((((........((((((........))))))))))) ( -24.40) >DroAna_CAF1 129820 101 - 1 -----CAAAGCUGCCAUCCCUUUCUUAGUCAUUGUUCAAGUUGCAUUUGAUGCUUUGACUGCGUCACUGCACGUCCUAUACGAAUAUAU----A----UAUCUAUAUGAA--CUUA -----............................(((((...((((..((((((.......)))))).))))(((.....))).......----.----........))))--)... ( -16.30) >consensus CAGAACAGAAC__CCAUCUCUUUCUUAGUCAUUGUUCAAGUUGCAUUUGAUGCUUUGACUGCGUCAGUGCACGUCCUAUGCGAAUGUAUCUAUA____UAUCUAUAUACAUUCGCA .......((((........((.....)).....))))..(.((((((.(((((.......))))))))))))......(((((((((((................))))))))))) (-18.61 = -19.77 + 1.16)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:53:19 2006