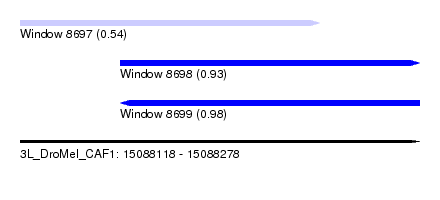
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,088,118 – 15,088,278 |
| Length | 160 |
| Max. P | 0.981661 |

| Location | 15,088,118 – 15,088,238 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.00 |
| Mean single sequence MFE | -52.29 |
| Consensus MFE | -32.57 |
| Energy contribution | -31.77 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.43 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.535392 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15088118 120 + 23771897 CCAGUACAGCAGCUGCCAGCCACCGGACAUGCUGGCGCACGGAUUCUGUGCGGGAUCACUCCACGGAGGCGGCACUAUUCCGGCUGCCACCUGCUGCGGAUUCUGGCCGGUGGUCAGUAG ............((((..((((((((.(.(((....)))((((.((((((((((.....((....))((((((.(......))))))).))))).))))).)))))))))))))..)))) ( -52.50) >DroVir_CAF1 4078 111 + 1 CCAGUAGAGCAGCUGCCAGCCGCCGGCCAUGCUGGCGCACGGAUUCUUUGCCGGGUCACUCCACGGCGGCGGCACAAUCCCGGCCAAUGCCUGCUGUU---GUUGU------GCCAGCAA ........((.((((((.((.((((((...)))))))).(((........)))((.....))..))))))((((((((..((((........))))..---)))))------))).)).. ( -49.90) >DroPse_CAF1 6245 120 + 1 CCAGUAGAGCAGCUGCCAGCCCCCGGACAUGCUGGCGCAGGGGUUCGUGGCCGGAUCACUCCAGGGUGGCGGCACAAUGCCGGCCGCCACCUGAUAGCCGUGCUGUGAGCUGGUCAGCAG ...........(((((((((((((.....(((....))))))).(((..(((((.........((((((((((.(......))))))))))).....))).))..)))))))).)))).. ( -57.74) >DroGri_CAF1 4118 108 + 1 CCAGUAAAGUAAUUGCCAGCCGCCGGCCAUGCUGGCGCAUGGAUUCUUGGCCGGAUCACUCCAUGGCGGCGGCACAAUGCCGGCCAAUGCCUGUU------GUUGU------GUCAGCAA ............((((..((((((((((((((....)))........)))))(((....)))..))))))((((((((..((((....))).)..------)))))------))).)))) ( -44.30) >DroAna_CAF1 4209 120 + 1 CCAGUAGAGGAACUGCCAGCCUCCGGCCAUGCUGGCACAGGGGUUCUUGGCGGGAUCGCUCCAGGGCGGUGGCACAAUGCCAGCCGCCACUUGGUGGGAGUUUUGGGUGGUGGUCAGUAG ((......))..(((((((((((.((((.....))).).)))))...))))))((((((((((((((..((((.....)))).(((((....)))))..)))))))..)))))))..... ( -52.00) >DroPer_CAF1 6394 120 + 1 CCAGUAGAGCAGCUGCCAGCCCCCGGACAUGCUGGCGCAGGGGUUCGUGGCCGGAUCACUCCAGGGUGGCGGCACAAUGCCGGCCGCCACCUGAUAGCCUUGCUGUGAGCUGGUCAGCAG ...((.((.(((((((((((..........))))))((((((.(((......(((....))).((((((((((.(......))))))))))))).).))))))....))))).)).)).. ( -57.30) >consensus CCAGUAGAGCAGCUGCCAGCCCCCGGACAUGCUGGCGCAGGGAUUCUUGGCCGGAUCACUCCAGGGCGGCGGCACAAUGCCGGCCGCCACCUGAUGGC__UGCUGUG_G_UGGUCAGCAG .((((......))))..............(((((((.....((((((....))))))....((.(((((((((.........)))))))))))...................))))))). (-32.57 = -31.77 + -0.80)



| Location | 15,088,158 – 15,088,278 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.78 |
| Mean single sequence MFE | -54.97 |
| Consensus MFE | -33.42 |
| Energy contribution | -33.15 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.96 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.19 |
| SVM RNA-class probability | 0.928066 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15088158 120 + 23771897 GGAUUCUGUGCGGGAUCACUCCACGGAGGCGGCACUAUUCCGGCUGCCACCUGCUGCGGAUUCUGGCCGGUGGUCAGUAGGAGGAGCAGGCAGGUGAGGAUGGAGCUGCCCGCCUGCAGA ....(((((((((.....(((....)))(((((.((((((((.(((((...((((.(.....((((((...)))))).....).))))))))).)).)))))).)))))))))..))))) ( -55.70) >DroPse_CAF1 6285 120 + 1 GGGUUCGUGGCCGGAUCACUCCAGGGUGGCGGCACAAUGCCGGCCGCCACCUGAUAGCCGUGCUGUGAGCUGGUCAGCAGCAACAGCAGGCAGGUAAUCAUGGUUGAGCCCGCCUGGAGG (((((((..(((((((.(((...((((((((((.(......)))))))))))....(((.((((((..((((.....)))).))))))))).))).))).)))))))))))......... ( -60.30) >DroGri_CAF1 4158 108 + 1 GGAUUCUUGGCCGGAUCACUCCAUGGCGGCGGCACAAUGCCGGCCAAUGCCUGUU------GUUGU------GUCAGCAAAAGCAGCAGGCAGGUGAGAAUGCUGCUGCACGCCUGUAUG .(((((......)))))....((.((((((((((((...(..(((..((((((((------(((.(------(....))..))))))))))))))..)..)).)))))).)))))).... ( -42.90) >DroYak_CAF1 5466 120 + 1 GGAUUCUGUGCGGGAUCACUCCACGGUGGGGGCACUAUUCCGGCCGCCACCUGCUGCGGAUGUUGGCCGGUGGUCAGUAGGAGCAGCAGGCAGGUGAGGAUGGAGCUGCCCGCCUGCAGA .((((((....)))))).((.((.(((((((((.((((((..(((....((((((((...((((((((...))))))))...))))))))..)))..)))))).))).)))))))).)). ( -56.90) >DroAna_CAF1 4249 120 + 1 GGGUUCUUGGCGGGAUCGCUCCAGGGCGGUGGCACAAUGCCAGCCGCCACUUGGUGGGAGUUUUGGGUGGUGGUCAGUAGCAGGAGCAGGCAGGUGAGGAUGGAGCUGCCCGCCUGCAGA .((((((....))))))(((((...((..((((.....))))(((((((((..(........)..))))))))).....)).)))))..(((((((.((.........)))))))))... ( -53.70) >DroPer_CAF1 6434 120 + 1 GGGUUCGUGGCCGGAUCACUCCAGGGUGGCGGCACAAUGCCGGCCGCCACCUGAUAGCCUUGCUGUGAGCUGGUCAGCAGCAACAGCAGGCAGGUAAUCAUGGUUGAGCCCGCCUGGAGG (((((((..(((((((.(((...((((((((((.(......)))))))))))....((((.(((((..((((.....)))).))))))))).))).))).)))))))))))......... ( -60.30) >consensus GGAUUCUUGGCCGGAUCACUCCAGGGUGGCGGCACAAUGCCGGCCGCCACCUGAUGGGCAUGCUGGGAGGUGGUCAGCAGCAGCAGCAGGCAGGUGAGGAUGGAGCUGCCCGCCUGCAGA .((((((....))))))....((.(((((((((.........))))))))))).......(((((.........)))))......((((((.(((..((......))))).))))))... (-33.42 = -33.15 + -0.27)

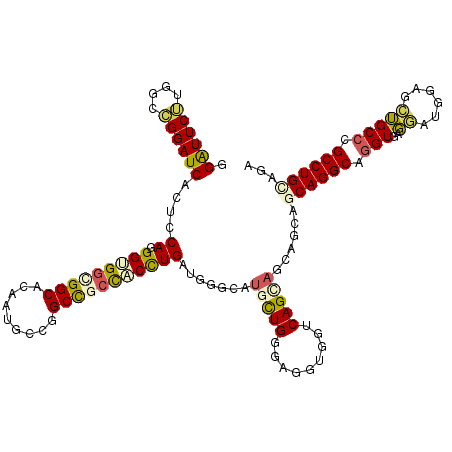
| Location | 15,088,158 – 15,088,278 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.78 |
| Mean single sequence MFE | -48.29 |
| Consensus MFE | -28.84 |
| Energy contribution | -29.67 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.89 |
| SVM RNA-class probability | 0.981661 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 15088158 120 - 23771897 UCUGCAGGCGGGCAGCUCCAUCCUCACCUGCCUGCUCCUCCUACUGACCACCGGCCAGAAUCCGCAGCAGGUGGCAGCCGGAAUAGUGCCGCCUCCGUGGAGUGAUCCCGCACAGAAUCC ((((...(((((..(((((((....((((((.(((........(((.((...)).))).....)))))))))(((.(((......).)).)))...)))))))...))))).)))).... ( -46.62) >DroPse_CAF1 6285 120 - 1 CCUCCAGGCGGGCUCAACCAUGAUUACCUGCCUGCUGUUGCUGCUGACCAGCUCACAGCACGGCUAUCAGGUGGCGGCCGGCAUUGUGCCGCCACCCUGGAGUGAUCCGGCCACGAACCC ......(((.((.(((.(((((((.....(((((((((.((((.....))))..)))))).))).))))(((((((((((....)).))))))))).)))..))).)).)))........ ( -55.00) >DroGri_CAF1 4158 108 - 1 CAUACAGGCGUGCAGCAGCAUUCUCACCUGCCUGCUGCUUUUGCUGAC------ACAAC------AACAGGCAUUGGCCGGCAUUGUGCCGCCGCCAUGGAGUGAUCCGGCCAAGAAUCC ......((((.(((((((((........)).)))))))...))))...------.....------....(((..((((((((.....))))..))))((((....)))))))........ ( -39.00) >DroYak_CAF1 5466 120 - 1 UCUGCAGGCGGGCAGCUCCAUCCUCACCUGCCUGCUGCUCCUACUGACCACCGGCCAACAUCCGCAGCAGGUGGCGGCCGGAAUAGUGCCCCCACCGUGGAGUGAUCCCGCACAGAAUCC ((((...(((((..(((((((........(((((((((......((.((...)).))......)))))))))((.((((......).))).))...)))))))...))))).)))).... ( -49.70) >DroAna_CAF1 4249 120 - 1 UCUGCAGGCGGGCAGCUCCAUCCUCACCUGCCUGCUCCUGCUACUGACCACCACCCAAAACUCCCACCAAGUGGCGGCUGGCAUUGUGCCACCGCCCUGGAGCGAUCCCGCCAAGAACCC (((...((((((..(((((....(((...((........))...)))......................((.(((((.((((.....))))))))))))))))...)))))).))).... ( -44.40) >DroPer_CAF1 6434 120 - 1 CCUCCAGGCGGGCUCAACCAUGAUUACCUGCCUGCUGUUGCUGCUGACCAGCUCACAGCAAGGCUAUCAGGUGGCGGCCGGCAUUGUGCCGCCACCCUGGAGUGAUCCGGCCACGAACCC ......(((.((.(((.(((((((.....(((((((((.((((.....))))..))))).)))).))))(((((((((((....)).))))))))).)))..))).)).)))........ ( -55.00) >consensus CCUGCAGGCGGGCAGCACCAUCCUCACCUGCCUGCUGCUGCUACUGACCACCGCACAACACCCCCAGCAGGUGGCGGCCGGCAUUGUGCCGCCACCCUGGAGUGAUCCCGCCAAGAACCC ....((((((((..............))))))))...................................((((((((((......).)))))))))..(((....)))............ (-28.84 = -29.67 + 0.84)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:51:53 2006