
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 15,085,639 – 15,085,742 |
| Length | 103 |
| Max. P | 0.866598 |

| Location | 15,085,639 – 15,085,742 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 81.81 |
| Mean single sequence MFE | -42.65 |
| Consensus MFE | -27.27 |
| Energy contribution | -28.75 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.27 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.776010 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
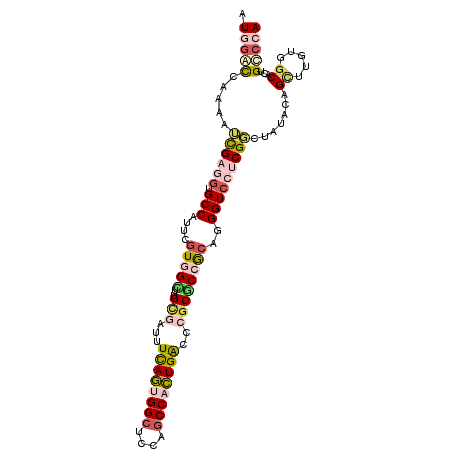
>3L_DroMel_CAF1 15085639 103 + 23771897 GUGGACCAAAAUUGAGGUGCCAUUCGUGGUUUGCGAUUUCAGUGGCUCUAGCCACUGACCCGCGCCGCAGGGUCCUCGGCUAUACAGCUUGUGGCACUGCCCA (((((((........))).))))..(((((..(((...((((((((....))))))))..)))))))).((((.....((((((.....))))))...)))). ( -42.90) >DroVir_CAF1 2818 103 + 1 AUAGCUCAAGAGCGCGGUGCCGUUGAUCGAUUGCGUUUUUAGCGGCUCCAGCCACUGGCCGGCACCGCAGGGUCCUCGCGAGUACAGUUUAUGGCACUGGCCA ...(((....))).(((((((((.(((.(.(((((..((..((((..((.(((...))).))..))))..))....)))))...).))).))))))))).... ( -37.90) >DroSec_CAF1 2993 103 + 1 GUGGACCAAAAUCGAGGUGCCAUUCGUGGCUUGGGAUUUCAGUGGCUCUAACCACUGACCCGCGCCGCAGGGUCCUCGGCUAUACAGCUUGUGGCACUGCCCA (..(........(((((.(((....(((((.((((...(((((((......))))))))))).)))))..))))))))((((((.....)))))).)..)... ( -44.30) >DroEre_CAF1 2872 103 + 1 GUGGGCCAAAAUUGAAGUGCCAUUCGUGGCUUGCGAUUUCAGUGGCUCCAGCCACUGACCCGCGCCGCAGGGUCCUCGGCUAUACAGCUUGUGGCACUGCCCA .(((((((....)).((((((((....((((.(((...((((((((....))))))))..)))((((.((....)))))).....)))).))))))))))))) ( -49.70) >DroYak_CAF1 2886 103 + 1 AUGUGCCACGAUCGAGGUGCCAUUCGUGGCUUGUGAUUUCAGUGGCUCCAGCCACUGACCCGCGCCGCAGGGUCCUCGUGUAUACAGCUUGUGGCACUGCCCA ..(((((((((.(((((.(((....(((((..(((...((((((((....))))))))..))))))))..))))))))((....))..)))))))))...... ( -48.60) >DroMoj_CAF1 2801 103 + 1 AUAGCCAAAGAGCGCACUGCCAUUGGUUGUUUGCGUUUUCAACGGCUCCAGCCAUUGGCCGGCACCACAAGGUCCUCGCGUGUAAAGCUUGUGACACUGGCCA ...((((..((((((((.((((.((((((...((((.....)).))..)))))).)))).((.(((....)))))....))))...)))).......)))).. ( -32.50) >consensus AUGGACCAAAAUCGAGGUGCCAUUCGUGGCUUGCGAUUUCAGUGGCUCCAGCCACUGACCCGCGCCGCAGGGUCCUCGGCUAUACAGCUUGUGGCACUGCCCA .(((((.....((((((.(((....(((((..(((...((((((((....))))))))..))))))))..))))))))).......((.....))...))))) (-27.27 = -28.75 + 1.48)

| Location | 15,085,639 – 15,085,742 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 81.81 |
| Mean single sequence MFE | -42.67 |
| Consensus MFE | -29.80 |
| Energy contribution | -29.89 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.866598 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
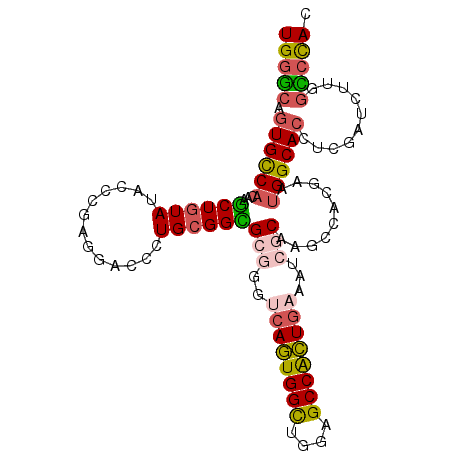
>3L_DroMel_CAF1 15085639 103 - 23771897 UGGGCAGUGCCACAAGCUGUAUAGCCGAGGACCCUGCGGCGCGGGUCAGUGGCUAGAGCCACUGAAAUCGCAAACCACGAAUGGCACCUCAAUUUUGGUCCAC (((((.((((((...........((((((....)).))))((((.((((((((....))))))))..))))..........))))))..((....))))))). ( -43.30) >DroVir_CAF1 2818 103 - 1 UGGCCAGUGCCAUAAACUGUACUCGCGAGGACCCUGCGGUGCCGGCCAGUGGCUGGAGCCGCUAAAAACGCAAUCGAUCAACGGCACCGCGCUCUUGAGCUAU ..((.(((((........))))).)).........(((((((((...((((((....)))))).....((....)).....)))))))))(((....)))... ( -39.20) >DroSec_CAF1 2993 103 - 1 UGGGCAGUGCCACAAGCUGUAUAGCCGAGGACCCUGCGGCGCGGGUCAGUGGUUAGAGCCACUGAAAUCCCAAGCCACGAAUGGCACCUCGAUUUUGGUCCAC (((((.((((((...(((.....((((((....)).))))..(((((((((((....))))))))...))).)))......))))))(........)))))). ( -41.00) >DroEre_CAF1 2872 103 - 1 UGGGCAGUGCCACAAGCUGUAUAGCCGAGGACCCUGCGGCGCGGGUCAGUGGCUGGAGCCACUGAAAUCGCAAGCCACGAAUGGCACUUCAAUUUUGGCCCAC ((((((((((((...(((.....((((((....)).))))((((.((((((((....))))))))..)))).)))......))))))).((....))))))). ( -48.10) >DroYak_CAF1 2886 103 - 1 UGGGCAGUGCCACAAGCUGUAUACACGAGGACCCUGCGGCGCGGGUCAGUGGCUGGAGCCACUGAAAUCACAAGCCACGAAUGGCACCUCGAUCGUGGCACAU ......(((((((....((....))(((((..(((((...)))))((((((((....))))))))........((((....)))).)))))...))))))).. ( -46.30) >DroMoj_CAF1 2801 103 - 1 UGGCCAGUGUCACAAGCUUUACACGCGAGGACCUUGUGGUGCCGGCCAAUGGCUGGAGCCGUUGAAAACGCAAACAACCAAUGGCAGUGCGCUCUUUGGCUAU (((((.(..(((((((((((......))))..)))))))..).)))))(((((..(((((((((..............)))))))((....)).))..))))) ( -38.14) >consensus UGGGCAGUGCCACAAGCUGUAUACCCGAGGACCCUGCGGCGCGGGUCAGUGGCUGGAGCCACUGAAAUCGCAAGCCACGAAUGGCACCUCGAUCUUGGCCCAC (((((.((((((...((((((.............))))))(((..((((((((....))))))))...)))..........))))))..........))))). (-29.80 = -29.89 + 0.09)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:51:50 2006