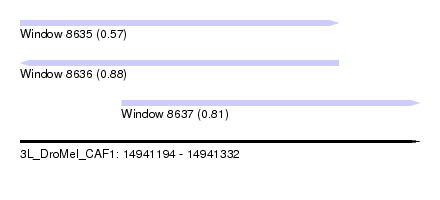
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 14,941,194 – 14,941,332 |
| Length | 138 |
| Max. P | 0.880674 |

| Location | 14,941,194 – 14,941,304 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 81.76 |
| Mean single sequence MFE | -30.77 |
| Consensus MFE | -17.23 |
| Energy contribution | -18.73 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.572783 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
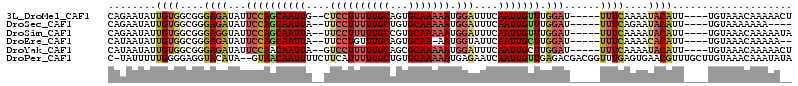
>3L_DroMel_CAF1 14941194 110 + 23771897 ACACUGGAAAAUCCC---AA-AAAGCGAUUGGGCAUCCACAGAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUG--CUCCUUUUUGCAGUGCAAAAAUGGAUUUCAAUUGUUUGGAU- ...((((((..((((---..-...((......))..((((((....)))))).))))....))))))((((((--.((((((((((...))))))).)))...)))))).......- ( -33.20) >DroSec_CAF1 121510 111 + 1 ACACUGGAAAUUCCC---AAAAAAGCGAUUGGGCAUCCACAGAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUA--UUCCUUUUUGCUGUGCAAAAAUGGAUUUCAAUUGUUUGGAU- ...((((((.(((((---......((......))..((((((....)))))).)))))...))))))(((((.--.((((((((((...))))))).)))....))))).......- ( -30.60) >DroSim_CAF1 119248 110 + 1 ACACUGGAAAUUCCC---AAAAAAGCGAUUGGG-AUAAACAGAAUAUUGUGGCGGGAGGUAUUCCAGCAAUUA--UUCCUUUUUGCCGUGCAAAAAUGGAUUUCAAUUGUUUGGAU- ...(((.....((((---((........)))))-)....)))......(((.(....).)))((((((((((.--.((((((((((...))))))).)))....))))).))))).- ( -28.10) >DroEre_CAF1 116250 112 + 1 UCGCUGGAAAUUCACUGGAA-AAGGCGAUUGGGCAUCCACAUAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUA--UUCCUGUUUGCAGUGCAA-AAUGGUAUUCAAUUGCUUGGAU- ..((((((((((..(((...-...((......))..(((((......))))))))..))).))))))).....--.(((.(((((.(((((..-....))))))))..))..))).- ( -30.20) >DroYak_CAF1 117203 110 + 1 GCACUGGAAAUUCCC---AA-AAAGCGGUUGGGCAUCCACAUAAUAUUGUGGCGGGAGAUAUUCCAACAAUUA--GUCCUUUUUGCAGCGCAAAAAUGGAUUUCAAUUGCUUGGAU- ..........(((((---..-...((......))..(((((......))))).)))))....(((((((((((--(((((((((((...))))))).)))))..))))).))))).- ( -33.00) >DroPer_CAF1 169882 107 + 1 --ACUGAAAAUGCCG---AA-AAAGCGGU-GGAUAGCCAC-UAUUUUUGGGGAGGUACAUA--GUAACAAUUUUCUUCAUUUUUGCUGUGCAAAAAUGAGAAUCAAUUGUUGAGACG --..((.(..(.((.---((-(((..(((-((....))))-).))))).)).)..).))..--.((((((((((((.(((((((((...)))))))))))))..))))))))..... ( -29.50) >consensus ACACUGGAAAUUCCC___AA_AAAGCGAUUGGGCAUCCACAGAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUA__UUCCUUUUUGCAGUGCAAAAAUGGAUUUCAAUUGUUUGGAU_ ......................((((((((((....(((((......))))).((((....))))...........((((((((((...))))))).)))..))))))))))..... (-17.23 = -18.73 + 1.50)



| Location | 14,941,194 – 14,941,304 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 81.76 |
| Mean single sequence MFE | -28.08 |
| Consensus MFE | -17.64 |
| Energy contribution | -20.37 |
| Covariance contribution | 2.73 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.880674 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14941194 110 - 23771897 -AUCCAAACAAUUGAAAUCCAUUUUUGCACUGCAAAAAGGAG--CAAUUGCUGGAAUAUCUCCCGCCACAAUAUUCUGUGGAUGCCCAAUCGCUUU-UU---GGGAUUUUCCAGUGU -.......((((((...(((.(((((((...)))))))))).--))))))((((((....((((.(((((......)))))..((......))...-..---))))..))))))... ( -34.40) >DroSec_CAF1 121510 111 - 1 -AUCCAAACAAUUGAAAUCCAUUUUUGCACAGCAAAAAGGAA--UAAUUGCUGGAAUAUCUCCCGCCACAAUAUUCUGUGGAUGCCCAAUCGCUUUUUU---GGGAAUUUCCAGUGU -.......((((((...(((.(((((((...)))))))))).--))))))((((((....((((.(((((......)))))..((......))......---))))..))))))... ( -30.40) >DroSim_CAF1 119248 110 - 1 -AUCCAAACAAUUGAAAUCCAUUUUUGCACGGCAAAAAGGAA--UAAUUGCUGGAAUACCUCCCGCCACAAUAUUCUGUUUAU-CCCAAUCGCUUUUUU---GGGAAUUUCCAGUGU -.......((((((...(((.(((((((...)))))))))).--))))))((((((...........(((......)))...(-(((((........))---))))..))))))... ( -24.90) >DroEre_CAF1 116250 112 - 1 -AUCCAAGCAAUUGAAUACCAUU-UUGCACUGCAAACAGGAA--UAAUUGCUGGAAUAUCUCCCGCCACAAUAUUAUGUGGAUGCCCAAUCGCCUU-UUCCAGUGAAUUUCCAGCGA -...(((....)))....((..(-((((...)))))..))..--...(((((((((.........(((((......)))))........((((...-.....))))..))))))))) ( -26.20) >DroYak_CAF1 117203 110 - 1 -AUCCAAGCAAUUGAAAUCCAUUUUUGCGCUGCAAAAAGGAC--UAAUUGUUGGAAUAUCUCCCGCCACAAUAUUAUGUGGAUGCCCAACCGCUUU-UU---GGGAAUUUCCAGUGC -.....((((((((...(((.(((((((...)))))))))).--))))))))((((....((((.(((((......)))))..((......))...-..---))))..))))..... ( -29.60) >DroPer_CAF1 169882 107 - 1 CGUCUCAACAAUUGAUUCUCAUUUUUGCACAGCAAAAAUGAAGAAAAUUGUUAC--UAUGUACCUCCCCAAAAAUA-GUGGCUAUCC-ACCGCUUU-UU---CGGCAUUUUCAGU-- .(((..(((((((..(((((((((((((...))))))))).)))))))))))..--...............(((.(-((((......-.))))).)-))---.))).........-- ( -23.00) >consensus _AUCCAAACAAUUGAAAUCCAUUUUUGCACUGCAAAAAGGAA__UAAUUGCUGGAAUAUCUCCCGCCACAAUAUUAUGUGGAUGCCCAAUCGCUUU_UU___GGGAAUUUCCAGUGU ........((((((...(((.(((((((...))))))))))...))))))((((((....((((.(((((......)))))..((......)).........))))..))))))... (-17.64 = -20.37 + 2.73)



| Location | 14,941,229 – 14,941,332 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.54 |
| Mean single sequence MFE | -23.57 |
| Consensus MFE | -15.84 |
| Energy contribution | -16.40 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.813431 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14941229 103 + 23771897 CAGAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUG--CUCCUUUUUGCAGUGCAAAAAUGGAUUUCAAUUGUUUGGAU-----UUUCAAAAUACAUU----UGUAAACAAAAACU ........((((....((((...(((((((((((--.((((((((((...))))))).)))...)))))).))))).-----))))....))))((----((....)))).... ( -25.40) >DroSec_CAF1 121546 99 + 1 CAGAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUA--UUCCUUUUUGCUGUGCAAAAAUGGAUUUCAAUUGUUUGGAU-----UUUCAGAAUACAUU----UGUAAAAAAA---- ........((((....((((...((((((((((.--.((((((((((...))))))).)))....))))).))))).-----))))....))))..----..........---- ( -22.20) >DroSim_CAF1 119283 103 + 1 CAGAAUAUUGUGGCGGGAGGUAUUCCAGCAAUUA--UUCCUUUUUGCCGUGCAAAAAUGGAUUUCAAUUGUUUGGAU-----UUUCAAAAUACAUU----UGUAAACAAAAAUA ........((((....((((...((((((((((.--.((((((((((...))))))).)))....))))).))))).-----))))....))))((----((....)))).... ( -22.20) >DroEre_CAF1 116288 100 + 1 CAUAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUA--UUCCUGUUUGCAGUGCAA-AAUGGUAUUCAAUUGCUUGGAU-----UUUCAAAACACAUU----UGUAAACAAAAA-- .((((...((((....((((...((((((((((.--..((..(((((...))))-)..)).....)))))).)))).-----))))....)))).)----))).........-- ( -23.30) >DroYak_CAF1 117238 103 + 1 CAUAAUAUUGUGGCGGGAGAUAUUCCAACAAUUA--GUCCUUUUUGCAGCGCAAAAAUGGAUUUCAAUUGCUUGGAU-----UUUCAAAAUACAUU----UGUAAACAAAAACU .((((...((((....((((...(((((((((((--(((((((((((...))))))).)))))..))))).))))).-----))))....)))).)----)))........... ( -24.90) >DroPer_CAF1 169914 111 + 1 C-UAUUUUUGGGGAGGUACAUA--GUAACAAUUUUCUUCAUUUUUGCUGUGCAAAAAUGAGAAUCAAUUGUUGAGACGACGGUUUGAGUGAACGUUUGCUUGUAAACAAAUAUA .-(((.((((.(.(((((....--.((((((((((((.(((((((((...)))))))))))))..))))))))....((((...........))))))))).)...)))).))) ( -23.40) >consensus CAGAAUAUUGUGGCGGGAGAUAUUCCAGCAAUUA__UUCCUUUUUGCAGUGCAAAAAUGGAUUUCAAUUGUUUGGAU_____UUUCAAAAUACAUU____UGUAAACAAAAA__ ........((((....((((...((((((((((....((((((((((...))))))).)))....))))))).)))......))))....)))).................... (-15.84 = -16.40 + 0.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:50:53 2006