
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 14,922,324 – 14,922,422 |
| Length | 98 |
| Max. P | 0.996149 |

| Location | 14,922,324 – 14,922,422 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 89.93 |
| Mean single sequence MFE | -38.98 |
| Consensus MFE | -31.82 |
| Energy contribution | -32.29 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.16 |
| Mean z-score | -4.55 |
| Structure conservation index | 0.82 |
| SVM decision value | 2.66 |
| SVM RNA-class probability | 0.996149 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
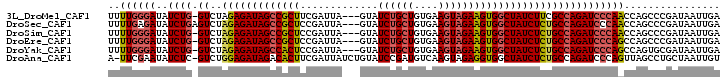
>3L_DroMel_CAF1 14922324 98 + 23771897 UUUUGGGAUAUCUG-GUCUAGAGAUAGCCGCUUCGAUUA---GUAUCUGCUGUGAAGUAGAAGUGGCUAUCUUCGCCAGAUCCCAACCAGCCCGAUAAUUGA ..((((((..((((-((...(((((((((((((((((..---..)))((((....)))))))))))))))))).))))))))))))................ ( -42.30) >DroSec_CAF1 102915 99 + 1 UUUUGAGAUAUCUGAGUCUAGAGAUAGCCGCUCCGAUUA---GUAUCUGCUGUGAAGUAGAAGUGGCUAUCUCUGCCAGAUCCCAACCAGCCCGAUAAUUGA ......(..(((((.((..(((((((((((((.......---...((((((....))))))))))))))))))))))))))..).................. ( -32.50) >DroSim_CAF1 100238 98 + 1 UUUUGGGAUAUCUG-GUCUAGAGAUAGCCGCUCCGAUUA---GUAUCUGCUGUGAAGUAGAAGUGGCUAUCUCUGCCAGAUCCCAACCAGCCCGAUAAUUGA ..((((((..((((-((..(((((((((((((.......---...((((((....)))))))))))))))))))))))))))))))................ ( -43.20) >DroEre_CAF1 98383 98 + 1 UUUUGGGAUAUCUG-GUCUAGAGAUAGCCGCUCCGAUUA---GUAUCUGCUGUGAAGUAGAAGUGGCUAUCUCUGCCAGAUCCCAGCCAGCCCGAUAAUUGA ..((((((..((((-((..(((((((((((((.......---...((((((....)))))))))))))))))))))))))))))))................ ( -42.90) >DroYak_CAF1 98364 98 + 1 UUUUGGGAUAUCUG-GUCUAGAGAUAGCCACUCCGAUUA---GUAUCUGCUGUGAAGUAGAAGUGGCUAUCUCUGCCAGAUCCCAGCCAGUGCGAUAAUUGA ..((((((..((((-((..(((((((((((((.......---...((((((....))))))))))))))))))))))))))))))).((((......)))). ( -44.80) >DroAna_CAF1 89605 100 + 1 A-UUCGAAUAUCUC-GUCUGGAGAUAGACACUUCGAUUAUCUGUAUCCGAUGUCAAGUAGAGGUGGCUAUCUCUGCCAGAUCCCAGUUAGCCUGCUAAUUGU .-............-((((((((((((.((((((...((((.......)))).......)))))).))))))...))))))..(((((((....))))))). ( -28.20) >consensus UUUUGGGAUAUCUG_GUCUAGAGAUAGCCGCUCCGAUUA___GUAUCUGCUGUGAAGUAGAAGUGGCUAUCUCUGCCAGAUCCCAACCAGCCCGAUAAUUGA ..((((((..((((.((..(((((((((((((.............((((((....)))))))))))))))))))))))))))))))................ (-31.82 = -32.29 + 0.48)

| Location | 14,922,324 – 14,922,422 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 89.93 |
| Mean single sequence MFE | -32.73 |
| Consensus MFE | -24.70 |
| Energy contribution | -25.09 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.16 |
| Mean z-score | -3.39 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.777694 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
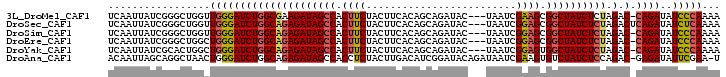
>3L_DroMel_CAF1 14922324 98 - 23771897 UCAAUUAUCGGGCUGGUUGGGAUCUGGCGAAGAUAGCCACUUCUACUUCACAGCAGAUAC---UAAUCGAAGCGGCUAUCUCUAGAC-CAGAUAUCCCAAAA ................((((((((((((..((((((((.((((..((....))..(((..---..))))))).))))))))...).)-))))..)))))).. ( -32.50) >DroSec_CAF1 102915 99 - 1 UCAAUUAUCGGGCUGGUUGGGAUCUGGCAGAGAUAGCCACUUCUACUUCACAGCAGAUAC---UAAUCGGAGCGGCUAUCUCUAGACUCAGAUAUCUCAAAA ................((((((((((((((((((((((.((((..((....))..(((..---..))))))).)))))))))).).).))))..)))))).. ( -29.90) >DroSim_CAF1 100238 98 - 1 UCAAUUAUCGGGCUGGUUGGGAUCUGGCAGAGAUAGCCACUUCUACUUCACAGCAGAUAC---UAAUCGGAGCGGCUAUCUCUAGAC-CAGAUAUCCCAAAA ................((((((((((((((((((((((.((((..((....))..(((..---..))))))).)))))))))).).)-))))..)))))).. ( -36.70) >DroEre_CAF1 98383 98 - 1 UCAAUUAUCGGGCUGGCUGGGAUCUGGCAGAGAUAGCCACUUCUACUUCACAGCAGAUAC---UAAUCGGAGCGGCUAUCUCUAGAC-CAGAUAUCCCAAAA .................(((((((((((((((((((((.((((..((....))..(((..---..))))))).)))))))))).).)-))))..)))))... ( -35.70) >DroYak_CAF1 98364 98 - 1 UCAAUUAUCGCACUGGCUGGGAUCUGGCAGAGAUAGCCACUUCUACUUCACAGCAGAUAC---UAAUCGGAGUGGCUAUCUCUAGAC-CAGAUAUCCCAAAA .........((....))((((((((((((((((((((((((((..((....))..(((..---..)))))))))))))))))).).)-))))..)))))... ( -40.30) >DroAna_CAF1 89605 100 - 1 ACAAUUAGCAGGCUAACUGGGAUCUGGCAGAGAUAGCCACCUCUACUUGACAUCGGAUACAGAUAAUCGAAGUGUCUAUCUCCAGAC-GAGAUAUUCGAA-U .((.((((....)))).))(((..((((.......))))..)))........(((((((.(((((.......))))).((((.....-))))))))))).-. ( -21.30) >consensus UCAAUUAUCGGGCUGGCUGGGAUCUGGCAGAGAUAGCCACUUCUACUUCACAGCAGAUAC___UAAUCGGAGCGGCUAUCUCUAGAC_CAGAUAUCCCAAAA .................(((((((((((((((((((((.((((.........................)))).)))))))))).).).))))..)))))... (-24.70 = -25.09 + 0.39)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:50:38 2006